+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3045 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Density map of the scanning state of the mammalian SRP-ribosome complex | |||||||||
 マップデータ マップデータ | This is the final masked and sharpened reconstruction | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  ribosomes (リボソーム) / SRP / ribosomes (リボソーム) / SRP /  mammal (哺乳類) / translocation mammal (哺乳類) / translocation | |||||||||
| 生物種 |   Oryctolagus cuniculus (ウサギ) Oryctolagus cuniculus (ウサギ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.9 Å クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Voorhees RM / Hegde RS | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2015 ジャーナル: Elife / 年: 2015タイトル: Structures of the scanning and engaged states of the mammalian SRP-ribosome complex. 著者: Rebecca M Voorhees / Ramanujan S Hegde /  要旨: The universally conserved signal recognition particle (SRP) is essential for the biogenesis of most integral membrane proteins. SRP scans the nascent chains of translating ribosomes, preferentially ...The universally conserved signal recognition particle (SRP) is essential for the biogenesis of most integral membrane proteins. SRP scans the nascent chains of translating ribosomes, preferentially engaging those with hydrophobic targeting signals, and delivers these ribosome-nascent chain complexes to the membrane. Here, we present structures of native mammalian SRP-ribosome complexes in the scanning and engaged states. These structures reveal the near-identical SRP architecture of these two states, show many of the SRP-ribosome interactions at atomic resolution, and suggest how the polypeptide-binding M domain selectively engages hydrophobic signals. The scanning M domain, pre-positioned at the ribosomal exit tunnel, is auto-inhibited by a C-terminal amphipathic helix occluding its hydrophobic binding groove. Upon engagement, the hydrophobic targeting signal displaces this amphipathic helix, which then acts as a protective lid over the signal. Biochemical experiments suggest how scanning and engagement are coordinated with translation elongation to minimize exposure of hydrophobic signals during membrane targeting. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3045.map.gz emd_3045.map.gz | 31.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3045-v30.xml emd-3045-v30.xml emd-3045.xml emd-3045.xml | 8.4 KB 8.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-3045.png EMD-3045.png | 354.4 KB | ||
| その他 |  scanning_half1_unfil.map scanning_half1_unfil.map scanning_half2_unfil.map scanning_half2_unfil.map | 282.6 MB 282.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3045 http://ftp.pdbj.org/pub/emdb/structures/EMD-3045 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3045 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3045 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3045.map.gz / 形式: CCP4 / 大きさ: 276 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3045.map.gz / 形式: CCP4 / 大きさ: 276 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is the final masked and sharpened reconstruction | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: scanning half1 unfil.map
| ファイル | scanning_half1_unfil.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
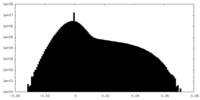
| 投影像・断面図 |
| ||||||||||||
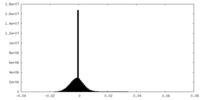
| 密度ヒストグラム |
-添付マップデータ: scanning half2 unfil.map
| ファイル | scanning_half2_unfil.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
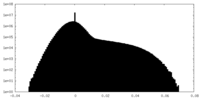
| 投影像・断面図 |
| ||||||||||||
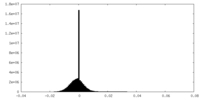
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of the mammalian ribosome and SRP in its scanning mode
| 全体 | 名称: Complex of the mammalian ribosome and SRP in its scanning mode |
|---|---|
| 要素 |
|
-超分子 #1000: Complex of the mammalian ribosome and SRP in its scanning mode
| 超分子 | 名称: Complex of the mammalian ribosome and SRP in its scanning mode タイプ: sample / ID: 1000 / 集合状態: Heterodimer of mammalian ribosome and SRP / Number unique components: 2 |
|---|
-超分子 #1: Mammalian ribosome
| 超分子 | 名称: Mammalian ribosome / タイプ: complex / ID: 1 / Name.synonym: 80S ribosome / 組換発現: No / Ribosome-details: ribosome-eukaryote: ALL |
|---|---|
| 由来(天然) | 生物種:   Oryctolagus cuniculus (ウサギ) / 別称: Rabbit / 細胞: reticulocyte Oryctolagus cuniculus (ウサギ) / 別称: Rabbit / 細胞: reticulocyte |
-分子 #1: Signal recognition particle
| 分子 | 名称: Signal recognition particle / タイプ: rna / ID: 1 / Name.synonym: SRP / 詳細: ribonucleoprotein complex / 分類: OTHER / Structure: OTHER / Synthetic?: No |
|---|---|
| 由来(天然) | 生物種:   Oryctolagus cuniculus (ウサギ) / 別称: Rabbit Oryctolagus cuniculus (ウサギ) / 別称: Rabbit |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 詳細: 50 mM HEPES pH 7.5, 200 mM 469 KAc, 15 mM MgAc2, and 1 mM DTT |
|---|---|
| グリッド | 詳細: glow-discharged holey carbon grids (Quantifoil R2/2), coated with a ~70 angstrom thick layer of amorphous carbon |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK IV 手法: sample was applied to the grid, followed by a 30 s incubation at 4C, 3 s of blotting |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 59000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 日付 | 2014年11月14日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 27 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.9 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Relion 詳細: Final maps were calculated from two averaged data sets 使用した粒子像数: 27627 |
| 詳細 | The particles were selected using the automated particle picker in Relion |
 ムービー
ムービー コントローラー
コントローラー



















 Z
Z Y
Y X
X