+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 9o62 | ||||||
|---|---|---|---|---|---|---|---|
| Title | 1C5H TCR bound to R-phycoerythrin | ||||||
 Components Components |
| ||||||
 Keywords Keywords | IMMUNE SYSTEM / T-cell / gamma delta TCR / phycoerythrin / direct | ||||||
| Function / homology |  Function and homology information Function and homology information | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) Pyropia tenera (asakusa nori) Pyropia tenera (asakusa nori) | ||||||
| Method | ELECTRON MICROSCOPY / single particle reconstruction / cryo EM / Resolution: 2.03 Å | ||||||
 Authors Authors | Rashleigh, L. / Venugopal, H. / Rossjohn, J. / Gully, B.S. | ||||||
| Funding support |  Australia, 1items Australia, 1items
| ||||||
 Citation Citation |  Journal: Structure / Year: 2025 Journal: Structure / Year: 2025Title: Antibody-like recognition of a γδ T cell receptor toward a foreign antigen. Authors: Liam Rashleigh / Hariprasad Venugopal / Michael T Rice / Sachith D Gunasinghe / Chhon Ling Sok / Nicholas A Gherardin / Catarina F Almeida / Ildiko Van Rhijn / D Branch Moody / Dale I ...Authors: Liam Rashleigh / Hariprasad Venugopal / Michael T Rice / Sachith D Gunasinghe / Chhon Ling Sok / Nicholas A Gherardin / Catarina F Almeida / Ildiko Van Rhijn / D Branch Moody / Dale I Godfrey / Jamie Rossjohn / Benjamin S Gully /    Abstract: The antigen recognition principles of B cells and αβ T cells have been well described compared to those of the γδ T cell. By way of their specificity conferring receptor (γδTCR), γδ T cells ...The antigen recognition principles of B cells and αβ T cells have been well described compared to those of the γδ T cell. By way of their specificity conferring receptor (γδTCR), γδ T cells can directly bind proteinaceous antigens. A known γδ T cell and B cell model antigen is phycoerythrin (PE), a light harvesting protein from rhodophytes and cyanobacteria. Here we probed human γδTCR reactivity to PE, in which a Vδ1Vγ5 TCR bound directly to induce proximal signaling and cellular activation. We determined the cryoelectron microscopy (cryo-EM) structure of the γδTCR-phycoerythrin immune complex. We then determined the cryo-EM structures of an antibody fragment and an αβTCR bound to PE. This revealed convergent use of apical aromatic residues to mediate contacts with a common PE epitope. Comparative analyses of the γδTCR revealed multiple antibody-like characteristics, including an enrichment of apical aromatic residues. Our findings reveal further distinct facets of antigen recognition by the γδTCR. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  9o62.cif.gz 9o62.cif.gz | 470.7 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb9o62.ent.gz pdb9o62.ent.gz | 394.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  9o62.json.gz 9o62.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/o6/9o62 https://data.pdbj.org/pub/pdb/validation_reports/o6/9o62 ftp://data.pdbj.org/pub/pdb/validation_reports/o6/9o62 ftp://data.pdbj.org/pub/pdb/validation_reports/o6/9o62 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  70157MC  9mgbC  9mkoC  9o60C  9o61C C: citing same article ( M: map data used to model this data |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
|
|---|---|
| 1 |
|
- Components
Components
-R-phycoerythrin class I ... , 2 types, 12 molecules ACEGIKBDFHJL
| #1: Protein | Mass: 17707.906 Da / Num. of mol.: 6 / Source method: isolated from a natural source / Source: (natural)  Pyropia tenera (asakusa nori) / References: UniProt: A0A1C9C9A7 Pyropia tenera (asakusa nori) / References: UniProt: A0A1C9C9A7#2: Protein | Mass: 18336.850 Da / Num. of mol.: 6 / Source method: isolated from a natural source / Source: (natural)  Pyropia tenera (asakusa nori) / References: UniProt: A0A1C9C989 Pyropia tenera (asakusa nori) / References: UniProt: A0A1C9C989 |
|---|
-Protein , 2 types, 2 molecules ab
| #3: Protein | Mass: 26886.508 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Cell line (production host): Expi293 / Production host: Homo sapiens (human) / Cell line (production host): Expi293 / Production host:  Homo sapiens (human) Homo sapiens (human) |
|---|---|
| #4: Protein | Mass: 27014.568 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Cell line (production host): Expi293 / Production host: Homo sapiens (human) / Cell line (production host): Expi293 / Production host:  Homo sapiens (human) Homo sapiens (human) |
-Non-polymers , 2 types, 30 molecules 
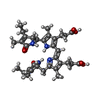

| #5: Chemical | ChemComp-PEB / #6: Chemical | ChemComp-PUB / |
|---|
-Details
| Has ligand of interest | Y |
|---|---|
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method: ELECTRON MICROSCOPY |
|---|---|
| EM experiment | Aggregation state: PARTICLE / 3D reconstruction method: single particle reconstruction |
- Sample preparation
Sample preparation
| Component |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Source (natural) |
| ||||||||||||||||||||||||
| Source (recombinant) |
| ||||||||||||||||||||||||
| Buffer solution | pH: 8.5 | ||||||||||||||||||||||||
| Specimen | Embedding applied: NO / Shadowing applied: NO / Staining applied: NO / Vitrification applied: YES | ||||||||||||||||||||||||
| Vitrification | Cryogen name: ETHANE |
- Electron microscopy imaging
Electron microscopy imaging
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
|---|---|
| Microscopy | Model: TFS KRIOS |
| Electron gun | Electron source:  FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM FIELD EMISSION GUN / Accelerating voltage: 300 kV / Illumination mode: FLOOD BEAM |
| Electron lens | Mode: BRIGHT FIELD / Nominal defocus max: 2000 nm / Nominal defocus min: 400 nm |
| Image recording | Electron dose: 60 e/Å2 / Film or detector model: GATAN K3 (6k x 4k) |
- Processing
Processing
| EM software | Name: PHENIX / Version: 1.21.1_5286: / Category: model refinement |
|---|---|
| CTF correction | Type: PHASE FLIPPING AND AMPLITUDE CORRECTION |
| 3D reconstruction | Resolution: 2.03 Å / Resolution method: OTHER / Num. of particles: 45513 / Symmetry type: POINT |
 Movie
Movie Controller
Controller









 PDBj
PDBj

