+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8ots | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | OCT4 and MYC-MAX co-bound to a nucleosome | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | TRANSCRIPTION | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Mad-Max complex / cell fate commitment involved in formation of primary germ layer / positive regulation of metanephric cap mesenchymal cell proliferation / positive regulation of acinar cell proliferation / acinar cell proliferation / negative regulation of transcription initiation by RNA polymerase II / SCF ubiquitin ligase complex binding / NK T cell proliferation / cardiac cell fate determination / Myc-Max complex ...Mad-Max complex / cell fate commitment involved in formation of primary germ layer / positive regulation of metanephric cap mesenchymal cell proliferation / positive regulation of acinar cell proliferation / acinar cell proliferation / negative regulation of transcription initiation by RNA polymerase II / SCF ubiquitin ligase complex binding / NK T cell proliferation / cardiac cell fate determination / Myc-Max complex / POU5F1 (OCT4), SOX2, NANOG repress genes related to differentiation / Formation of the anterior neural plate / endodermal-mesodermal cell signaling / regulation of asymmetric cell division / endodermal cell fate specification / regulation of somatic stem cell population maintenance / regulation of cell cycle process / Binding of TCF/LEF:CTNNB1 to target gene promoters / cellular response to interferon-alpha / RNA polymerase II transcription repressor complex / heart induction / Specification of primordial germ cells / myotube differentiation / positive regulation of B cell apoptotic process / POU5F1 (OCT4), SOX2, NANOG activate genes related to proliferation / RUNX3 regulates WNT signaling / TFAP2 (AP-2) family regulates transcription of cell cycle factors / Specification of the neural plate border / Transcriptional regulation of pluripotent stem cells / negative regulation of cell division / negative regulation of monocyte differentiation / Germ layer formation at gastrulation / detection of mechanical stimulus involved in sensory perception of sound / response to growth factor / response to alkaloid / B cell apoptotic process / transcription regulator activator activity / protein-DNA complex disassembly / Transcription of E2F targets under negative control by DREAM complex / negative regulation of stress-activated MAPK cascade / fibroblast apoptotic process / positive regulation of intrinsic apoptotic signaling pathway by p53 class mediator / Regulation of NFE2L2 gene expression / regulation of telomere maintenance / miRNA binding / positive regulation of mesenchymal cell proliferation / skeletal system morphogenesis / middle ear morphogenesis / Signaling by ALK / branching involved in ureteric bud morphogenesis / negative regulation of gene expression via chromosomal CpG island methylation / pigmentation / rRNA metabolic process / E-box binding / Transcriptional Regulation by E2F6 / positive regulation of telomere maintenance / skeletal muscle cell differentiation / MLL1 complex / blastocyst development / somatic stem cell population maintenance / anatomical structure morphogenesis / negative regulation of tumor necrosis factor-mediated signaling pathway / BMP signaling pathway / chromosome organization / positive regulation of transcription initiation by RNA polymerase II / negative regulation of fibroblast proliferation / negative regulation of megakaryocyte differentiation / Cyclin E associated events during G1/S transition / core promoter sequence-specific DNA binding / protein localization to CENP-A containing chromatin / Cyclin A:Cdk2-associated events at S phase entry / Chromatin modifying enzymes / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / Packaging Of Telomere Ends / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / ERK1 and ERK2 cascade / Deposition of new CENPA-containing nucleosomes at the centromere / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / telomere organization / Interleukin-7 signaling / Inhibition of DNA recombination at telomere / RNA Polymerase I Promoter Opening / Meiotic synapsis / negative regulation of miRNA transcription / Assembly of the ORC complex at the origin of replication / Regulation of endogenous retroelements by the Human Silencing Hub (HUSH) complex / SUMOylation of chromatin organization proteins / DNA methylation / bioluminescence / Condensation of Prophase Chromosomes / epigenetic regulation of gene expression / Chromatin modifications during the maternal to zygotic transition (MZT) / SIRT1 negatively regulates rRNA expression / HCMV Late Events / SMAD2/SMAD3:SMAD4 heterotrimer regulates transcription / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / innate immune response in mucosa 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) synthetic construct (人工物) | |||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Michael, A.K. / Stoos, L. / Kempf, G. / Cavadini, S. / Thoma, N. | |||||||||
| 資金援助 | European Union,  フランス, 2件 フランス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: Cooperation between bHLH transcription factors and histones for DNA access. 著者: Alicia K Michael / Lisa Stoos / Priya Crosby / Nikolas Eggers / Xinyu Y Nie / Kristina Makasheva / Martina Minnich / Kelly L Healy / Joscha Weiss / Georg Kempf / Simone Cavadini / Lukas Kater ...著者: Alicia K Michael / Lisa Stoos / Priya Crosby / Nikolas Eggers / Xinyu Y Nie / Kristina Makasheva / Martina Minnich / Kelly L Healy / Joscha Weiss / Georg Kempf / Simone Cavadini / Lukas Kater / Jan Seebacher / Luca Vecchia / Deyasini Chakraborty / Luke Isbel / Ralph S Grand / Florian Andersch / Jennifer L Fribourgh / Dirk Schübeler / Johannes Zuber / Andrew C Liu / Peter B Becker / Beat Fierz / Carrie L Partch / Jerome S Menet / Nicolas H Thomä /     要旨: The basic helix-loop-helix (bHLH) family of transcription factors recognizes DNA motifs known as E-boxes (CANNTG) and includes 108 members. Here we investigate how chromatinized E-boxes are engaged ...The basic helix-loop-helix (bHLH) family of transcription factors recognizes DNA motifs known as E-boxes (CANNTG) and includes 108 members. Here we investigate how chromatinized E-boxes are engaged by two structurally diverse bHLH proteins: the proto-oncogene MYC-MAX and the circadian transcription factor CLOCK-BMAL1 (refs. ). Both transcription factors bind to E-boxes preferentially near the nucleosomal entry-exit sites. Structural studies with engineered or native nucleosome sequences show that MYC-MAX or CLOCK-BMAL1 triggers the release of DNA from histones to gain access. Atop the H2A-H2B acidic patch, the CLOCK-BMAL1 Per-Arnt-Sim (PAS) dimerization domains engage the histone octamer disc. Binding of tandem E-boxes at endogenous DNA sequences occurs through direct interactions between two CLOCK-BMAL1 protomers and histones and is important for circadian cycling. At internal E-boxes, the MYC-MAX leucine zipper can also interact with histones H2B and H3, and its binding is indirectly enhanced by OCT4 elsewhere on the nucleosome. The nucleosomal E-box position and the type of bHLH dimerization domain jointly determine the histone contact, the affinity and the degree of competition and cooperativity with other nucleosome-bound factors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8ots.cif.gz 8ots.cif.gz | 361.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8ots.ent.gz pdb8ots.ent.gz | 265 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8ots.json.gz 8ots.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  8ots_validation.pdf.gz 8ots_validation.pdf.gz | 1.5 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  8ots_full_validation.pdf.gz 8ots_full_validation.pdf.gz | 1.5 MB | 表示 | |
| XML形式データ |  8ots_validation.xml.gz 8ots_validation.xml.gz | 43.1 KB | 表示 | |
| CIF形式データ |  8ots_validation.cif.gz 8ots_validation.cif.gz | 68.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ot/8ots https://data.pdbj.org/pub/pdb/validation_reports/ot/8ots ftp://data.pdbj.org/pub/pdb/validation_reports/ot/8ots ftp://data.pdbj.org/pub/pdb/validation_reports/ot/8ots | HTTPS FTP |
-関連構造データ
| 関連構造データ |  17183MC  8osjC  8oskC  8oslC  8ottC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 7種, 11分子 AEBFCGDHKMN
| #1: タンパク質 | 分子量: 15719.445 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: HIST1H3A, H3FA, HIST1H3B, H3FL, HIST1H3C, H3FC, HIST1H3D, H3FB, HIST1H3E, H3FD, HIST1H3F, H3FI, HIST1H3G, H3FH, HIST1H3H, H3FK, HIST1H3I, H3FF, HIST1H3J, H3FJ 発現宿主:  #2: タンパク質 | 分子量: 11676.703 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: HIST1H4A, H4/A, H4FA, HIST1H4B, H4/I, H4FI, HIST1H4C, H4/G, H4FG, HIST1H4D, H4/B, H4FB, HIST1H4E, H4/J, H4FJ, HIST1H4F, H4/C, H4FC, HIST1H4H, H4/H, H4FH, HIST1H4I, H4/M, H4FM, HIST1H4J, ...遺伝子: HIST1H4A, H4/A, H4FA, HIST1H4B, H4/I, H4FI, HIST1H4C, H4/G, H4FG, HIST1H4D, H4/B, H4FB, HIST1H4E, H4/J, H4FJ, HIST1H4F, H4/C, H4FC, HIST1H4H, H4/H, H4FH, HIST1H4I, H4/M, H4FM, HIST1H4J, H4/E, H4FE, HIST1H4K, H4/D, H4FD, HIST1H4L, H4/K, H4FK, HIST2H4A, H4/N, H4F2, H4FN, HIST2H4, HIST2H4B, H4/O, H4FO, HIST4H4 発現宿主:  #3: タンパク質 | 分子量: 14447.825 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: HIST1H2AB, H2AFM, HIST1H2AE, H2AFA / 発現宿主: Homo sapiens (ヒト) / 遺伝子: HIST1H2AB, H2AFM, HIST1H2AE, H2AFA / 発現宿主:  #4: タンパク質 | 分子量: 14088.336 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: HIST1H2BJ, H2BFR / 発現宿主: Homo sapiens (ヒト) / 遺伝子: HIST1H2BJ, H2BFR / 発現宿主:  #7: タンパク質 | | 分子量: 59814.410 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: GFP, POU5F1, OCT3, OCT4, OTF3 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P42212, UniProt: Q01860 Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: P42212, UniProt: Q01860#8: タンパク質 | | 分子量: 11501.192 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: MYC, BHLHE39 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: MYC, BHLHE39 / 発現宿主:  #9: タンパク質 | | 分子量: 9780.037 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: MAX, BHLHD4 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: MAX, BHLHD4 / 発現宿主:  |
|---|
-DNA鎖 , 2種, 2分子 IJ
| #5: DNA鎖 | 分子量: 39092.895 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成) synthetic construct (人工物) |
|---|---|
| #6: DNA鎖 | 分子量: 39298.047 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成) synthetic construct (人工物) |
-非ポリマー , 1種, 8分子 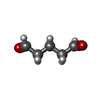
| #10: 化合物 | ChemComp-PTD / |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 由来(天然) |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| 由来(組換発現) |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| 緩衝液 | pH: 7.2 | ||||||||||||||||||||||||||||||||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | ||||||||||||||||||||||||||||||||||||||||||||||||
| 急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 500 nm |
| 撮影 | 電子線照射量: 50 e/Å2 フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) |
- 解析
解析
| EMソフトウェア |
| |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | タイプ: NONE | |||||||||||||||||||||
| 3次元再構成 | 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 102411 / 対称性のタイプ: POINT | |||||||||||||||||||||
| 原子モデル構築 |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj




















































