+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7nd1 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | First-in-class small molecule inhibitors of Polycomb Repressive Complex 1 (PRC1) RING domain | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | GENE REGULATION / Polycomb Repressive Complex 1 (PRC1) / Inhibitor | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報histone H2AK119 ubiquitin ligase activity / PRC1 complex / regulation of kidney development / RING-like zinc finger domain binding / segment specification / rostrocaudal neural tube patterning / ubiquitin-protein transferase activator activity / somatic stem cell division / embryonic skeletal system morphogenesis / regulation of adaxial/abaxial pattern formation ...histone H2AK119 ubiquitin ligase activity / PRC1 complex / regulation of kidney development / RING-like zinc finger domain binding / segment specification / rostrocaudal neural tube patterning / ubiquitin-protein transferase activator activity / somatic stem cell division / embryonic skeletal system morphogenesis / regulation of adaxial/abaxial pattern formation / sex chromatin / PcG protein complex / positive regulation of immature T cell proliferation in thymus / SUMOylation of DNA methylation proteins / gastrulation with mouth forming second / SUMOylation of RNA binding proteins / anterior/posterior axis specification / positive regulation of ubiquitin-protein transferase activity / DNA methylation-dependent constitutive heterochromatin formation / negative regulation of gene expression, epigenetic / Transcriptional Regulation by E2F6 / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / germ cell development / humoral immune response / hemopoiesis / MLL1 complex / negative regulation of apoptotic signaling pathway / cellular response to interleukin-1 / ubiquitin ligase complex / cellular response to dexamethasone stimulus / negative regulation of DNA-binding transcription factor activity / SUMOylation of DNA damage response and repair proteins / heterochromatin / positive regulation of B cell proliferation / SUMOylation of transcription cofactors / SUMOylation of chromatin organization proteins / epigenetic regulation of gene expression / Regulation of PTEN gene transcription / apoptotic signaling pathway / promoter-specific chromatin binding / euchromatin / brain development / RING-type E3 ubiquitin transferase / positive regulation of fibroblast proliferation / ubiquitin protein ligase activity / mitotic cell cycle / regulation of gene expression / Oxidative Stress Induced Senescence / gene expression / in utero embryonic development / protein ubiquitination / nuclear body / chromatin remodeling / chromatin binding / negative regulation of transcription by RNA polymerase II / zinc ion binding / nucleoplasm / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 溶液NMR / molecular dynamics | ||||||
 データ登録者 データ登録者 | Cierpicki, T. / Lund, G. / Jaremko, L. | ||||||
 引用 引用 |  ジャーナル: Nat.Chem.Biol. / 年: 2021 ジャーナル: Nat.Chem.Biol. / 年: 2021タイトル: Small-molecule inhibitors targeting Polycomb repressive complex 1 RING domain. 著者: Shukla, S. / Ying, W. / Gray, F. / Yao, Y. / Simes, M.L. / Zhao, Q. / Miao, H. / Cho, H.J. / Gonzalez-Alonso, P. / Winkler, A. / Lund, G. / Purohit, T. / Kim, E. / Zhang, X. / Ray, J.M. / He, ...著者: Shukla, S. / Ying, W. / Gray, F. / Yao, Y. / Simes, M.L. / Zhao, Q. / Miao, H. / Cho, H.J. / Gonzalez-Alonso, P. / Winkler, A. / Lund, G. / Purohit, T. / Kim, E. / Zhang, X. / Ray, J.M. / He, S. / Nikolaidis, C. / Ndoj, J. / Wang, J. / Jaremko, L. / Jaremko, M. / Ryan, R.J.H. / Guzman, M.L. / Grembecka, J. / Cierpicki, T. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
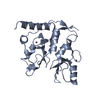
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7nd1.cif.gz 7nd1.cif.gz | 1.3 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7nd1.ent.gz pdb7nd1.ent.gz | 1.1 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7nd1.json.gz 7nd1.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7nd1_validation.pdf.gz 7nd1_validation.pdf.gz | 365.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7nd1_full_validation.pdf.gz 7nd1_full_validation.pdf.gz | 593.5 KB | 表示 | |
| XML形式データ |  7nd1_validation.xml.gz 7nd1_validation.xml.gz | 92.1 KB | 表示 | |
| CIF形式データ |  7nd1_validation.cif.gz 7nd1_validation.cif.gz | 101.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/nd/7nd1 https://data.pdbj.org/pub/pdb/validation_reports/nd/7nd1 ftp://data.pdbj.org/pub/pdb/validation_reports/nd/7nd1 ftp://data.pdbj.org/pub/pdb/validation_reports/nd/7nd1 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6wi7C 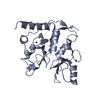 6wi8C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 12766.979 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: RNF2, BAP1, DING, HIPI3, RING1B / 発現宿主: Homo sapiens (ヒト) / 遺伝子: RNF2, BAP1, DING, HIPI3, RING1B / 発現宿主:  | ||||
|---|---|---|---|---|---|
| #2: タンパク質 | 分子量: 11727.883 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: BMI1, PCGF4, RNF51 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: BMI1, PCGF4, RNF51 / 発現宿主:  | ||||
| #3: 化合物 | ChemComp-ZN / #4: 化合物 | ChemComp-U9E / | 研究の焦点であるリガンドがあるか | Y | |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | タイプ: solution 内容: 0.12 mM [U-2H; U-13C; U-15N; CH3 ILV] Ring1B-BMI1, 95% H2O/5% D2O Label: sample1 / 溶媒系: 95% H2O/5% D2O |
|---|---|
| 試料 | 濃度: 0.12 mM / 構成要素: Ring1B-BMI1 / Isotopic labeling: [U-2H; U-13C; U-15N; CH3 ILV] |
| 試料状態 | 詳細: 50 mM sodium phosphate, pH 6.8, 150 mM NaCl, 1 mM TCEP, 10 uM ZnCl2, 5% DMSO イオン強度: 150 mM / Label: conditions1 / pH: 6.8 / 圧: 1 atm / 温度: 303.3 K |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker AVANCE III / 製造業者: Bruker / モデル: AVANCE III / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: molecular dynamics / ソフトェア番号: 1 | ||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 200 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj










 gel filtration
gel filtration



