+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 7mwk | ||||||
|---|---|---|---|---|---|---|---|
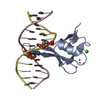
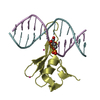
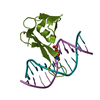
| Title | Crystal structure of MBD2 with DNA | ||||||
 Components Components |
| ||||||
 Keywords Keywords | DNA BINDING PROTEIN/DNA / DNA binding protein-DNA complex / MBD / TAM / Structural Genomics / Structural Genomics Consortium / SGC | ||||||
| Function / homology |  Function and homology information Function and homology informationsatellite DNA binding / ventricular cardiac muscle tissue development / NuRD complex / siRNA binding / maternal behavior / C2H2 zinc finger domain binding / methyl-CpG binding / DNA methylation-dependent constitutive heterochromatin formation / embryonic organ development / response to mechanical stimulus ...satellite DNA binding / ventricular cardiac muscle tissue development / NuRD complex / siRNA binding / maternal behavior / C2H2 zinc finger domain binding / methyl-CpG binding / DNA methylation-dependent constitutive heterochromatin formation / embryonic organ development / response to mechanical stimulus / positive regulation of Wnt signaling pathway / heterochromatin / RNA Polymerase I Promoter Opening / response to nutrient levels / NoRC negatively regulates rRNA expression / Wnt signaling pathway / response to estradiol / regulation of cell population proliferation / protein-containing complex assembly / molecular adaptor activity / chromatin remodeling / protein domain specific binding / negative regulation of DNA-templated transcription / mRNA binding / chromatin binding / positive regulation of DNA-templated transcription / chromatin / negative regulation of transcription by RNA polymerase II / protein-containing complex / nucleoplasm / identical protein binding / nucleus / cytosol Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human)synthetic construct (others) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.453 Å MOLECULAR REPLACEMENT / Resolution: 2.453 Å | ||||||
 Authors Authors | Liu, K. / Dong, A. / Edwards, A.M. / Arrowsmith, C.H. / Min, J. / Structural Genomics Consortium (SGC) | ||||||
 Citation Citation |  Journal: To Be Published Journal: To Be PublishedTitle: Crystal structure of MBD2 with DNA Authors: Liu, K. / Dong, A. / Edwards, A.M. / Arrowsmith, C.H. / Min, J. / Structural Genomics Consortium (SGC) | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  7mwk.cif.gz 7mwk.cif.gz | 121.1 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb7mwk.ent.gz pdb7mwk.ent.gz | 88.7 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  7mwk.json.gz 7mwk.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/mw/7mwk https://data.pdbj.org/pub/pdb/validation_reports/mw/7mwk ftp://data.pdbj.org/pub/pdb/validation_reports/mw/7mwk ftp://data.pdbj.org/pub/pdb/validation_reports/mw/7mwk | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  7mwmC  7rayC  6c2e S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Unit cell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS domain:
NCS domain segments:
|
 Movie
Movie Controller
Controller









 PDBj
PDBj





































