| 登録情報 | データベース: PDB / ID: 7lux
|
|---|
| タイトル | AALALL segment from the Nucleoprotein of SARS-CoV-2, residues 217-222, crystal form 2 |
|---|
 要素 要素 | Nucleoprotein AALALL |
|---|
 キーワード キーワード | PROTEIN FIBRIL / amyloid fibril |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
: / response to host immune response / viral RNA genome packaging / negative regulation of interferon-beta production / poly(U) RNA binding / Maturation of nucleoprotein / intracellular membraneless organelle / positive regulation of NLRP3 inflammasome complex assembly / MHC class I protein binding / CD28 dependent PI3K/Akt signaling ...: / response to host immune response / viral RNA genome packaging / negative regulation of interferon-beta production / poly(U) RNA binding / Maturation of nucleoprotein / intracellular membraneless organelle / positive regulation of NLRP3 inflammasome complex assembly / MHC class I protein binding / CD28 dependent PI3K/Akt signaling / SARS-CoV-2 targets host intracellular signalling and regulatory pathways / protein sequestering activity / VEGFR2 mediated vascular permeability / NOD1/2 Signaling Pathway / TAK1-dependent IKK and NF-kappa-B activation / DDX58/IFIH1-mediated induction of interferon-alpha/beta / molecular condensate scaffold activity / MHC class I protein complex / Interleukin-1 signaling / Interferon alpha/beta signaling / RNA stem-loop binding / viral capsid / PIP3 activates AKT signaling / Transcription of SARS-CoV-2 sgRNAs / viral nucleocapsid / host cell endoplasmic reticulum-Golgi intermediate compartment / host cell Golgi apparatus / Translation of Structural Proteins / Virion Assembly and Release / host extracellular space / Induction of Cell-Cell Fusion / Attachment and Entry / host cell perinuclear region of cytoplasm / ribonucleoprotein complex / SARS-CoV-2 activates/modulates innate and adaptive immune responses / protein homodimerization activity / RNA binding / extracellular region / identical protein binding / cytoplasm類似検索 - 分子機能 Nucleocapsid protein, betacoronavirus / Nucleocapsid protein, coronavirus / Nucleocapsid protein, C-terminal / Nucleocapsid protein, N-terminal / Nucleocapsid (N) protein, C-terminal domain, coronavirus / Nucleocapsid (N) protein, N-terminal domain, coronavirus / Coronavirus nucleocapsid / Coronavirus nucleocapsid (CoV N) protein N-terminal (NTD) domain profile. / Coronavirus nucleocapsid (CoV N) protein C-terminal (CTD) domain profile.類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Severe acute respiratory syndrome coronavirus 2 (ウイルス) Severe acute respiratory syndrome coronavirus 2 (ウイルス) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.303 Å 分子置換 / 解像度: 1.303 Å |
|---|
| Model details | Amyloid Fibril |
|---|
 データ登録者 データ登録者 | Lu, J. / Zee, C.-T. / Sawaya, M.R. / Rodriguez, J.A. / Eisenberg, D.S. |
|---|
| 資金援助 |  米国, 3件 米国, 3件 | 組織 | 認可番号 | 国 |
|---|
| Department of Energy (DOE, United States) | DE-FC02-02ER63421 |  米国 米国 | | National Science Foundation (NSF, United States) | MCB 1616265 |  米国 米国 | | National Institutes of Health/National Institute on Aging (NIH/NIA) | R01 AG048120 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Biorxiv / 年: 2021 ジャーナル: Biorxiv / 年: 2021
タイトル: Inhibition of amyloid formation of the Nucleoprotein of SARS-CoV-2.
著者: Tayeb-Fligelman, E. / Cheng, X. / Tai, C. / Bowler, J.T. / Griner, S. / Sawaya, M.R. / Seidler, P.M. / Jiang, Y.X. / Lu, J. / Rosenberg, G.M. / Salwinski, L. / Abskharon, R. / Zee, C.T. / ...著者: Tayeb-Fligelman, E. / Cheng, X. / Tai, C. / Bowler, J.T. / Griner, S. / Sawaya, M.R. / Seidler, P.M. / Jiang, Y.X. / Lu, J. / Rosenberg, G.M. / Salwinski, L. / Abskharon, R. / Zee, C.T. / Hou, K. / Li, Y. / Boyer, D.R. / Murray, K.A. / Falcon, G. / Anderson, D.H. / Cascio, D. / Saelices, L. / Damoiseaux, R. / Guo, F. / Eisenberg, D.S. |
|---|
| 履歴 | | 登録 | 2021年2月23日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2021年3月17日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2021年3月24日 | Group: Database references / カテゴリ: citation / Item: _citation.pdbx_database_id_PubMed / _citation.title |
|---|
| 改定 1.2 | 2024年3月6日 | Group: Data collection / Database references
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / citation / database_2
Item: _citation.journal_id_ISSN / _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
| 改定 1.3 | 2024年4月3日 | Group: Refinement description / カテゴリ: pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.303 Å
分子置換 / 解像度: 1.303 Å  データ登録者
データ登録者 米国, 3件
米国, 3件  引用
引用 ジャーナル: Biorxiv / 年: 2021
ジャーナル: Biorxiv / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7lux.cif.gz
7lux.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7lux.ent.gz
pdb7lux.ent.gz PDB形式
PDB形式 7lux.json.gz
7lux.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7lux_validation.pdf.gz
7lux_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7lux_full_validation.pdf.gz
7lux_full_validation.pdf.gz 7lux_validation.xml.gz
7lux_validation.xml.gz 7lux_validation.cif.gz
7lux_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/lu/7lux
https://data.pdbj.org/pub/pdb/validation_reports/lu/7lux ftp://data.pdbj.org/pub/pdb/validation_reports/lu/7lux
ftp://data.pdbj.org/pub/pdb/validation_reports/lu/7lux リンク
リンク 集合体
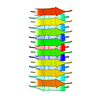
集合体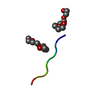

 要素
要素
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 24-ID-E / 波長: 0.9792 Å
/ ビームライン: 24-ID-E / 波長: 0.9792 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj







