| 登録情報 | データベース: PDB / ID: 7d6f
|
|---|
| タイトル | The crystal structure of ARMS-PBM/MAGI2-PDZ4 |
|---|
 要素 要素 | - Kinase D-interacting substrate of 220 kDa
- Membrane-associated guanylate kinase, WW and PDZ domain-containing protein 2
|
|---|
 キーワード キーワード | STRUCTURAL PROTEIN / PBM/PDZ interaction / scaffold protein / synaptic polaricity |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
ARMS-mediated activation / RND1 GTPase cycle / RND2 GTPase cycle / type II activin receptor binding / neuroligin clustering involved in postsynaptic membrane assembly / podocyte development / slit diaphragm / positive regulation of synaptic vesicle clustering / beta-1 adrenergic receptor binding / activin receptor binding ...ARMS-mediated activation / RND1 GTPase cycle / RND2 GTPase cycle / type II activin receptor binding / neuroligin clustering involved in postsynaptic membrane assembly / podocyte development / slit diaphragm / positive regulation of synaptic vesicle clustering / beta-1 adrenergic receptor binding / activin receptor binding / nerve growth factor signaling pathway / SMAD protein signal transduction / clathrin-dependent endocytosis / negative regulation of activin receptor signaling pathway / protein kinase regulator activity / dendrite morphogenesis / ciliary base / SMAD binding / receptor clustering / positive regulation of receptor internalization / photoreceptor outer segment / bicellular tight junction / phosphatase binding / photoreceptor inner segment / centriole / negative regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / negative regulation of cell migration / PDZ domain binding / positive regulation of neuron projection development / cellular response to nerve growth factor stimulus / late endosome / signaling receptor complex adaptor activity / in utero embryonic development / postsynapse / postsynaptic density / negative regulation of cell population proliferation / dendrite / synapse / centrosome / protein kinase binding / perinuclear region of cytoplasm / glutamatergic synapse / signal transduction / protein-containing complex / nucleus / membrane / plasma membrane / cytosol / cytoplasm類似検索 - 分子機能 : / KAP family P-loop domain / KAP family P-loop domain / Unstructured region on MAGI / Guanylate kinase, conserved site / Ankyrin repeats (many copies) / Guanylate kinase-like signature. / Guanylate kinase-like domain profile. / Guanylate kinase-like domain / Guanylate kinase/L-type calcium channel beta subunit ...: / KAP family P-loop domain / KAP family P-loop domain / Unstructured region on MAGI / Guanylate kinase, conserved site / Ankyrin repeats (many copies) / Guanylate kinase-like signature. / Guanylate kinase-like domain profile. / Guanylate kinase-like domain / Guanylate kinase/L-type calcium channel beta subunit / Guanylate kinase / Guanylate kinase homologues. / WW domain / WW/rsp5/WWP domain signature. / WW domain superfamily / WW/rsp5/WWP domain profile. / Domain with 2 conserved Trp (W) residues / WW domain / Sterile alpha motif/pointed domain superfamily / Ankyrin repeat / PDZ domain / PDZ domain profile. / Domain present in PSD-95, Dlg, and ZO-1/2. / PDZ domain / PDZ superfamily / Ankyrin repeats (3 copies) / Ankyrin repeat profile. / Ankyrin repeat region circular profile. / ankyrin repeats / Ankyrin repeat / Ankyrin repeat-containing domain superfamily / P-loop containing nucleoside triphosphate hydrolase類似検索 - ドメイン・相同性 Kinase D-interacting substrate of 220 kDa / Membrane-associated guanylate kinase, WW and PDZ domain-containing protein 2類似検索 - 構成要素 |
|---|
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ)
  Rattus norvegicus (ドブネズミ) Rattus norvegicus (ドブネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.001 Å 分子置換 / 解像度: 3.001 Å |
|---|
 データ登録者 データ登録者 | Ye, J. / Zhang, Y. / Zhong, Z. / Wang, C. |
|---|
 引用 引用 |  ジャーナル: Neurochem.Int. / 年: 2021 ジャーナル: Neurochem.Int. / 年: 2021
タイトル: Crystal structure of the PDZ4 domain of MAGI2 in complex with PBM of ARMS reveals a canonical PDZ recognition mode.
著者: Zhang, Y. / Zhong, Z. / Ye, J. / Wang, C. |
|---|
| 履歴 | | 登録 | 2020年9月30日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2020年11月4日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 2.0 | 2021年9月1日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Author supporting evidence / Data collection / Database references / Derived calculations / Non-polymer description / Refinement description / Structure summary
カテゴリ: atom_site / atom_site_anisotrop ...atom_site / atom_site_anisotrop / audit_author / chem_comp / citation_author / database_2 / entity / pdbx_entity_instance_feature / pdbx_entity_nonpoly / pdbx_entry_details / pdbx_nonpoly_scheme / pdbx_poly_seq_scheme / pdbx_refine_tls / pdbx_refine_tls_group / pdbx_struct_assembly_gen / pdbx_struct_assembly_prop / pdbx_struct_sheet_hbond / pdbx_validate_rmsd_angle / pdbx_validate_symm_contact / pdbx_validate_torsion / refine / refine_hist / refine_ls_shell / reflns / software / struct_asym / struct_conf / struct_sheet / struct_sheet_order / struct_sheet_range
Item: _audit_author.name / _chem_comp.formula ..._audit_author.name / _chem_comp.formula / _chem_comp.formula_weight / _chem_comp.id / _chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.pdbx_synonyms / _chem_comp.type / _citation_author.name / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_poly_seq_scheme.auth_mon_id / _pdbx_refine_tls.L[1][1] / _pdbx_refine_tls.L[1][2] / _pdbx_refine_tls.L[1][3] / _pdbx_refine_tls.L[2][2] / _pdbx_refine_tls.L[2][3] / _pdbx_refine_tls.L[3][3] / _pdbx_refine_tls.S[1][1] / _pdbx_refine_tls.S[1][2] / _pdbx_refine_tls.S[1][3] / _pdbx_refine_tls.S[2][1] / _pdbx_refine_tls.S[2][2] / _pdbx_refine_tls.S[2][3] / _pdbx_refine_tls.S[3][1] / _pdbx_refine_tls.S[3][2] / _pdbx_refine_tls.S[3][3] / _pdbx_refine_tls.T[1][1] / _pdbx_refine_tls.T[1][2] / _pdbx_refine_tls.T[1][3] / _pdbx_refine_tls.T[2][2] / _pdbx_refine_tls.T[2][3] / _pdbx_refine_tls.T[3][3] / _pdbx_refine_tls.origin_x / _pdbx_refine_tls.origin_y / _pdbx_refine_tls.origin_z / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_struct_assembly_prop.value / _refine.B_iso_max / _refine.B_iso_mean / _refine.B_iso_min / _refine.ls_R_factor_R_free / _refine.ls_R_factor_R_work / _refine.ls_R_factor_obs / _refine.ls_d_res_high / _refine.ls_number_reflns_R_free / _refine.ls_number_reflns_R_work / _refine.ls_number_reflns_obs / _refine.ls_percent_reflns_R_free / _refine.ls_percent_reflns_obs / _refine.overall_SU_ML / _refine.pdbx_ls_sigma_F / _refine.pdbx_overall_phase_error / _refine.pdbx_starting_model / _refine_hist.d_res_high / _refine_hist.number_atoms_solvent / _refine_hist.number_atoms_total / _refine_hist.pdbx_B_iso_mean_ligand / _refine_hist.pdbx_B_iso_mean_solvent / _refine_hist.pdbx_number_atoms_ligand / _refine_ls_shell.R_factor_R_free / _refine_ls_shell.R_factor_R_work / _refine_ls_shell.d_res_high / _refine_ls_shell.d_res_low / _refine_ls_shell.number_reflns_R_free / _refine_ls_shell.number_reflns_R_work / _refine_ls_shell.percent_reflns_obs / _reflns.B_iso_Wilson_estimate / _reflns.d_resolution_high / _reflns.pdbx_number_measured_all / _software.version / _struct_conf.end_auth_comp_id / _struct_conf.end_auth_seq_id / _struct_conf.end_label_comp_id / _struct_conf.end_label_seq_id / _struct_conf.pdbx_PDB_helix_length / _struct_sheet.number_strands
解説: Real space R-factor
詳細: To cut off the high resolution to 3.0 Angstrom when running the refinement program.
Provider: author / タイプ: Coordinate replacement |
|---|
| 改定 2.1 | 2021年11月17日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _citation_author.name |
|---|
| 改定 2.2 | 2023年11月29日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報

 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 3.001 Å
分子置換 / 解像度: 3.001 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Neurochem.Int. / 年: 2021
ジャーナル: Neurochem.Int. / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 7d6f.cif.gz
7d6f.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb7d6f.ent.gz
pdb7d6f.ent.gz PDB形式
PDB形式 7d6f.json.gz
7d6f.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 7d6f_validation.pdf.gz
7d6f_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 7d6f_full_validation.pdf.gz
7d6f_full_validation.pdf.gz 7d6f_validation.xml.gz
7d6f_validation.xml.gz 7d6f_validation.cif.gz
7d6f_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/d6/7d6f
https://data.pdbj.org/pub/pdb/validation_reports/d6/7d6f ftp://data.pdbj.org/pub/pdb/validation_reports/d6/7d6f
ftp://data.pdbj.org/pub/pdb/validation_reports/d6/7d6f
 リンク
リンク 集合体
集合体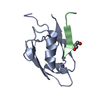
 要素
要素



 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL19U1 / 波長: 0.97776 Å
/ ビームライン: BL19U1 / 波長: 0.97776 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj





