| ソフトウェア | | 名称 | バージョン | 分類 |
|---|
| REFMAC | 5.8.0258| 精密化 | | XDS | | データ削減 | | Aimless | 0.7.4| データスケーリング | | PHASER | | 位相決定 | | PDB_EXTRACT | 3.25 | データ抽出 | | |
|
|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換
開始モデル: 6JJH
解像度: 1.21→27.69 Å / Cor.coef. Fo:Fc: 0.963 / Cor.coef. Fo:Fc free: 0.953 / SU B: 0.926 / SU ML: 0.04 / 交差検証法: FREE R-VALUE / ESU R: 0.056 / ESU R Free: 0.056 / 詳細: Hydrogens have been added in their riding positions
| Rfactor | 反射数 | %反射 | Selection details |
|---|
| Rfree | 0.217 | 1025 | 4.829 % | Random Selection |
|---|
| Rwork | 0.199 | 20202 | - | - |
|---|
| all | 0.2 | - | - | - |
|---|
| obs | 0.1999 | 21227 | 93.879 % | - |
|---|
|
|---|
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK BULK SOLVENT |
|---|
| 原子変位パラメータ | Biso mean: 17.82 Å2
| Baniso -1 | Baniso -2 | Baniso -3 |
|---|
| 1- | -0.412 Å2 | -0 Å2 | -0.107 Å2 |
|---|
| 2- | - | 1.5 Å2 | -0 Å2 |
|---|
| 3- | - | - | -0.833 Å2 |
|---|
|
|---|
| 精密化ステップ | サイクル: final / 解像度: 1.21→27.69 Å
| タンパク質 | 核酸 | リガンド | 溶媒 | 全体 |
|---|
| 原子数 | 0 | 606 | 119 | 97 | 822 |
|---|
| Biso mean | - | - | 18.26 | 27.65 | - |
|---|
| 残基数 | - | - | - | - | 28 |
|---|
|
|---|
| 拘束条件 | | Refine-ID | タイプ | Dev ideal | Dev ideal target | 数 |
|---|
| X-RAY DIFFRACTION | r_bond_refined_d| 0.025 | 0.012 | 853 | | X-RAY DIFFRACTION | r_bond_other_d| 0.004 | 0.02 | 290 | | X-RAY DIFFRACTION | r_angle_refined_deg| 2.001 | 1.489 | 1283 | | X-RAY DIFFRACTION | r_angle_other_deg| 1.82 | 3 | 696 | | X-RAY DIFFRACTION | r_chiral_restr| 0.12 | 0.2 | 116 | | X-RAY DIFFRACTION | r_gen_planes_refined| 0.032 | 0.02 | 418 | | X-RAY DIFFRACTION | r_gen_planes_other| 0.005 | 0.02 | 168 | | X-RAY DIFFRACTION | r_nbd_refined| 0.237 | 0.2 | 138 | | X-RAY DIFFRACTION | r_symmetry_nbd_other| 0.228 | 0.2 | 342 | | X-RAY DIFFRACTION | r_nbtor_refined| 0.274 | 0.2 | 319 | | X-RAY DIFFRACTION | r_symmetry_nbtor_other| 0.063 | 0.2 | 142 | | X-RAY DIFFRACTION | r_xyhbond_nbd_refined| 0.12 | 0.2 | 67 | | X-RAY DIFFRACTION | r_metal_ion_refined| 0.063 | 0.2 | 2 | | X-RAY DIFFRACTION | r_symmetry_nbd_refined| 0.129 | 0.2 | 32 | | X-RAY DIFFRACTION | r_nbd_other| 0.203 | 0.2 | 23 | | X-RAY DIFFRACTION | r_symmetry_xyhbond_nbd_refined| 0.068 | 0.2 | 8 | | X-RAY DIFFRACTION | r_scbond_it| 1.917 | 1.747 | 853 | | X-RAY DIFFRACTION | r_scbond_other| 1.832 | 1.738 | 724 | | X-RAY DIFFRACTION | r_scangle_it| 2.943 | 2.633 | 1283 | | X-RAY DIFFRACTION | r_scangle_other| 2.828 | 2.647 | 1104 | | X-RAY DIFFRACTION | r_lrange_it| 4.1 | 26.179 | 3561 | | X-RAY DIFFRACTION | r_lrange_other| 3.947 | 27.386 | 2942 | | | | | | | | | | | | | | | | | | | | | | |
|
|---|
| LS精密化 シェル | | 解像度 (Å) | Rfactor Rfree | Num. reflection Rfree | Rfactor Rwork | Num. reflection Rwork | Refine-ID | % reflection obs (%) |
|---|
| 1.21-1.241 | 0.319 | 56 | 0.337 | 917 | X-RAY DIFFRACTION | 58.5792 | | 1.241-1.275 | 0.372 | 49 | 0.302 | 1127 | X-RAY DIFFRACTION | 72.5478 | | 1.275-1.312 | 0.329 | 66 | 0.285 | 1309 | X-RAY DIFFRACTION | 87.6354 | | 1.312-1.353 | 0.287 | 69 | 0.268 | 1420 | X-RAY DIFFRACTION | 96.4378 | | 1.353-1.397 | 0.31 | 71 | 0.235 | 1404 | X-RAY DIFFRACTION | 100 | | 1.397-1.446 | 0.308 | 49 | 0.235 | 1375 | X-RAY DIFFRACTION | 100 | | 1.446-1.5 | 0.224 | 50 | 0.234 | 1315 | X-RAY DIFFRACTION | 100 | | 1.5-1.561 | 0.259 | 48 | 0.205 | 1318 | X-RAY DIFFRACTION | 99.9268 | | 1.561-1.631 | 0.179 | 66 | 0.182 | 1224 | X-RAY DIFFRACTION | 100 | | 1.631-1.71 | 0.175 | 63 | 0.176 | 1157 | X-RAY DIFFRACTION | 100 | | 1.71-1.802 | 0.215 | 61 | 0.172 | 1097 | X-RAY DIFFRACTION | 100 | | 1.802-1.911 | 0.205 | 64 | 0.193 | 1039 | X-RAY DIFFRACTION | 100 | | 1.911-2.042 | 0.223 | 56 | 0.188 | 995 | X-RAY DIFFRACTION | 100 | | 2.042-2.205 | 0.191 | 47 | 0.187 | 925 | X-RAY DIFFRACTION | 100 | | 2.205-2.414 | 0.178 | 50 | 0.204 | 831 | X-RAY DIFFRACTION | 100 | | 2.414-2.697 | 0.232 | 62 | 0.193 | 759 | X-RAY DIFFRACTION | 100 | | 2.697-3.109 | 0.192 | 42 | 0.202 | 677 | X-RAY DIFFRACTION | 100 | | 3.109-3.797 | 0.208 | 36 | 0.168 | 574 | X-RAY DIFFRACTION | 100 | | 3.797-5.325 | 0.2 | 16 | 0.164 | 457 | X-RAY DIFFRACTION | 100 | | 5.325-27.69 | 0.392 | 4 | 0.255 | 282 | X-RAY DIFFRACTION | 100 |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Pseudorabies virus Ea (ウイルス)
Pseudorabies virus Ea (ウイルス) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.21 Å
分子置換 / 解像度: 1.21 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Biochimie / 年: 2024
ジャーナル: Biochimie / 年: 2024 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6xrq.cif.gz
6xrq.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6xrq.ent.gz
pdb6xrq.ent.gz PDB形式
PDB形式 6xrq.json.gz
6xrq.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6xrq_validation.pdf.gz
6xrq_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6xrq_full_validation.pdf.gz
6xrq_full_validation.pdf.gz 6xrq_validation.xml.gz
6xrq_validation.xml.gz 6xrq_validation.cif.gz
6xrq_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/xr/6xrq
https://data.pdbj.org/pub/pdb/validation_reports/xr/6xrq ftp://data.pdbj.org/pub/pdb/validation_reports/xr/6xrq
ftp://data.pdbj.org/pub/pdb/validation_reports/xr/6xrq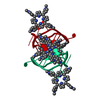
 リンク
リンク 集合体
集合体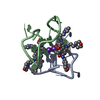
 要素
要素 Pseudorabies virus Ea (ウイルス)
Pseudorabies virus Ea (ウイルス) X線回折 / Absorpt correction T max: 1 / Absorpt correction T min: 0.949905760669 / Absorpt correction type: empirical
X線回折 / Absorpt correction T max: 1 / Absorpt correction T min: 0.949905760669 / Absorpt correction type: empirical 試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I24 / 波長: 0.96864 Å
/ ビームライン: I24 / 波長: 0.96864 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj