[English] 日本語
 Yorodumi
Yorodumi- PDB-6wyd: CRYSTAL STRUCTURE OF MYELOPEROXIDASE SUBFORM C (MPO) COMPLEX WITH... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6wyd | ||||||
|---|---|---|---|---|---|---|---|
| Title | CRYSTAL STRUCTURE OF MYELOPEROXIDASE SUBFORM C (MPO) COMPLEX WITH Compound-12 (AKA; 7-benzyl-1H-[1,2,3]triazolo[4,5-b]pyrid | ||||||
 Components Components | (Myeloperoxidase ...) x 2 | ||||||
 Keywords Keywords | OXIDOREDUCTASE / MYELOPEROXIDASE | ||||||
| Function / homology |  Function and homology information Function and homology informationmyeloperoxidase / hypochlorous acid biosynthetic process / Events associated with phagocytolytic activity of PMN cells / phagocytic vesicle lumen / response to gold nanoparticle / response to yeast / respiratory burst involved in defense response / low-density lipoprotein particle remodeling / azurophil granule / response to food ...myeloperoxidase / hypochlorous acid biosynthetic process / Events associated with phagocytolytic activity of PMN cells / phagocytic vesicle lumen / response to gold nanoparticle / response to yeast / respiratory burst involved in defense response / low-density lipoprotein particle remodeling / azurophil granule / response to food / defense response to fungus / response to mechanical stimulus / removal of superoxide radicals / secretory granule / hydrogen peroxide catabolic process / peroxidase activity / defense response / azurophil granule lumen / heparin binding / response to oxidative stress / response to lipopolysaccharide / lysosome / defense response to bacterium / intracellular membrane-bounded organelle / heme binding / chromatin binding / Neutrophil degranulation / negative regulation of apoptotic process / extracellular space / extracellular exosome / extracellular region / nucleoplasm / metal ion binding / nucleus Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.55 Å MOLECULAR REPLACEMENT / Resolution: 2.55 Å | ||||||
 Authors Authors | Khan, J.A. | ||||||
 Citation Citation |  Journal: Bioorg.Med.Chem. / Year: 2020 Journal: Bioorg.Med.Chem. / Year: 2020Title: Discovery and structure activity relationships of 7-benzyl triazolopyridines as stable, selective, and reversible inhibitors of myeloperoxidase. Authors: Shaw, S.A. / Vokits, B.P. / Dilger, A.K. / Viet, A. / Clark, C.G. / Abell, L.M. / Locke, G.A. / Duke, G. / Kopcho, L.M. / Dongre, A. / Gao, J. / Krishnakumar, A. / Jusuf, S. / Khan, J. / ...Authors: Shaw, S.A. / Vokits, B.P. / Dilger, A.K. / Viet, A. / Clark, C.G. / Abell, L.M. / Locke, G.A. / Duke, G. / Kopcho, L.M. / Dongre, A. / Gao, J. / Krishnakumar, A. / Jusuf, S. / Khan, J. / Spronk, S.A. / Basso, M.D. / Zhao, L. / Cantor, G.H. / Onorato, J.M. / Wexler, R.R. / Duclos, F. / Kick, E.K. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6wyd.cif.gz 6wyd.cif.gz | 470.6 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6wyd.ent.gz pdb6wyd.ent.gz | 384.2 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6wyd.json.gz 6wyd.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/wy/6wyd https://data.pdbj.org/pub/pdb/validation_reports/wy/6wyd ftp://data.pdbj.org/pub/pdb/validation_reports/wy/6wyd ftp://data.pdbj.org/pub/pdb/validation_reports/wy/6wyd | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  6wxzC  6wy0C  6wy5C  6wy7C  6qj2S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| Unit cell |
|
- Components
Components
-Myeloperoxidase ... , 2 types, 8 molecules ADFHBEGI
| #1: Protein | Mass: 11974.420 Da / Num. of mol.: 4 / Source method: isolated from a natural source / Source: (natural)  Homo sapiens (human) / Cell: Blood Neutrophill / References: UniProt: P05164, myeloperoxidase Homo sapiens (human) / Cell: Blood Neutrophill / References: UniProt: P05164, myeloperoxidase#2: Protein | Mass: 53305.266 Da / Num. of mol.: 4 / Source method: isolated from a natural source / Source: (natural)  Homo sapiens (human) / References: UniProt: P05164, myeloperoxidase Homo sapiens (human) / References: UniProt: P05164, myeloperoxidase |
|---|
-Sugars , 4 types, 35 molecules 






| #4: Sugar | ChemComp-NAG / #5: Sugar | ChemComp-FUC / #7: Sugar | ChemComp-BMA / #8: Sugar | ChemComp-MAN / |
|---|
-Non-polymers , 5 types, 280 molecules 

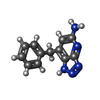






| #3: Chemical | ChemComp-CL / #6: Chemical | ChemComp-HEC / #9: Chemical | ChemComp-7GD / #10: Chemical | ChemComp-CA / #11: Water | ChemComp-HOH / | |
|---|
-Details
| Has ligand of interest | Y |
|---|---|
| Has protein modification | Y |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.39 Å3/Da / Density % sol: 48.64 % |
|---|---|
| Crystal grow | Temperature: 296 K / Method: vapor diffusion, hanging drop Details: 0.1M Hepes pH 7.5, 150mM NaCl, 20-25%(V/V)PEG3350.Crystals were cryoprotected by supplementing the mother liquor with 15% (v/v) ethylene glycol and harvested by flash-cooling in liquid nitrogen PH range: 7.5? |
-Data collection
| Diffraction | Mean temperature: 100 K / Serial crystal experiment: N |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 17-ID / Wavelength: 1 Å / Beamline: 17-ID / Wavelength: 1 Å |
| Detector | Type: DECTRIS PILATUS 6M / Detector: PIXEL / Date: Mar 2, 2011 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 2.55→200 Å / Num. obs: 82041 / % possible obs: 100 % / Redundancy: 6.7 % / Biso Wilson estimate: 62.29 Å2 / Rmerge(I) obs: 0.072 / Net I/σ(I): 20.7 |
| Reflection shell | Resolution: 2.55→2.68 Å / Redundancy: 7 % / Rmerge(I) obs: 0.488 / Mean I/σ(I) obs: 4.4 / Num. unique obs: 5972 / % possible all: 100 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 6QJ2 Resolution: 2.55→115.53 Å / Cor.coef. Fo:Fc: 0.9357 / Cor.coef. Fo:Fc free: 0.8939 / SU R Cruickshank DPI: 0.824 / Cross valid method: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.732 / SU Rfree Blow DPI: 0.301 / SU Rfree Cruickshank DPI: 0.309
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 128.16 Å2 / Biso mean: 50.55 Å2 / Biso min: 13.98 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.322 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 2.55→115.53 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 2.55→2.62 Å / Rfactor Rfree error: 0 / Total num. of bins used: 20
|
 Movie
Movie Controller
Controller












 PDBj
PDBj



















