| 登録情報 | データベース: PDB / ID: 6vtp
|
|---|
| タイトル | Crystal structure of G16C human Galectin-7 mutant |
|---|
 要素 要素 | Galectin-7 |
|---|
 キーワード キーワード | SUGAR BINDING PROTEIN / Human Galectin-7 / G16C mutant / disulfide bond |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Differentiation of Keratinocytes in Interfollicular Epidermis in Mammalian Skin / heterophilic cell-cell adhesion / carbohydrate binding / apoptotic process / extracellular space / extracellular exosome / nucleus / cytoplasm類似検索 - 分子機能 Galectin-like / Galactoside-binding lectin / Galectin / Galectin, carbohydrate recognition domain / Galactoside-binding lectin / Galactoside-binding lectin (galectin) domain profile. / Concanavalin A-like lectin/glucanase domain superfamily類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.3 Å 分子置換 / 解像度: 2.3 Å |
|---|
 データ登録者 データ登録者 | Pham, N.T.H. / Calmettes, C. / Doucet, N. |
|---|
| 資金援助 |  カナダ, カナダ,  米国, 6件 米国, 6件 | 組織 | 認可番号 | 国 |
|---|
| Natural Sciences and Engineering Research Council (NSERC, Canada) | RGPIN 2016-05557 |  カナダ カナダ | | National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | R01GM105978 |  米国 米国 | | Fonds de Recherche du Quebec - Sante (FRQS) | Research Scholar Senior Career Award (281993) |  カナダ カナダ | | Fonds de Recherche du Quebec - Sante (FRQS) | Junior 1 (251848) |  カナダ カナダ | | Natural Sciences and Engineering Research Council (NSERC, Canada) | RGPIN-2017-06091 |  カナダ カナダ | | Fonds de Recherche du Quebec - Sante (FRQS) | Doctoral Training scholarship (287239) |  カナダ カナダ |
|
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2021 ジャーナル: J.Biol.Chem. / 年: 2021
タイトル: Perturbing dimer interactions and allosteric communication modulates the immunosuppressive activity of human galectin-7.
著者: Pham, N.T.H. / Letourneau, M. / Fortier, M. / Begin, G. / Al-Abdul-Wahid, M.S. / Pucci, F. / Folch, B. / Rooman, M. / Chatenet, D. / St-Pierre, Y. / Lague, P. / Calmettes, C. / Doucet, N. |
|---|
| 履歴 | | 登録 | 2020年2月13日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2021年8月25日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2021年11月17日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year |
|---|
| 改定 1.2 | 2021年11月24日 | Group: Database references / カテゴリ: citation / Item: _citation.journal_volume |
|---|
| 改定 1.3 | 2023年10月11日 | Group: Data collection / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / pdbx_initial_refinement_model / struct_ncs_dom_lim
Item: _struct_ncs_dom_lim.beg_auth_comp_id / _struct_ncs_dom_lim.beg_label_asym_id ..._struct_ncs_dom_lim.beg_auth_comp_id / _struct_ncs_dom_lim.beg_label_asym_id / _struct_ncs_dom_lim.beg_label_comp_id / _struct_ncs_dom_lim.beg_label_seq_id / _struct_ncs_dom_lim.end_auth_comp_id / _struct_ncs_dom_lim.end_label_asym_id / _struct_ncs_dom_lim.end_label_comp_id / _struct_ncs_dom_lim.end_label_seq_id |
|---|
| 改定 1.4 | 2024年11月20日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature
Item: _pdbx_entry_details.has_protein_modification |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.3 Å
分子置換 / 解像度: 2.3 Å  データ登録者
データ登録者 カナダ,
カナダ,  米国, 6件
米国, 6件  引用
引用 ジャーナル: J.Biol.Chem. / 年: 2021
ジャーナル: J.Biol.Chem. / 年: 2021 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6vtp.cif.gz
6vtp.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6vtp.ent.gz
pdb6vtp.ent.gz PDB形式
PDB形式 6vtp.json.gz
6vtp.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6vtp_validation.pdf.gz
6vtp_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6vtp_full_validation.pdf.gz
6vtp_full_validation.pdf.gz 6vtp_validation.xml.gz
6vtp_validation.xml.gz 6vtp_validation.cif.gz
6vtp_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/vt/6vtp
https://data.pdbj.org/pub/pdb/validation_reports/vt/6vtp ftp://data.pdbj.org/pub/pdb/validation_reports/vt/6vtp
ftp://data.pdbj.org/pub/pdb/validation_reports/vt/6vtp リンク
リンク 集合体
集合体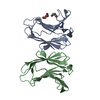
 ムービー
ムービー コントローラー
コントローラー















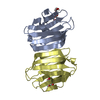


 PDBj
PDBj