+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6t48 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bovine enterovirus F3 in complex with glutathione and a Cysteinylglycine dipeptide | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | VIRUS / enterovirus F3 / enterovirus capsid assembly / glutathione / Cys-Gly dipeptide | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ribonucleoside triphosphate phosphatase activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / nucleoside-triphosphate phosphatase / channel activity / monoatomic ion transmembrane transport ...ribonucleoside triphosphate phosphatase activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / nucleoside-triphosphate phosphatase / channel activity / monoatomic ion transmembrane transport / RNA helicase activity / symbiont-mediated suppression of host innate immune response / endocytosis involved in viral entry into host cell / symbiont-mediated activation of host autophagy / RNA-directed RNA polymerase / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription / virion attachment to host cell / host cell nucleus / structural molecule activity / proteolysis / RNA binding / zinc ion binding / ATP binding / membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Enterovirus F (エンテロウイルス) Enterovirus F (エンテロウイルス) | ||||||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.17 Å 分子置換 / 解像度: 2.17 Å | ||||||||||||
 データ登録者 データ登録者 | Duyvesteyn, H.M.E. / Ren, J. / Walter, T.S. / Fry, E.E. / Stuart, D.I. | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2020 ジャーナル: Commun Biol / 年: 2020タイトル: Glutathione facilitates enterovirus assembly by binding at a druggable pocket. 著者: Duyvesteyn, H.M.E. / Ren, J. / Walter, T.S. / Fry, E.E. / Stuart, D.I. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6t48.cif.gz 6t48.cif.gz | 188.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6t48.ent.gz pdb6t48.ent.gz | 146.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6t48.json.gz 6t48.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6t48_validation.pdf.gz 6t48_validation.pdf.gz | 857.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6t48_full_validation.pdf.gz 6t48_full_validation.pdf.gz | 864 KB | 表示 | |
| XML形式データ |  6t48_validation.xml.gz 6t48_validation.xml.gz | 37.2 KB | 表示 | |
| CIF形式データ |  6t48_validation.cif.gz 6t48_validation.cif.gz | 54.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/t4/6t48 https://data.pdbj.org/pub/pdb/validation_reports/t4/6t48 ftp://data.pdbj.org/pub/pdb/validation_reports/t4/6t48 ftp://data.pdbj.org/pub/pdb/validation_reports/t4/6t48 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | x 60
| ||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
|
- 要素
要素
-タンパク質 , 4種, 4分子 ABCD
| #1: タンパク質 | 分子量: 30247.699 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Enterovirus F (エンテロウイルス) Enterovirus F (エンテロウイルス)参照: UniProt: Q2LKZ0, picornain 2A, nucleoside-triphosphate phosphatase, picornain 3C, RNA-directed RNA polymerase |
|---|---|
| #2: タンパク質 | 分子量: 26757.965 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Enterovirus F (エンテロウイルス) Enterovirus F (エンテロウイルス)参照: UniProt: Q2LKZ0, picornain 2A, nucleoside-triphosphate phosphatase, picornain 3C, RNA-directed RNA polymerase |
| #3: タンパク質 | 分子量: 27149.947 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Enterovirus F (エンテロウイルス) Enterovirus F (エンテロウイルス)参照: UniProt: Q2LKZ0, picornain 2A, nucleoside-triphosphate phosphatase, picornain 3C, RNA-directed RNA polymerase |
| #4: タンパク質 | 分子量: 7705.456 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  Enterovirus F (エンテロウイルス) Enterovirus F (エンテロウイルス)参照: UniProt: Q2LKZ0, picornain 2A, nucleoside-triphosphate phosphatase, picornain 3C, RNA-directed RNA polymerase |
-非ポリマー , 9種, 629分子 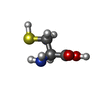
















| #5: 化合物 | ChemComp-CYS / | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| #6: 化合物 | ChemComp-STE / | ||||||||||
| #7: 化合物 | ChemComp-GSH / | ||||||||||
| #8: 化合物 | ChemComp-SO4 / #9: 化合物 | #10: 化合物 | ChemComp-K / #11: 化合物 | ChemComp-GLY / | #12: 化合物 | #13: 水 | ChemComp-HOH / | |
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / 詳細: 1.5 M Ammonium Sulfate and 0.1 M Tris at pH 8.5 |
|---|
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I03 / 波長: 0.976 Å / ビームライン: I03 / 波長: 0.976 Å |
| 検出器 | タイプ: DECTRIS EIGER2 XE 16M / 検出器: PIXEL / 日付: 2019年5月12日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.976 Å / 相対比: 1 |
| 反射 | 解像度: 2.17→20 Å / Num. obs: 908128 / % possible obs: 82.6 % / 冗長度: 3.7 % / Rmerge(I) obs: 0.374 / Rpim(I) all: 0.187 / Net I/σ(I): 5.7 |
| 反射 シェル | 解像度: 2.17→2.2 Å / Rmerge(I) obs: 0.798 / Mean I/σ(I) obs: 3.6 / Num. unique obs: 37002 / CC1/2: 0.275 / Rpim(I) all: 0.787 / % possible all: 68 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 5OSN 解像度: 2.17→19.98 Å / Rfactor Rfree error: 0.001 / Data cutoff high absF: 32369612.68 / Data cutoff low absF: 0 / 交差検証法: THROUGHOUT / σ(F): 0 / 詳細: BULK SOLVENT MODEL USED.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | Bsol: 35.72 Å2 / ksol: 0.35 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 18.5 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 2.17→19.98 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | NCS model details: CONSTR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.17→2.25 Å / Rfactor Rfree error: 0.005 / Total num. of bins used: 6
|
 ムービー
ムービー コントローラー
コントローラー


























 PDBj
PDBj
















