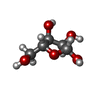
Entry Database : PDB / ID : 6o0qTitle Crystal structure of the TIR domain from human SARM1 in complex with ribose Sterile alpha and TIR motif-containing protein 1 Keywords / Function / homology Function Domain/homology Component
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / Biological species Homo sapiens (human)Method / / / Resolution : 1.8 Å Authors Horsefield, S. / Burdett, H. / Zhang, X. / Manik, M.K. / Shi, Y. / Chen, J. / Tiancong, Q. / Gilley, J. / Lai, J. / Gu, W. ...Horsefield, S. / Burdett, H. / Zhang, X. / Manik, M.K. / Shi, Y. / Chen, J. / Tiancong, Q. / Gilley, J. / Lai, J. / Gu, W. / Rank, M. / Deerain, N. / Casey, L. / Ericsson, D.J. / Foley, G. / Hughes, R.O. / Bosanac, T. / von Itzstein, M. / Rathjen, J.P. / Nanson, J.D. / Boden, M. / Dry, I.B. / Williams, S.J. / Staskawicz, B.J. / Coleman, M.P. / Ve, T. / Dodds, P.N. / Kobe, B. Funding support Organization Grant number Country National Health and Medical Research Council (NHMRC, Australia) 1107804 National Health and Medical Research Council (NHMRC, Australia) 1160570 National Health and Medical Research Council (NHMRC, Australia) 1071659 National Health and Medical Research Council (NHMRC, Australia) 1108859 Australian Research Council (ARC) DP160102244 Australian Research Council (ARC) DP190102526
Journal : Science / Year : 2019Title : NAD+cleavage activity by animal and plant TIR domains in cell death pathways.Authors: Horsefield, S. / Burdett, H. / Zhang, X. / Manik, M.K. / Shi, Y. / Chen, J. / Qi, T. / Gilley, J. / Lai, J.S. / Rank, M.X. / Casey, L.W. / Gu, W. / Ericsson, D.J. / Foley, G. / Hughes, R.O. ... Authors : Horsefield, S. / Burdett, H. / Zhang, X. / Manik, M.K. / Shi, Y. / Chen, J. / Qi, T. / Gilley, J. / Lai, J.S. / Rank, M.X. / Casey, L.W. / Gu, W. / Ericsson, D.J. / Foley, G. / Hughes, R.O. / Bosanac, T. / von Itzstein, M. / Rathjen, J.P. / Nanson, J.D. / Boden, M. / Dry, I.B. / Williams, S.J. / Staskawicz, B.J. / Coleman, M.P. / Ve, T. / Dodds, P.N. / Kobe, B. History Deposition Feb 17, 2019 Deposition site / Processing site Revision 1.0 Sep 4, 2019 Provider / Type Revision 1.1 Jan 1, 2020 Group / Data collection / Category / pdbx_audit_supportItem / _pdbx_audit_support.funding_organizationRevision 2.0 Jul 29, 2020 Group Atomic model / Data collection ... Atomic model / Data collection / Derived calculations / Structure summary Category atom_site / chem_comp ... atom_site / chem_comp / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / struct_site / struct_site_gen Item _atom_site.auth_atom_id / _atom_site.label_atom_id ... _atom_site.auth_atom_id / _atom_site.label_atom_id / _chem_comp.name / _chem_comp.type / _entity.pdbx_description / _pdbx_entity_nonpoly.name Description / Provider / Type Revision 2.1 Mar 13, 2024 Group / Database references / Structure summaryCategory chem_comp / chem_comp_atom ... chem_comp / chem_comp_atom / chem_comp_bond / database_2 Item / _database_2.pdbx_DOI / _database_2.pdbx_database_accession
Show all Show less
 Yorodumi
Yorodumi Open data
Open data Basic information
Basic information Components
Components Keywords
Keywords Function and homology information
Function and homology information Homo sapiens (human)
Homo sapiens (human) X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  SYNCHROTRON /
SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.8 Å
MOLECULAR REPLACEMENT / Resolution: 1.8 Å  Authors
Authors Australia, 6items
Australia, 6items  Citation
Citation Journal: Science / Year: 2019
Journal: Science / Year: 2019 Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 6o0q.cif.gz
6o0q.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb6o0q.ent.gz
pdb6o0q.ent.gz PDB format
PDB format 6o0q.json.gz
6o0q.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads https://data.pdbj.org/pub/pdb/validation_reports/o0/6o0q
https://data.pdbj.org/pub/pdb/validation_reports/o0/6o0q ftp://data.pdbj.org/pub/pdb/validation_reports/o0/6o0q
ftp://data.pdbj.org/pub/pdb/validation_reports/o0/6o0q






 Links
Links Assembly
Assembly

 Components
Components Homo sapiens (human) / Gene: SARM1, KIAA0524, SAMD2, SARM / Production host:
Homo sapiens (human) / Gene: SARM1, KIAA0524, SAMD2, SARM / Production host: 
 X-RAY DIFFRACTION / Number of used crystals: 1
X-RAY DIFFRACTION / Number of used crystals: 1  Sample preparation
Sample preparation SYNCHROTRON / Site:
SYNCHROTRON / Site:  Australian Synchrotron
Australian Synchrotron  / Beamline: MX2 / Wavelength: 0.9537 Å
/ Beamline: MX2 / Wavelength: 0.9537 Å Processing
Processing MOLECULAR REPLACEMENT / Resolution: 1.8→48.28 Å / SU ML: 0.21 / Cross valid method: FREE R-VALUE / σ(F): 1.34 / Phase error: 23.23
MOLECULAR REPLACEMENT / Resolution: 1.8→48.28 Å / SU ML: 0.21 / Cross valid method: FREE R-VALUE / σ(F): 1.34 / Phase error: 23.23  Movie
Movie Controller
Controller











 PDBj
PDBj