| 登録情報 | データベース: PDB / ID: 6hc8
|
|---|
| タイトル | Factor Inhibiting HIF (FIH) in complex with zinc, NOG and TRPA1 (313-339) |
|---|
 要素 要素 | - Hypoxia-inducible factor 1-alpha inhibitor
- Transient receptor potential cation channel subfamily A member 1
|
|---|
 キーワード キーワード | GENE REGULATION / NON-HEME / DIOXYGENASE / OXYGENASE / METAL-BINDING / TRANSCRIPTION / DSBH / FACIAL TRIAD / ASPARAGINYL/ASPARTYL HYDROXYLASE / SIGNALING / ARD / BETA-HYDROXYLATION / ACTIVATOR-INHIBITOR / OXIDOREDUCTASE-PEPTIDE COMPLEX / OXIDOREDUCTASE ANKYRIN / CHANNEL / PROTEIN BINDING / ION CHANNEL / TRPA CHANNEL / MEMBRANE PROTEIN |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
regulation of blood circulation / positive regulation of monoatomic anion transport / cellular response to food / temperature-gated cation channel activity / regulation of neuronal action potential / hypoxia-inducible factor-asparagine dioxygenase / : / [protein]-asparagine 3-dioxygenase activity / cellular response to carbon dioxide / osmolarity-sensing monoatomic cation channel activity ...regulation of blood circulation / positive regulation of monoatomic anion transport / cellular response to food / temperature-gated cation channel activity / regulation of neuronal action potential / hypoxia-inducible factor-asparagine dioxygenase / : / [protein]-asparagine 3-dioxygenase activity / cellular response to carbon dioxide / osmolarity-sensing monoatomic cation channel activity / peptidyl-histidine dioxygenase activity / peptidyl-aspartic acid 3-dioxygenase activity / regulation of vascular endothelial growth factor receptor signaling pathway / Cellular response to hypoxia / stereocilium bundle / positive regulation of vasculogenesis / carboxylic acid binding / detection of chemical stimulus involved in sensory perception of pain / urinary bladder smooth muscle contraction / ankyrin repeat binding / thermoception / cellular response to toxic substance / Notch binding / oxygen sensor activity / cellular response to caffeine / response to pain / calcium ion transmembrane import into cytosol / TRP channels / cellular response to cold / intracellularly gated calcium channel activity / negative regulation of Notch signaling pathway / NF-kappaB binding / detection of mechanical stimulus involved in sensory perception of pain / positive regulation of myoblast differentiation / positive regulation of insulin secretion involved in cellular response to glucose stimulus / voltage-gated calcium channel activity / monoatomic ion transport / sensory perception of pain / response to cold / ferrous iron binding / calcium ion transmembrane transport / calcium channel activity / cellular response to hydrogen peroxide / intracellular calcium ion homeostasis / transcription corepressor activity / cellular response to heat / channel activity / protein homotetramerization / RNA polymerase II-specific DNA-binding transcription factor binding / cell surface receptor signaling pathway / apical plasma membrane / response to xenobiotic stimulus / axon / perinuclear region of cytoplasm / negative regulation of transcription by RNA polymerase II / protein homodimerization activity / zinc ion binding / nucleoplasm / identical protein binding / plasma membrane / cytoplasm / cytosol類似検索 - 分子機能 : / Clavaminate synthase-like / Hypoxia-inducible factor 1-alpha inhibitor, domain II / Cupin-like domain 8 / Cupin-like domain / A domain family that is part of the cupin metalloenzyme superfamily. / JmjC domain / JmjC domain profile. / Jelly Rolls / Ankyrin repeat ...: / Clavaminate synthase-like / Hypoxia-inducible factor 1-alpha inhibitor, domain II / Cupin-like domain 8 / Cupin-like domain / A domain family that is part of the cupin metalloenzyme superfamily. / JmjC domain / JmjC domain profile. / Jelly Rolls / Ankyrin repeat / RmlC-like jelly roll fold / Ankyrin repeats (3 copies) / Ankyrin repeat profile. / Ankyrin repeat region circular profile. / ankyrin repeats / Ankyrin repeat / Ankyrin repeat-containing domain superfamily / Ion transport domain / Ion transport protein / Helix Hairpins / Jelly Rolls / Sandwich / Orthogonal Bundle / Mainly Beta / Mainly Alpha類似検索 - ドメイン・相同性 N-OXALYLGLYCINE / Transient receptor potential cation channel subfamily A member 1 / Hypoxia-inducible factor 1-alpha inhibitor類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å |
|---|
 データ登録者 データ登録者 | Leissing, T.M. / Clifton, I.J. / Saward, B.G. / Lu, X. / Hopkinson, R.J. / Schofield, C.J. |
|---|
| 資金援助 |  英国, 1件 英国, 1件 | 組織 | 認可番号 | 国 |
|---|
| Engineering and Physical Sciences Research Council | P/G03706X/1 |  英国 英国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Factor Inhibiting HIF (FIH) in complex with zinc, NOG and TRPA1 (313-339)
著者: Leissing, T.M. / Clifton, I.J. / Saward, B.G. / Lu, X. / Hopkinson, R.J. / Schofield, C.J. |
|---|
| 履歴 | | 登録 | 2018年8月14日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2019年8月28日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年1月17日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 英国, 1件
英国, 1件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6hc8.cif.gz
6hc8.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6hc8.ent.gz
pdb6hc8.ent.gz PDB形式
PDB形式 6hc8.json.gz
6hc8.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/hc/6hc8
https://data.pdbj.org/pub/pdb/validation_reports/hc/6hc8 ftp://data.pdbj.org/pub/pdb/validation_reports/hc/6hc8
ftp://data.pdbj.org/pub/pdb/validation_reports/hc/6hc8
 リンク
リンク 集合体
集合体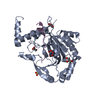

 要素
要素 Homo sapiens (ヒト) / 遺伝子: HIF1AN, FIH1 / プラスミド: pET28 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: HIF1AN, FIH1 / プラスミド: pET28 / 発現宿主: 
 Homo sapiens (ヒト) / 参照: UniProt: O75762
Homo sapiens (ヒト) / 参照: UniProt: O75762







 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I03 / 波長: 0.9795 Å
/ ビームライン: I03 / 波長: 0.9795 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj







