+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 6h4p | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal structure of human KDM4A in complex with compound 16a | ||||||
 Components Components | Lysine-specific demethylase 4A | ||||||
 Keywords Keywords | OXIDOREDUCTASE / Histone demethylase / Inhibitor / transcription | ||||||
| Function / homology |  Function and homology information Function and homology information[histone H3]-trimethyl-L-lysine36 demethylase / histone H3K36me2/H3K36me3 demethylase activity / histone H4K20me2 reader activity / histone H3K36 demethylase activity / cardiac muscle hypertrophy in response to stress / [histone H3]-trimethyl-L-lysine9 demethylase / histone H3K9me2/H3K9me3 demethylase activity / histone H3K9 demethylase activity / histone demethylase activity / pericentric heterochromatin ...[histone H3]-trimethyl-L-lysine36 demethylase / histone H3K36me2/H3K36me3 demethylase activity / histone H4K20me2 reader activity / histone H3K36 demethylase activity / cardiac muscle hypertrophy in response to stress / [histone H3]-trimethyl-L-lysine9 demethylase / histone H3K9me2/H3K9me3 demethylase activity / histone H3K9 demethylase activity / histone demethylase activity / pericentric heterochromatin / NR1H3 & NR1H2 regulate gene expression linked to cholesterol transport and efflux / negative regulation of autophagy / HDMs demethylate histones / fibrillar center / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / regulation of gene expression / chromatin remodeling / negative regulation of gene expression / negative regulation of DNA-templated transcription / ubiquitin protein ligase binding / chromatin / zinc ion binding / nucleoplasm / nucleus / cytosol Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.19 Å MOLECULAR REPLACEMENT / Resolution: 2.19 Å | ||||||
 Authors Authors | Le Bihan, Y.V. / van Montfort, R.L.M. | ||||||
| Funding support |  United Kingdom, 1items United Kingdom, 1items
| ||||||
 Citation Citation |  Journal: Eur.J.Med.Chem. / Year: 2019 Journal: Eur.J.Med.Chem. / Year: 2019Title: C8-substituted pyrido[3,4-d]pyrimidin-4(3H)-ones: Studies towards the identification of potent, cell penetrant Jumonji C domain containing histone lysine demethylase 4 subfamily (KDM4) ...Title: C8-substituted pyrido[3,4-d]pyrimidin-4(3H)-ones: Studies towards the identification of potent, cell penetrant Jumonji C domain containing histone lysine demethylase 4 subfamily (KDM4) inhibitors, compound profiling in cell-based target engagement assays. Authors: Le Bihan, Y.V. / Lanigan, R.M. / Atrash, B. / McLaughlin, M.G. / Velupillai, S. / Malcolm, A.G. / England, K.S. / Ruda, G.F. / Mok, N.Y. / Tumber, A. / Tomlin, K. / Saville, H. / Shehu, E. / ...Authors: Le Bihan, Y.V. / Lanigan, R.M. / Atrash, B. / McLaughlin, M.G. / Velupillai, S. / Malcolm, A.G. / England, K.S. / Ruda, G.F. / Mok, N.Y. / Tumber, A. / Tomlin, K. / Saville, H. / Shehu, E. / McAndrew, C. / Carmichael, L. / Bennett, J.M. / Jeganathan, F. / Eve, P. / Donovan, A. / Hayes, A. / Wood, F. / Raynaud, F.I. / Fedorov, O. / Brennan, P.E. / Burke, R. / van Montfort, R.L.M. / Rossanese, O.W. / Blagg, J. / Bavetsias, V. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  6h4p.cif.gz 6h4p.cif.gz | 576.9 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb6h4p.ent.gz pdb6h4p.ent.gz | 475.9 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  6h4p.json.gz 6h4p.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/h4/6h4p https://data.pdbj.org/pub/pdb/validation_reports/h4/6h4p ftp://data.pdbj.org/pub/pdb/validation_reports/h4/6h4p ftp://data.pdbj.org/pub/pdb/validation_reports/h4/6h4p | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  6h4oC  6h4qC  6h4rC  6h4sC  6h4tC  6h4uC  6h4vC  6h4wC  6h4xC  6h4yC  6h4zC  6h50C  6h51C  6h52C  2oq7S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 4 molecules ABCD
| #1: Protein | Mass: 41854.617 Da / Num. of mol.: 4 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: KDM4A, JHDM3A, JMJD2, JMJD2A, KIAA0677 / Production host: Homo sapiens (human) / Gene: KDM4A, JHDM3A, JMJD2, JMJD2A, KIAA0677 / Production host:  References: UniProt: O75164, Oxidoreductases; Acting on paired donors, with incorporation or reduction of molecular oxygen; With 2-oxoglutarate as one donor, and incorporation of one atom of oxygen into each donor |
|---|
-Non-polymers , 6 types, 673 molecules 
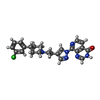









| #2: Chemical | ChemComp-ZN / #3: Chemical | ChemComp-FQ5 / #4: Chemical | ChemComp-DMS / #5: Chemical | ChemComp-CL / #6: Chemical | #7: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.49 Å3/Da / Density % sol: 50.67 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion, hanging drop / pH: 7.5 Details: Crystallisation solution is 0.1M Bis-Tris-Propane pH7.5, 12-16% PEG-4000. Inhibitor is soaked in crystals by addition directly to the drops of DMSO dissolved compound |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  Diamond Diamond  / Beamline: I03 / Wavelength: 0.9763 Å / Beamline: I03 / Wavelength: 0.9763 Å |
| Detector | Type: DECTRIS PILATUS3 6M / Detector: PIXEL / Date: Feb 20, 2016 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.9763 Å / Relative weight: 1 |
| Reflection | Resolution: 2.19→82.58 Å / Num. obs: 84505 / % possible obs: 99.9 % / Redundancy: 6.7 % / Biso Wilson estimate: 55.38 Å2 / CC1/2: 1 / Rmerge(I) obs: 0.119 / Net I/σ(I): 9.2 |
| Reflection shell | Resolution: 2.19→2.24 Å / Redundancy: 6.6 % / Rmerge(I) obs: 1.462 / Mean I/σ(I) obs: 1.3 / Num. unique obs: 6259 / CC1/2: 0.5 / % possible all: 100 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 2OQ7 Resolution: 2.19→82.57 Å / Cor.coef. Fo:Fc: 0.957 / Cor.coef. Fo:Fc free: 0.95 / SU R Cruickshank DPI: 0.206 / Cross valid method: THROUGHOUT / σ(F): 0 / SU R Blow DPI: 0.205 / SU Rfree Blow DPI: 0.166 / SU Rfree Cruickshank DPI: 0.168
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 61.35 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.25 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: 1 / Resolution: 2.19→82.57 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 2.19→2.25 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller













 PDBj
PDBj








