+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6ao5 | ||||||
|---|---|---|---|---|---|---|---|
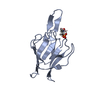
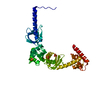
| タイトル | Crystal structure of human MST2 in complex with SAV1 SARAH domain | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | SIGNALING PROTEIN / Hippo / mst autoactivation / dimerization | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報keratinocyte apoptotic process / regulation of cell differentiation involved in embryonic placenta development / cell differentiation involved in embryonic placenta development / primitive hemopoiesis / neural tube formation / intestinal epithelial cell differentiation / positive regulation of extrinsic apoptotic signaling pathway via death domain receptors / regulation of stem cell population maintenance / positive regulation of keratinocyte apoptotic process / positive regulation of hippo signaling ...keratinocyte apoptotic process / regulation of cell differentiation involved in embryonic placenta development / cell differentiation involved in embryonic placenta development / primitive hemopoiesis / neural tube formation / intestinal epithelial cell differentiation / positive regulation of extrinsic apoptotic signaling pathway via death domain receptors / regulation of stem cell population maintenance / positive regulation of keratinocyte apoptotic process / positive regulation of hippo signaling / endocardium development / negative regulation of cardiac muscle cell proliferation / negative regulation of organ growth / lung epithelial cell differentiation / hippo signaling / transcription regulator activator activity / Signaling by Hippo / protein localization to centrosome / organ growth / ventricular septum morphogenesis / hepatocyte apoptotic process / regulation of MAPK cascade / extrinsic apoptotic signaling pathway via death domain receptors / hair follicle development / positive regulation of fat cell differentiation / cardiac muscle cell proliferation / canonical Wnt signaling pathway / keratinocyte differentiation / JNK cascade / protein serine/threonine kinase activator activity / central nervous system development / epithelial cell proliferation / protein tetramerization / positive regulation of JNK cascade / phosphatidylinositol 3-kinase/protein kinase B signal transduction / negative regulation of canonical Wnt signaling pathway / protein import into nucleus / negative regulation of epithelial cell proliferation / molecular adaptor activity / protein phosphorylation / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / non-specific serine/threonine protein kinase / protein kinase activity / protein stabilization / intracellular signal transduction / positive regulation of apoptotic process / negative regulation of cell population proliferation / protein serine kinase activity / protein serine/threonine kinase activity / apoptotic process / centrosome / magnesium ion binding / protein-containing complex / ATP binding / identical protein binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.955 Å 分子置換 / 解像度: 2.955 Å | ||||||
 データ登録者 データ登録者 | Tomchick, D.R. / Luo, X. / Ni, L. | ||||||
 引用 引用 |  ジャーナル: Elife / 年: 2017 ジャーナル: Elife / 年: 2017タイトル: SAV1 promotes Hippo kinase activation through antagonizing the PP2A phosphatase STRIPAK. 著者: Bae, S.J. / Ni, L. / Osinski, A. / Tomchick, D.R. / Brautigam, C.A. / Luo, X. #1:  ジャーナル: Structure / 年: 2013 ジャーナル: Structure / 年: 2013タイトル: Structural basis for autoactivation of human Mst2 kinase and its regulation by RASSF5. 著者: Ni, L. / Li, S. / Yu, J. / Min, J. / Brautigam, C.A. / Tomchick, D.R. / Pan, D. / Luo, X. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6ao5.cif.gz 6ao5.cif.gz | 256.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6ao5.ent.gz pdb6ao5.ent.gz | 211.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6ao5.json.gz 6ao5.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6ao5_validation.pdf.gz 6ao5_validation.pdf.gz | 797.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6ao5_full_validation.pdf.gz 6ao5_full_validation.pdf.gz | 801.9 KB | 表示 | |
| XML形式データ |  6ao5_validation.xml.gz 6ao5_validation.xml.gz | 16.1 KB | 表示 | |
| CIF形式データ |  6ao5_validation.cif.gz 6ao5_validation.cif.gz | 21.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ao/6ao5 https://data.pdbj.org/pub/pdb/validation_reports/ao/6ao5 ftp://data.pdbj.org/pub/pdb/validation_reports/ao/6ao5 ftp://data.pdbj.org/pub/pdb/validation_reports/ao/6ao5 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 41733.961 Da / 分子数: 1 / 断片: kinase domain (UNP residues 16-313, 428-491) / 変異: D146N / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: STK3, KRS1, MST2 / プラスミド: PET29 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: STK3, KRS1, MST2 / プラスミド: PET29 / 発現宿主:  参照: UniProt: Q13188, non-specific serine/threonine protein kinase |
|---|---|
| #2: タンパク質 | 分子量: 11270.966 Da / 分子数: 1 / 断片: SARAH domain (UNP residues 291-383) / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: SAV1, WW45 / プラスミド: PET29 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: SAV1, WW45 / プラスミド: PET29 / 発現宿主:  |
| #3: 化合物 | ChemComp-ANP / |
| #4: 化合物 | ChemComp-MG / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.62 Å3/Da / 溶媒含有率: 65.99 % / Mosaicity: 0.579 ° |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 詳細: 0.05 M NaCl, 0.1 M Hepes, 0.19 mM CYMAL-7, 1 mM TCEP, 40% PEG 400 |
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 19-ID / 波長: 0.97918 Å / ビームライン: 19-ID / 波長: 0.97918 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: ADSC QUANTUM 315r / 検出器: CCD / 日付: 2015年10月12日 / 詳細: monochromator | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長: 0.97918 Å / 相対比: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2.95→50 Å / Num. obs: 15976 / % possible obs: 99.9 % / 冗長度: 19 % / Biso Wilson estimate: 48.45 Å2 / Rmerge(I) obs: 0.124 / Rpim(I) all: 0.029 / Rrim(I) all: 0.128 / Χ2: 1.085 / Net I/σ(I): 7 / Num. measured all: 303392 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 4LGD 解像度: 2.955→42.271 Å / SU ML: 0.32 / 交差検証法: FREE R-VALUE / σ(F): 1.36 / 位相誤差: 28.54 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 192.97 Å2 / Biso mean: 60.0404 Å2 / Biso min: 15.87 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.955→42.271 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0 / Total num. of bins used: 5
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj





















