+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5ykv | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | The crystal structure of Macrobrachium rosenbergii nodavirus P-domain | ||||||
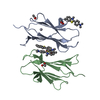
 要素 要素 | Capsid protein | ||||||
 キーワード キーワード | VIRAL PROTEIN / viral capsid protein | ||||||
| 機能・相同性 | virion component / Viral coat protein subunit / metal ion binding / Capsid protein alpha 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Macrobrachium rosenbergii nodavirus (ウイルス) Macrobrachium rosenbergii nodavirus (ウイルス) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2.31 Å シンクロトロン / 解像度: 2.31 Å | ||||||
 データ登録者 データ登録者 | Chen, N.C. / Yoshimura, M. / Lin, C.C. / Guan, H.H. / Chuankhayan, P. / Chen, C.J. | ||||||
| 資金援助 |  台湾, 1件 台湾, 1件
| ||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2019 ジャーナル: Commun Biol / 年: 2019タイトル: The atomic structures of shrimp nodaviruses reveal new dimeric spike structures and particle polymorphism. 著者: Nai-Chi Chen / Masato Yoshimura / Naoyuki Miyazaki / Hong-Hsiang Guan / Phimonphan Chuankhayan / Chien-Chih Lin / Shao-Kang Chen / Pei-Ju Lin / Yen-Chieh Huang / Kenji Iwasaki / Atsushi ...著者: Nai-Chi Chen / Masato Yoshimura / Naoyuki Miyazaki / Hong-Hsiang Guan / Phimonphan Chuankhayan / Chien-Chih Lin / Shao-Kang Chen / Pei-Ju Lin / Yen-Chieh Huang / Kenji Iwasaki / Atsushi Nakagawa / Sunney I Chan / Chun-Jung Chen /    要旨: Shrimp nodaviruses, including (PvNV) and nodaviruses (MrNV), cause white-tail disease in shrimps, with high mortality. The viral capsid structure determines viral assembly and host specificity ...Shrimp nodaviruses, including (PvNV) and nodaviruses (MrNV), cause white-tail disease in shrimps, with high mortality. The viral capsid structure determines viral assembly and host specificity during infections. Here, we show cryo-EM structures of = 3 and = 1 PvNV-like particles (PvNV-LPs), crystal structures of the protrusion-domains (P-domains) of PvNV and MrNV, and the crystal structure of the ∆N-ARM-PvNV shell-domain (S-domain) in = 1 subviral particles. The capsid protein of PvNV reveals five domains: the P-domain with a new jelly-roll structure forming cuboid-like spikes; the jelly-roll S-domain with two calcium ions; the linker between the S- and P-domains exhibiting new cross and parallel conformations; the N-arm interacting with nucleotides organized along icosahedral two-fold axes; and a disordered region comprising the basic -terminal arginine-rich motif (N-ARM) interacting with RNA. The N-ARM controls = 3 and = 1 assemblies. Increasing the /-termini flexibility leads to particle polymorphism. Linker flexibility may influence the dimeric-spike arrangement. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5ykv.cif.gz 5ykv.cif.gz | 214.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5ykv.ent.gz pdb5ykv.ent.gz | 172.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5ykv.json.gz 5ykv.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5ykv_validation.pdf.gz 5ykv_validation.pdf.gz | 472.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5ykv_full_validation.pdf.gz 5ykv_full_validation.pdf.gz | 477.6 KB | 表示 | |
| XML形式データ |  5ykv_validation.xml.gz 5ykv_validation.xml.gz | 42.7 KB | 表示 | |
| CIF形式データ |  5ykv_validation.cif.gz 5ykv_validation.cif.gz | 62.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/yk/5ykv https://data.pdbj.org/pub/pdb/validation_reports/yk/5ykv ftp://data.pdbj.org/pub/pdb/validation_reports/yk/5ykv ftp://data.pdbj.org/pub/pdb/validation_reports/yk/5ykv | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 13833.484 Da / 分子数: 8 / 断片: UNP residues 247-371 / 由来タイプ: 組換発現 由来: (組換発現)  Macrobrachium rosenbergii nodavirus (ウイルス) Macrobrachium rosenbergii nodavirus (ウイルス)発現宿主:  #2: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.71 Å3/Da / 溶媒含有率: 66.82 % |
|---|---|
| 結晶化 | 温度: 291 K / 手法: 蒸気拡散法, ハンギングドロップ法 詳細: 0.1 M MES monohydrate pH 6.5, 1.6 M Magnesium sulfate heptahydrate |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SPring-8 SPring-8  / ビームライン: BL44XU / 波長: 0.9 Å / ビームライン: BL44XU / 波長: 0.9 Å |
| 検出器 | タイプ: RAYONIX MX300HE / 検出器: CCD / 日付: 2015年6月25日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9 Å / 相対比: 1 |
| 反射 | 解像度: 2.3→30 Å / Num. obs: 71611 / % possible obs: 99.3 % / 冗長度: 5.3 % / Rpim(I) all: 0.067 / Rrim(I) all: 0.157 / Rsym value: 0.142 / Net I/σ(I): 11.2 |
| 反射 シェル | 解像度: 2.3→2.38 Å / 冗長度: 4.9 % / CC1/2: 0.793 / Rpim(I) all: 0.304 / Rrim(I) all: 0.702 / Rsym value: 0.63 / % possible all: 97.6 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2.31→29.99 Å / Cor.coef. Fo:Fc: 0.965 / Cor.coef. Fo:Fc free: 0.944 / SU B: 5.217 / SU ML: 0.122 / 交差検証法: THROUGHOUT / ESU R: 0.191 / ESU R Free: 0.168 / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 34.033 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 2.31→29.99 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー





















 PDBj
PDBj