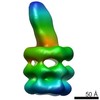
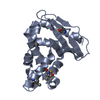
登録情報 データベース : PDB / ID : 5uwbタイトル Re-refined 4FCZ: lipid-bound crystal structure of toluene-tolerance protein from Pseudomonas putida Toluene tolerance protein キーワード / / / / 機能・相同性 / / / / / / / / 生物種 Pseudomonas putida (バクテリア)手法 / / 解像度 : 2.604 Å データ登録者 Bhabha, G. / Ekiert, D.C. 資金援助 組織 認可番号 国 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) K99GM112982 Damon Runyon Cancer Research Foundation DRG-2140-12 Howard Hughes Medical Institute (HHMI)
ジャーナル : To be Published タイトル : Northeast Structural Genomics Consortium Target PpR99著者 : Kuzin, A. / Su, M. / Seetharaman, J. / Sahdev, S. / Xiao, R. / Ciccosanti, C. / Wang, H. / Everett, J.K. / Acton, T.B. / Montelione, G.T. / Tong, L. / Hunt, J.F. 履歴 登録 2017年2月20日 登録サイト / 処理サイト 改定 1.0 2017年4月19日 Provider / タイプ 改定 1.1 2017年6月14日 Group / カテゴリ / Item 改定 1.2 2017年9月13日 Group / カテゴリ / Item 改定 1.3 2019年11月20日 Group / カテゴリ / Item 改定 1.4 2024年10月23日 Group / Database references / Structure summaryカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature Item / _database_2.pdbx_database_accession
すべて表示 表示を減らす Remark 0 THIS ENTRY 5UWB REFLECTS AN ALTERNATIVE MODELING OF THE ORIGINAL STRUCTURAL DATA R4FCZSF DETERMINED ... THIS ENTRY 5UWB REFLECTS AN ALTERNATIVE MODELING OF THE ORIGINAL STRUCTURAL DATA R4FCZSF DETERMINED BY AUTHORS OF THE PDB ENTRY 4FCZ: AUTHORS A.KUZIN, M.SU, J.SEETHARAMAN, S.SAHDEV, R.XIAO, C.CICCOSANTI, H.WANG, J.K.EVERETT, T.B.ACTON, G.T.MONTELIONE, L.TONG, J.F.HUNT, NORTHEAST STRUCTURAL GENOMICS CONSORTIUM (NESG) Remark 200 AUTHOR USED THE SF DATA FROM ENTRY 4FCZ.
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Pseudomonas putida (バクテリア)
Pseudomonas putida (バクテリア) X線回折 /
X線回折 /  単波長異常分散 / 解像度: 2.604 Å
単波長異常分散 / 解像度: 2.604 Å  データ登録者
データ登録者 米国, 3件
米国, 3件  引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5uwb.cif.gz
5uwb.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5uwb.ent.gz
pdb5uwb.ent.gz PDB形式
PDB形式 5uwb.json.gz
5uwb.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5uwb_validation.pdf.gz
5uwb_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5uwb_full_validation.pdf.gz
5uwb_full_validation.pdf.gz 5uwb_validation.xml.gz
5uwb_validation.xml.gz 5uwb_validation.cif.gz
5uwb_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/uw/5uwb
https://data.pdbj.org/pub/pdb/validation_reports/uw/5uwb ftp://data.pdbj.org/pub/pdb/validation_reports/uw/5uwb
ftp://data.pdbj.org/pub/pdb/validation_reports/uw/5uwb リンク
リンク 集合体
集合体


 要素
要素 Pseudomonas putida (strain ATCC 47054 / DSM 6125 / NCIMB 11950 / KT2440) (バクテリア)
Pseudomonas putida (strain ATCC 47054 / DSM 6125 / NCIMB 11950 / KT2440) (バクテリア)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 解析
解析 単波長異常分散 / 解像度: 2.604→39.086 Å / SU ML: 0.39 / 交差検証法: FREE R-VALUE / σ(F): 0.12 / 位相誤差: 29.65
単波長異常分散 / 解像度: 2.604→39.086 Å / SU ML: 0.39 / 交差検証法: FREE R-VALUE / σ(F): 0.12 / 位相誤差: 29.65  ムービー
ムービー コントローラー
コントローラー




















 PDBj
PDBj

