| 登録情報 | データベース: PDB / ID: 5tcd
|
|---|
| タイトル | Human alkaline sphingomyelinase (ENPP7) in complex with phosphocholine |
|---|
 要素 要素 | Ectonucleotide pyrophosphatase/phosphodiesterase family member 7 |
|---|
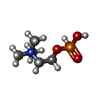
 キーワード キーワード | HYDROLASE / sphingomyelinase / sphingomyelin / phosphocholine / glycosylphosphatidylinositol |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
positive regulation of intestinal cholesterol absorption / regulation of intestinal lipid absorption / sphingomyelin metabolic process / positive regulation of sphingomyelin catabolic process / lipid digestion / sphingomyelin phosphodiesterase / sphingomyelin phosphodiesterase activity / positive regulation of ceramide biosynthetic process / glycosphingolipid catabolic process / Glycosphingolipid catabolism ...positive regulation of intestinal cholesterol absorption / regulation of intestinal lipid absorption / sphingomyelin metabolic process / positive regulation of sphingomyelin catabolic process / lipid digestion / sphingomyelin phosphodiesterase / sphingomyelin phosphodiesterase activity / positive regulation of ceramide biosynthetic process / glycosphingolipid catabolic process / Glycosphingolipid catabolism / phosphoric diester hydrolase activity / negative regulation of DNA replication / microvillus / fatty acid homeostasis / negative regulation of cell population proliferation / Golgi apparatus / zinc ion binding / membrane / plasma membrane類似検索 - 分子機能 Alkaline Phosphatase, subunit A / Alkaline Phosphatase, subunit A / Type I phosphodiesterase/nucleotide pyrophosphatase/phosphate transferase / Type I phosphodiesterase / nucleotide pyrophosphatase / Alkaline-phosphatase-like, core domain superfamily / 3-Layer(aba) Sandwich / Alpha Beta類似検索 - ドメイン・相同性 IODIDE ION / PHOSPHOCHOLINE / Ectonucleotide pyrophosphatase/phosphodiesterase family member 7類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 2.4 Å 単波長異常分散 / 解像度: 2.4 Å |
|---|
 データ登録者 データ登録者 | Gorelik, A. / Liu, F. / Illes, K. / Nagar, B. |
|---|
| 資金援助 |  カナダ, 1件 カナダ, 1件 | 組織 | 認可番号 | 国 |
|---|
| Canadian Institutes of Health Research (CIHR) | MOP-133535 |  カナダ カナダ |
|
|---|
 引用 引用 |  ジャーナル: J. Biol. Chem. / 年: 2017 ジャーナル: J. Biol. Chem. / 年: 2017
タイトル: Crystal structure of the human alkaline sphingomyelinase provides insights into substrate recognition.
著者: Gorelik, A. / Liu, F. / Illes, K. / Nagar, B. |
|---|
| 履歴 | | 登録 | 2016年9月14日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2017年3月22日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年5月10日 | Group: Database references |
|---|
| 改定 1.2 | 2020年1月8日 | Group: Author supporting evidence / Data collection / カテゴリ: chem_comp / pdbx_audit_support
Item: _chem_comp.type / _pdbx_audit_support.funding_organization |
|---|
| 改定 2.0 | 2020年7月29日 | Group: Atomic model / Data collection ...Atomic model / Data collection / Derived calculations / Structure summary
カテゴリ: atom_site / atom_site_anisotrop ...atom_site / atom_site_anisotrop / chem_comp / entity / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / pdbx_struct_conn_angle / struct_asym / struct_conn / struct_conn_type / struct_site / struct_site_gen
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.type_symbol / _atom_site_anisotrop.id / _atom_site_anisotrop.pdbx_label_asym_id / _chem_comp.name / _chem_comp.type / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_role / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn_type.id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 2.1 | 2024年11月6日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / struct_conn
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI ..._chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_leaving_atom_flag |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  単波長異常分散 / 解像度: 2.4 Å
単波長異常分散 / 解像度: 2.4 Å  データ登録者
データ登録者 カナダ, 1件
カナダ, 1件  引用
引用 ジャーナル: J. Biol. Chem. / 年: 2017
ジャーナル: J. Biol. Chem. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5tcd.cif.gz
5tcd.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5tcd.ent.gz
pdb5tcd.ent.gz PDB形式
PDB形式 5tcd.json.gz
5tcd.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5tcd_validation.pdf.gz
5tcd_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5tcd_full_validation.pdf.gz
5tcd_full_validation.pdf.gz 5tcd_validation.xml.gz
5tcd_validation.xml.gz 5tcd_validation.cif.gz
5tcd_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/tc/5tcd
https://data.pdbj.org/pub/pdb/validation_reports/tc/5tcd ftp://data.pdbj.org/pub/pdb/validation_reports/tc/5tcd
ftp://data.pdbj.org/pub/pdb/validation_reports/tc/5tcd リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: ENPP7, UNQ3077/PRO9912
Homo sapiens (ヒト) / 遺伝子: ENPP7, UNQ3077/PRO9912









 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  CLSI
CLSI  / ビームライン: 08ID-1 / 波長: 0.97949 Å
/ ビームライン: 08ID-1 / 波長: 0.97949 Å 解析
解析 単波長異常分散 / 解像度: 2.4→38.444 Å / SU ML: 0.22 / 交差検証法: FREE R-VALUE / σ(F): 1.35 / 位相誤差: 22.22 / 立体化学のターゲット値: ML
単波長異常分散 / 解像度: 2.4→38.444 Å / SU ML: 0.22 / 交差検証法: FREE R-VALUE / σ(F): 1.35 / 位相誤差: 22.22 / 立体化学のターゲット値: ML ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj





