| 登録情報 | データベース: PDB / ID: 5t5w
|
|---|
| タイトル | Structure of an affinity matured lambda-IFN/IFN-lambdaR1/IL-10Rbeta receptor complex |
|---|
 要素 要素 | - Interferon lambda receptor 1
- Interferon lambda-3
- Interleukin-10 receptor subunit beta
|
|---|
 キーワード キーワード | CYTOKINE/CYTOKINE RECEPTOR / receptor / cytokine / CYTOKINE-CYTOKINE RECEPTOR complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
interleukin-10 receptor activity / response to type III interferon / interleukin-28 receptor complex / mucosal immune response / positive regulation of cellular respiration / type III interferon-mediated signaling pathway / interleukin-10-mediated signaling pathway / regulation of defense response to virus by host / cytokine receptor activity / Other interleukin signaling ...interleukin-10 receptor activity / response to type III interferon / interleukin-28 receptor complex / mucosal immune response / positive regulation of cellular respiration / type III interferon-mediated signaling pathway / interleukin-10-mediated signaling pathway / regulation of defense response to virus by host / cytokine receptor activity / Other interleukin signaling / Interleukin-20 family signaling / negative regulation of viral genome replication / Interleukin-10 signaling / cell surface receptor signaling pathway via JAK-STAT / coreceptor activity / cytokine activity / positive regulation of receptor signaling pathway via JAK-STAT / positive regulation of immune response / cellular response to virus / cytokine-mediated signaling pathway / signaling receptor activity / defense response to virus / immune response / inflammatory response / signaling receptor binding / negative regulation of cell population proliferation / innate immune response / signal transduction / extracellular space / extracellular region / membrane / plasma membrane類似検索 - 分子機能 Interferon lambda / Interferon lambda superfamily / Interleukin-28A / Interferon/interleukin receptor domain / Interferon-alpha/beta receptor, fibronectin type III / : / Tissue factor / Fibronectin type-III domain profile. / Fibronectin type III / Fibronectin type III superfamily ...Interferon lambda / Interferon lambda superfamily / Interleukin-28A / Interferon/interleukin receptor domain / Interferon-alpha/beta receptor, fibronectin type III / : / Tissue factor / Fibronectin type-III domain profile. / Fibronectin type III / Fibronectin type III superfamily / Immunoglobulin-like fold / Immunoglobulins / Immunoglobulin-like / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 Interleukin-10 receptor subunit beta / Interferon lambda receptor 1 / Interferon lambda-3類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.847 Å 分子置換 / 解像度: 2.847 Å |
|---|
 データ登録者 データ登録者 | Mendoza, J.L. / Jude, K.M. / Garcia, K.C. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute Of Allergy and Infectious Diseases (NIH/NIAID) | 1U19AI109662 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Immunity / 年: 2017 ジャーナル: Immunity / 年: 2017
タイトル: The IFN-lambda-IFN-lambda R1-IL-10R beta Complex Reveals Structural Features Underlying Type III IFN Functional Plasticity.
著者: Mendoza, J.L. / Schneider, W.M. / Hoffmann, H.H. / Vercauteren, K. / Jude, K.M. / Xiong, A. / Moraga, I. / Horton, T.M. / Glenn, J.S. / de Jong, Y.P. / Rice, C.M. / Garcia, K.C. |
|---|
| 履歴 | | 登録 | 2016年8月31日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2017年3月29日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年4月5日 | Group: Database references |
|---|
| 改定 1.2 | 2017年8月9日 | Group: Database references / カテゴリ: pdbx_related_exp_data_set / Item: _pdbx_related_exp_data_set.data_reference |
|---|
| 改定 1.3 | 2017年9月13日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.4 | 2018年8月15日 | Group: Data collection / Database references / カテゴリ: pdbx_related_exp_data_set / Item: _pdbx_related_exp_data_set.data_reference |
|---|
| 改定 1.5 | 2019年12月11日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.6 | 2020年7月29日 | Group: Data collection / Derived calculations / Structure summary
カテゴリ: chem_comp / entity ...chem_comp / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / struct_conn / struct_site / struct_site_gen
Item: _chem_comp.name / _chem_comp.type ..._chem_comp.name / _chem_comp.type / _entity.pdbx_description / _pdbx_entity_nonpoly.name / _struct_conn.pdbx_role
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 1.7 | 2021年3月24日 | Group: Source and taxonomy / Structure summary / カテゴリ: chem_comp / entity_src_gen
Item: _chem_comp.pdbx_synonyms / _entity_src_gen.pdbx_host_org_cell_line / _entity_src_gen.pdbx_host_org_strain |
|---|
| 改定 1.8 | 2023年10月4日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
| 改定 1.9 | 2024年10月23日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.847 Å
分子置換 / 解像度: 2.847 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Immunity / 年: 2017
ジャーナル: Immunity / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5t5w.cif.gz
5t5w.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5t5w.ent.gz
pdb5t5w.ent.gz PDB形式
PDB形式 5t5w.json.gz
5t5w.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5t5w_validation.pdf.gz
5t5w_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5t5w_full_validation.pdf.gz
5t5w_full_validation.pdf.gz 5t5w_validation.xml.gz
5t5w_validation.xml.gz 5t5w_validation.cif.gz
5t5w_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/t5/5t5w
https://data.pdbj.org/pub/pdb/validation_reports/t5/5t5w ftp://data.pdbj.org/pub/pdb/validation_reports/t5/5t5w
ftp://data.pdbj.org/pub/pdb/validation_reports/t5/5t5w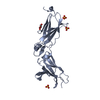

 10.15785/SBGRID/416 / データの種類: diffraction image data
10.15785/SBGRID/416 / データの種類: diffraction image data リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: IL10RB, CRFB4, D21S58, D21S66 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: IL10RB, CRFB4, D21S58, D21S66 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q08334
Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q08334 Homo sapiens (ヒト) / 遺伝子: IFNLR1, IL28RA, LICR2 / 細胞株 (発現宿主): HEK 293 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: IFNLR1, IL28RA, LICR2 / 細胞株 (発現宿主): HEK 293 / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: Q8IU57
Homo sapiens (ヒト) / 参照: UniProt: Q8IU57 Homo sapiens (ヒト) / 遺伝子: IFNL3, IL28B, IL28C, ZCYTO22 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: IFNL3, IL28B, IL28C, ZCYTO22 / 発現宿主:  Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q8IZI9
Trichoplusia ni (イラクサキンウワバ) / 参照: UniProt: Q8IZI9 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 8.2.1 / 波長: 0.999975 Å
/ ビームライン: 8.2.1 / 波長: 0.999975 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj










