| 登録情報 | データベース: PDB / ID: 5t00
|
|---|
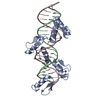
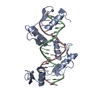
| タイトル | Human CTCF ZnF3-7 and methylated DNA complex |
|---|
 要素 要素 | - DNA (5'-GCCAGCAGGGGG(5CM)GCTA-3')
- DNA (5'-TAG(5CM)GCCCCCTGCTGGC-3')
- Transcriptional repressor CTCF
|
|---|
 キーワード キーワード | TRANSCRIPTION REGULATOR/DNA / CCCTC-binding factor / zinc finger / CTCF / TRANSCRIPTION REGULATOR-DNA complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
chromatin loop anchoring activity / chromatin insulator sequence binding / regulation of centromeric sister chromatid cohesion / protein localization to chromosome, centromeric region / genomic imprinting / chromatin looping / cardiac muscle cell development / DNA methylation-dependent constitutive heterochromatin formation / negative regulation of gene expression via chromosomal CpG island methylation / chromosome, centromeric region ...chromatin loop anchoring activity / chromatin insulator sequence binding / regulation of centromeric sister chromatid cohesion / protein localization to chromosome, centromeric region / genomic imprinting / chromatin looping / cardiac muscle cell development / DNA methylation-dependent constitutive heterochromatin formation / negative regulation of gene expression via chromosomal CpG island methylation / chromosome, centromeric region / condensed chromosome / epigenetic regulation of gene expression / transcription coregulator binding / male germ cell nucleus / mitochondrion organization / chromosome segregation / DNA-binding transcription repressor activity, RNA polymerase II-specific / Activation of anterior HOX genes in hindbrain development during early embryogenesis / sequence-specific double-stranded DNA binding / gene expression / sequence-specific DNA binding / in utero embryonic development / transcription cis-regulatory region binding / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / negative regulation of cell population proliferation / negative regulation of DNA-templated transcription / positive regulation of gene expression / regulation of transcription by RNA polymerase II / positive regulation of DNA-templated transcription / nucleolus / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / zinc ion binding / nucleoplasm / nucleus類似検索 - 分子機能 : / : / C2H2 zinc finger / C2H2-type zinc-finger domain / Zinc finger, C2H2 type / zinc finger / Zinc finger C2H2 type domain profile. / Zinc finger C2H2 superfamily / Zinc finger C2H2 type domain signature. / Zinc finger C2H2-type類似検索 - ドメイン・相同性 DNA / DNA (> 10) / Transcriptional repressor CTCF類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)
synthetic construct (人工物) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.19 Å 分子置換 / 解像度: 2.19 Å |
|---|
 データ登録者 データ登録者 | Hashimoto, H. / Wang, D. / Cheng, X. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | GM049245 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Mol. Cell / 年: 2017 ジャーナル: Mol. Cell / 年: 2017
タイトル: Structural Basis for the Versatile and Methylation-Dependent Binding of CTCF to DNA.
著者: Hashimoto, H. / Wang, D. / Horton, J.R. / Zhang, X. / Corces, V.G. / Cheng, X. |
|---|
| 履歴 | | 登録 | 2016年8月15日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2017年5月24日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年5月31日 | Group: Database references |
|---|
| 改定 1.2 | 2017年6月14日 | Group: Database references / カテゴリ: citation
Item: _citation.country / _citation.journal_id_ASTM ..._citation.country / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.3 | 2017年9月27日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.4 | 2019年12月25日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.5 | 2023年10月4日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_conn / struct_conn_type
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_conn_type.id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.19 Å
分子置換 / 解像度: 2.19 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Mol. Cell / 年: 2017
ジャーナル: Mol. Cell / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5t00.cif.gz
5t00.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5t00.ent.gz
pdb5t00.ent.gz PDB形式
PDB形式 5t00.json.gz
5t00.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5t00_validation.pdf.gz
5t00_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5t00_full_validation.pdf.gz
5t00_full_validation.pdf.gz 5t00_validation.xml.gz
5t00_validation.xml.gz 5t00_validation.cif.gz
5t00_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/t0/5t00
https://data.pdbj.org/pub/pdb/validation_reports/t0/5t00 ftp://data.pdbj.org/pub/pdb/validation_reports/t0/5t00
ftp://data.pdbj.org/pub/pdb/validation_reports/t0/5t00 リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / 遺伝子: CTCF / プラスミド: pXC1551 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: CTCF / プラスミド: pXC1551 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 22-BM / 波長: 1 Å
/ ビームライン: 22-BM / 波長: 1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj









































