+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5oph | ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
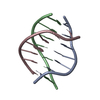
| タイトル | G-quadruplex structure of DNA oligonucleotide containing GGGGCC repeats linked to ALS and FTD | ||||||||||||||||||||||||||||
 要素 要素 | DNA (5'-D(* キーワード キーワードDNA / G-quadruplex / Nucelar Magnetic Resonance / K+ solution / Antiparallel / Four G-quartets / Edgewise loops / Stacking / GGGGCC-repat / ALS and FTD / Neurodegenerative disorders / Repetitive Sequences / Nucleic Acid / 8Br-dG / modified residue | 機能・相同性 | DNA / DNA (> 10) |  機能・相同性情報 機能・相同性情報生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)手法 | 溶液NMR / simulated annealing |  データ登録者 データ登録者Brcic, J. / Plavec, J. | 資金援助 | |  スロベニア, 1件 スロベニア, 1件
 引用 引用 ジャーナル: Nucleic Acids Res. / 年: 2018 ジャーナル: Nucleic Acids Res. / 年: 2018タイトル: NMR structure of a G-quadruplex formed by four d(G4C2) repeats: insights into structural polymorphism. 著者: Brcic, J. / Plavec, J. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5oph.cif.gz 5oph.cif.gz | 141.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5oph.ent.gz pdb5oph.ent.gz | 115.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5oph.json.gz 5oph.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5oph_validation.pdf.gz 5oph_validation.pdf.gz | 325.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5oph_full_validation.pdf.gz 5oph_full_validation.pdf.gz | 379.3 KB | 表示 | |
| XML形式データ |  5oph_validation.xml.gz 5oph_validation.xml.gz | 6.5 KB | 表示 | |
| CIF形式データ |  5oph_validation.cif.gz 5oph_validation.cif.gz | 10 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/op/5oph https://data.pdbj.org/pub/pdb/validation_reports/op/5oph ftp://data.pdbj.org/pub/pdb/validation_reports/op/5oph ftp://data.pdbj.org/pub/pdb/validation_reports/op/5oph | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 7036.323 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||||||||||||||||||||||
| 試料状態 | イオン強度: 53 mM / Label: 298 K / pH: 5.8 / 圧: 1 bar / 温度: 298 K |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 2 | ||||||||||||||||||||||||
| 代表構造 | 選択基準: closest to the average | ||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the least restraint violations 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 10 |
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj






































 HSQC
HSQC