| 登録情報 | データベース: PDB / ID: 5nce
|
|---|
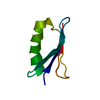
| タイトル | Structure of PsDef1 defensin from Pinus sylvestris |
|---|
 要素 要素 | Defensin-1 |
|---|
 キーワード キーワード | PLANT PROTEIN / Defensin / Plant Defensin / Scotts pine defensin / conformational dynamics / antibacterial activity / amylase inhibition |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
defense response to fungus / killing of cells of another organism / extracellular region類似検索 - 分子機能 Defensin, plant / Gamma-thionins family signature. / Gamma-thionin family / Knottins / Knottin, scorpion toxin-like / Knottin, scorpion toxin-like / Knottin, scorpion toxin-like superfamily / Defensin A-like / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Pinus sylvestris (植物) Pinus sylvestris (植物) |
|---|
| 手法 | 溶液NMR |
|---|
 データ登録者 データ登録者 | Khairutdinov, B.I. / Ermakova, E.A. / Bessolitsyna, E.K. / Toporkova, Y.Y. / Tarasova, N.B. / Kovaleva, V. / Zuev, Y.F. / Nesmelova, I.V. |
|---|
| 資金援助 |  ロシア, 3件 ロシア, 3件 | 組織 | 認可番号 | 国 |
|---|
| Russian Foundation for Basic Research | 12-04-01286-a |  ロシア ロシア | | Russian Foundation for Basic Research | 15-44-02309 |  ロシア ロシア | | Russian Foundation for Basic Research | 15-29-01239 |  ロシア ロシア |
|
|---|
 引用 引用 |  ジャーナル: Biochim. Biophys. Acta / 年: 2017 ジャーナル: Biochim. Biophys. Acta / 年: 2017
タイトル: NMR structure, conformational dynamics, and biological activity of PsDef1 defensin from Pinus sylvestris.
著者: Khairutdinov, B.I. / Ermakova, E.A. / Yusypovych, Y.M. / Bessolicina, E.K. / Tarasova, N.B. / Toporkova, Y.Y. / Kovaleva, V. / Zuev, Y.F. / Nesmelova, I.V. |
|---|
| 履歴 | | 登録 | 2017年3月3日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2017年6月7日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年6月14日 | Group: Database references / カテゴリ: citation
Item: _citation.country / _citation.journal_id_ASTM ..._citation.country / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_volume / _citation.page_first / _citation.page_last |
|---|
| 改定 1.2 | 2018年5月2日 | Group: Author supporting evidence / Data collection / Experimental preparation
カテゴリ: pdbx_audit_support / pdbx_nmr_exptl_sample_conditions
Item: _pdbx_audit_support.funding_organization / _pdbx_nmr_exptl_sample_conditions.pressure / _pdbx_nmr_exptl_sample_conditions.pressure_units |
|---|
| 改定 1.3 | 2024年11月6日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / pdbx_nmr_spectrometer
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_spectrometer.model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 ロシア, 3件
ロシア, 3件  引用
引用 ジャーナル: Biochim. Biophys. Acta / 年: 2017
ジャーナル: Biochim. Biophys. Acta / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5nce.cif.gz
5nce.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5nce.ent.gz
pdb5nce.ent.gz PDB形式
PDB形式 5nce.json.gz
5nce.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5nce_validation.pdf.gz
5nce_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5nce_full_validation.pdf.gz
5nce_full_validation.pdf.gz 5nce_validation.xml.gz
5nce_validation.xml.gz 5nce_validation.cif.gz
5nce_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/nc/5nce
https://data.pdbj.org/pub/pdb/validation_reports/nc/5nce ftp://data.pdbj.org/pub/pdb/validation_reports/nc/5nce
ftp://data.pdbj.org/pub/pdb/validation_reports/nc/5nce リンク
リンク 集合体
集合体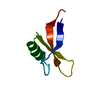
 要素
要素

 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj

 HSQC
HSQC