+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5mlw | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of human PCNA in complex with ZRANB3 APIM motif peptide | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | HYDROLASE / endonuclease / metalloprotein / PCNA / DNA-binding | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of deoxyribonuclease activity / dinucleotide insertion or deletion binding / PCNA-p21 complex / mitotic telomere maintenance via semi-conservative replication / purine-specific mismatch base pair DNA N-glycosylase activity / nuclear lamina / replication fork reversal / positive regulation of DNA-directed DNA polymerase activity / Polymerase switching / MutLalpha complex binding ...positive regulation of deoxyribonuclease activity / dinucleotide insertion or deletion binding / PCNA-p21 complex / mitotic telomere maintenance via semi-conservative replication / purine-specific mismatch base pair DNA N-glycosylase activity / nuclear lamina / replication fork reversal / positive regulation of DNA-directed DNA polymerase activity / Polymerase switching / MutLalpha complex binding / Telomere C-strand (Lagging Strand) Synthesis / Processive synthesis on the lagging strand / PCNA complex / Removal of the Flap Intermediate / Processive synthesis on the C-strand of the telomere / Polymerase switching on the C-strand of the telomere / Mismatch repair (MMR) directed by MSH2:MSH3 (MutSbeta) / Mismatch repair (MMR) directed by MSH2:MSH6 (MutSalpha) / Removal of the Flap Intermediate from the C-strand / Transcription of E2F targets under negative control by DREAM complex / ATP-dependent DNA/DNA annealing activity / replisome / response to L-glutamate / K63-linked polyubiquitin modification-dependent protein binding / response to dexamethasone / histone acetyltransferase binding / DNA polymerase processivity factor activity / leading strand elongation / G1/S-Specific Transcription / nuclear replication fork / replication fork processing / SUMOylation of DNA replication proteins / PCNA-Dependent Long Patch Base Excision Repair / response to cadmium ion / translesion synthesis / estrous cycle / mismatch repair / response to UV / cyclin-dependent protein kinase holoenzyme complex / base-excision repair, gap-filling / DNA polymerase binding / liver regeneration / epithelial cell differentiation / positive regulation of DNA repair / TP53 Regulates Transcription of Genes Involved in G2 Cell Cycle Arrest / Translesion synthesis by REV1 / Translesion synthesis by POLK / Translesion synthesis by POLI / replication fork / Gap-filling DNA repair synthesis and ligation in GG-NER / positive regulation of DNA replication / nuclear estrogen receptor binding / DNA endonuclease activity / male germ cell nucleus / Termination of translesion DNA synthesis / Recognition of DNA damage by PCNA-containing replication complex / helicase activity / Translesion Synthesis by POLH / receptor tyrosine kinase binding / HDR through Homologous Recombination (HRR) / Dual Incision in GG-NER / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / cellular response to xenobiotic stimulus / cellular response to hydrogen peroxide / Dual incision in TC-NER / Gap-filling DNA repair synthesis and ligation in TC-NER / cellular response to UV / response to estradiol / E3 ubiquitin ligases ubiquitinate target proteins / heart development / chromatin organization / damaged DNA binding / 加水分解酵素; エステル加水分解酵素 / chromosome, telomeric region / nuclear body / DNA repair / DNA damage response / centrosome / chromatin binding / chromatin / protein-containing complex binding / enzyme binding / negative regulation of transcription by RNA polymerase II / extracellular exosome / zinc ion binding / nucleoplasm / ATP binding / identical protein binding / nucleus 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.45 Å 分子置換 / 解像度: 2.45 Å | ||||||
 データ登録者 データ登録者 | Ariza, A. | ||||||
| 資金援助 |  英国, 1件 英国, 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: Structural insights into the function of ZRANB3 in replication stress response. 著者: Sebesta, M. / Cooper, C.D.O. / Ariza, A. / Carnie, C.J. / Ahel, D. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5mlw.cif.gz 5mlw.cif.gz | 315.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5mlw.ent.gz pdb5mlw.ent.gz | 259 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5mlw.json.gz 5mlw.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5mlw_validation.pdf.gz 5mlw_validation.pdf.gz | 478.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5mlw_full_validation.pdf.gz 5mlw_full_validation.pdf.gz | 484.5 KB | 表示 | |
| XML形式データ |  5mlw_validation.xml.gz 5mlw_validation.xml.gz | 29.5 KB | 表示 | |
| CIF形式データ |  5mlw_validation.cif.gz 5mlw_validation.cif.gz | 40.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ml/5mlw https://data.pdbj.org/pub/pdb/validation_reports/ml/5mlw ftp://data.pdbj.org/pub/pdb/validation_reports/ml/5mlw ftp://data.pdbj.org/pub/pdb/validation_reports/ml/5mlw | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 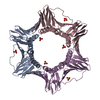
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 単位格子 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Component-ID: _ / Refine code: _
NCSアンサンブル:
|
- 要素
要素
| #1: タンパク質 | 分子量: 28795.752 Da / 分子数: 3 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: PCNA / 発現宿主: Homo sapiens (ヒト) / 遺伝子: PCNA / 発現宿主:  #2: タンパク質・ペプチド | 分子量: 1266.510 Da / 分子数: 3 / 由来タイプ: 合成 / 由来: (合成)  Homo sapiens (ヒト) / 参照: UniProt: Q5FWF4*PLUS Homo sapiens (ヒト) / 参照: UniProt: Q5FWF4*PLUS#3: 化合物 | ChemComp-SO4 / #4: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.4 Å3/Da / 溶媒含有率: 48.67 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7 詳細: 100 mM lithium sulfate + 30% (w/v) polyvinylpyrrolidone + 100 mM HEPES pH 7.0 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I03 / 波長: 1.2822 Å / ビームライン: I03 / 波長: 1.2822 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2015年12月12日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.2822 Å / 相対比: 1 |
| 反射 | 解像度: 2.45→73.29 Å / Num. obs: 31686 / % possible obs: 100 % / 冗長度: 11.2 % / CC1/2: 0.998 / Rmerge(I) obs: 0.104 / Net I/σ(I): 15.1 |
| 反射 シェル | 解像度: 2.45→2.55 Å / 冗長度: 9.7 % / Rmerge(I) obs: 1.297 / Mean I/σ(I) obs: 2 / CC1/2: 0.565 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 1VYM 解像度: 2.45→73.29 Å / Cor.coef. Fo:Fc: 0.94 / Cor.coef. Fo:Fc free: 0.925 / SU B: 25.41 / SU ML: 0.28 / 交差検証法: THROUGHOUT / ESU R: 0.645 / ESU R Free: 0.303 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 66.671 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 解像度: 2.45→73.29 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



















 PDBj
PDBj


















