+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5jlf | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
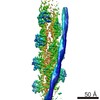
| タイトル | Structure of the F-actin-tropomyosin complex (Reprocessed) | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 要素 要素 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 キーワード キーワード | CONTRACTILE PROTEIN / CONTRACTILE FILAMENT / MUSCLE / THIN FILAMENT / CYTOSKELETON / STRUCTURAL PROTEIN / HYDROLASE COMPLEX / F-actin / tropomyosin / filament / protein polymers / cryo EM | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytoskeletal motor activator activity / myosin heavy chain binding / tropomyosin binding / actin filament bundle / troponin I binding / filamentous actin / mesenchyme migration / actin filament bundle assembly / skeletal muscle myofibril / striated muscle thin filament ...cytoskeletal motor activator activity / myosin heavy chain binding / tropomyosin binding / actin filament bundle / troponin I binding / filamentous actin / mesenchyme migration / actin filament bundle assembly / skeletal muscle myofibril / striated muscle thin filament / skeletal muscle thin filament assembly / actin monomer binding / skeletal muscle fiber development / stress fiber / titin binding / actin filament polymerization / actin filament / filopodium / 加水分解酵素; 酸無水物に作用; 酸無水物に作用・細胞または細胞小器官の運動に関与 / calcium-dependent protein binding / lamellipodium / cell body / hydrolase activity / protein domain specific binding / calcium ion binding / positive regulation of gene expression / magnesium ion binding / ATP binding / identical protein binding / cytoplasm 類似検索 - 分子機能 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 生物種 |   | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | von der Ecken, J. / Raunser, S. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 資金援助 |  ドイツ, 2件 ドイツ, 2件
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2016 ジャーナル: Nature / 年: 2016タイトル: Cryo-EM structure of a human cytoplasmic actomyosin complex at near-atomic resolution. 要旨: The interaction of myosin with actin filaments is the central feature of muscle contraction and cargo movement along actin filaments of the cytoskeleton. The energy for these movements is generated ...The interaction of myosin with actin filaments is the central feature of muscle contraction and cargo movement along actin filaments of the cytoskeleton. The energy for these movements is generated during a complex mechanochemical reaction cycle. Crystal structures of myosin in different states have provided important structural insights into the myosin motor cycle when myosin is detached from F-actin. The difficulty of obtaining diffracting crystals, however, has prevented structure determination by crystallography of actomyosin complexes. Thus, although structural models exist of F-actin in complex with various myosins, a high-resolution structure of the F-actin–myosin complex is missing. Here, using electron cryomicroscopy, we present the structure of a human rigor actomyosin complex at an average resolution of 3.9 Å. The structure reveals details of the actomyosin interface, which is mainly stabilized by hydrophobic interactions. The negatively charged amino (N) terminus of actin interacts with a conserved basic motif in loop 2 of myosin, promoting cleft closure in myosin. Surprisingly, the overall structure of myosin is similar to rigor-like myosin structures in the absence of F-actin, indicating that F-actin binding induces only minimal conformational changes in myosin. A comparison with pre-powerstroke and intermediate (Pi-release) states of myosin allows us to discuss the general mechanism of myosin binding to F-actin. Our results serve as a strong foundation for the molecular understanding of cytoskeletal diseases, such as autosomal dominant hearing loss and diseases affecting skeletal and cardiac muscles, in particular nemaline myopathy and hypertrophic cardiomyopathy. #1:  ジャーナル: Nature / 年: 2015 ジャーナル: Nature / 年: 2015タイトル: Structure of the F-actin-tropomyosin complex. 著者: Julian von der Ecken / Mirco Müller / William Lehman / Dietmar J Manstein / Pawel A Penczek / Stefan Raunser /   要旨: Filamentous actin (F-actin) is the major protein of muscle thin filaments, and actin microfilaments are the main component of the eukaryotic cytoskeleton. Mutations in different actin isoforms lead ...Filamentous actin (F-actin) is the major protein of muscle thin filaments, and actin microfilaments are the main component of the eukaryotic cytoskeleton. Mutations in different actin isoforms lead to early-onset autosomal dominant non-syndromic hearing loss, familial thoracic aortic aneurysms and dissections, and multiple variations of myopathies. In striated muscle fibres, the binding of myosin motors to actin filaments is mainly regulated by tropomyosin and troponin. Tropomyosin also binds to F-actin in smooth muscle and in non-muscle cells and stabilizes and regulates the filaments there in the absence of troponin. Although crystal structures for monomeric actin (G-actin) are available, a high-resolution structure of F-actin is still missing, hampering our understanding of how disease-causing mutations affect the function of thin muscle filaments and microfilaments. Here we report the three-dimensional structure of F-actin at a resolution of 3.7 Å in complex with tropomyosin at a resolution of 6.5 Å, determined by electron cryomicroscopy. The structure reveals that the D-loop is ordered and acts as a central region for hydrophobic and electrostatic interactions that stabilize the F-actin filament. We clearly identify map density corresponding to ADP and Mg(2+) and explain the possible effect of prominent disease-causing mutants. A comparison of F-actin with G-actin reveals the conformational changes during filament formation and identifies the D-loop as their key mediator. We also confirm that negatively charged tropomyosin interacts with a positively charged groove on F-actin. Comparison of the position of tropomyosin in F-actin-tropomyosin with its position in our previously determined F-actin-tropomyosin-myosin structure reveals a myosin-induced transition of tropomyosin. Our results allow us to understand the role of individual mutations in the genesis of actin- and tropomyosin-related diseases and will serve as a strong foundation for the targeted development of drugs. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5jlf.cif.gz 5jlf.cif.gz | 396.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5jlf.ent.gz pdb5jlf.ent.gz | 318.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5jlf.json.gz 5jlf.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5jlf_validation.pdf.gz 5jlf_validation.pdf.gz | 1 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5jlf_full_validation.pdf.gz 5jlf_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  5jlf_validation.xml.gz 5jlf_validation.xml.gz | 58 KB | 表示 | |
| CIF形式データ |  5jlf_validation.cif.gz 5jlf_validation.cif.gz | 87.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jl/5jlf https://data.pdbj.org/pub/pdb/validation_reports/jl/5jlf ftp://data.pdbj.org/pub/pdb/validation_reports/jl/5jlf ftp://data.pdbj.org/pub/pdb/validation_reports/jl/5jlf | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域:
NCS oper:
|
- 要素
要素
| #1: タンパク質 | 分子量: 41875.633 Da / 分子数: 5 / 由来タイプ: 天然 / 由来: (天然)  #2: タンパク質 | 分子量: 11507.176 Da / 分子数: 2 / 由来タイプ: 組換発現 詳細: MOUSE TROPOMYSIN WAS USED (UNP P58771, RESIDUES 97-231). DUE TO THE LIMITED RESOLUTION OF THE CRYO-EM DENSITY IN THE REGION OF TROPOMYOSIN, TROPOMYOSIN HAS BEEN REPRESENTED AS POLY(UNK). 由来: (組換発現)   #3: 化合物 | ChemComp-ADP / #4: 化合物 | ChemComp-MG / Has protein modification | Y | 配列の詳細 | MOUSE TROPOMYSIN WAS USED (UNP P58771, TPM1_MOUSE, RESIDUES 97-231). DUE TO THE LIMITED RESOLUTION ...MOUSE TROPOMYSIN | |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: FILAMENT / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.2 MDa / 実験値: NO | ||||||||||||||||||||||||||||
| 由来(天然) |
| ||||||||||||||||||||||||||||
| 由来(組換発現) | 生物種:  | ||||||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 詳細: 5 mM Tris-HCl pH 7.5, 1 mM DTT, 100 mM KCl, and 2 mM MgCl2 | ||||||||||||||||||||||||||||
| 緩衝液成分 |
| ||||||||||||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES | ||||||||||||||||||||||||||||
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: C-flat-2/1 | ||||||||||||||||||||||||||||
| 急速凍結 | 装置: GATAN CRYOPLUNGE 3 / 凍結剤: ETHANE / 湿度: 90 % 詳細: Sample was applied to a glow-discharged holey carbon grid, incubated for 10 s and manually blotted for 3 s from the backside with filter paper. |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS / 詳細: Cs corrected microscope |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: OTHER |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 59000 X / Calibrated defocus min: 800 nm / 最大 デフォーカス(補正後): 2600 nm |
| 撮影 | 平均露光時間: 0.475 sec. / 電子線照射量: 16 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: FEI FALCON II (4k x 4k) |
| 画像スキャン | 動画フレーム数/画像: 8 / 利用したフレーム数/画像: 2-8 |
- 解析
解析
| ソフトウェア | 名称: REFMAC / バージョン: 5.8.0088 / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| らせん対称 | 回転角度/サブユニット: 166.9 ° / 軸方向距離/サブユニット: 27.5 Å / らせん対称軸の対称性: C1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 119000 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3次元再構成 | 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 91000 詳細: THE TROPOMYOSIN MAP FILTERED TO 6.5 ANGSTROM WAS MERGED WITH THE FINAL F-ACTIN MAP (3.6 ANGSTROM) TO OBTAIN A MAP OF THE ENTIRE F-ACTIN-TROPOMYOSIN COMPLEX. 対称性のタイプ: HELICAL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 3J8A Accession code: 3J8A / Source name: PDB / タイプ: experimental model | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 3.6→193.6 Å / Cor.coef. Fo:Fc: 0.857 / SU B: 33.198 / SU ML: 0.463 / ESU R: 1.724 立体化学のターゲット値: MAXIMUM LIKELIHOOD WITH PHASES 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS MERGED WITH THE FINAL F-ACTIN MAP (3.6 ANGSTROM) TO OBTAIN A MAP OF THE ENTIRE F-ACTIN TROPOMYOSIN COMPLEX.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 97.923 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 合計: 14450 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj






