| 登録情報 | データベース: PDB / ID: 5j42
|
|---|
| タイトル | Crystal structure of m2hTDP2-CAT in complex with a small molecule inhibitor |
|---|
 要素 要素 | Tyrosyl-DNA phosphodiesterase 2 |
|---|
 キーワード キーワード | HYDROLASE / tyrosyl DNA phosphodiesterase 2 catalytic domain |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
tyrosyl-RNA phosphodiesterase activity / 5'-tyrosyl-DNA phosphodiesterase activity / Nonhomologous End-Joining (NHEJ) / 加水分解酵素; エステル加水分解酵素; リン酸ジエステル加水分解酵素 / phosphoric diester hydrolase activity / aggresome / neuron development / PML body / double-strand break repair / single-stranded DNA binding ...tyrosyl-RNA phosphodiesterase activity / 5'-tyrosyl-DNA phosphodiesterase activity / Nonhomologous End-Joining (NHEJ) / 加水分解酵素; エステル加水分解酵素; リン酸ジエステル加水分解酵素 / phosphoric diester hydrolase activity / aggresome / neuron development / PML body / double-strand break repair / single-stranded DNA binding / manganese ion binding / endonuclease activity / nucleolus / magnesium ion binding / cytoplasm類似検索 - 分子機能 Deoxyribonuclease I; Chain A / Endonuclease/exonuclease/phosphatase / Endonuclease/exonuclease/phosphatase / Endonuclease/Exonuclease/phosphatase family / Endonuclease/exonuclease/phosphatase superfamily / 4-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 Chem-6FV / : / L(+)-TARTARIC ACID / Tyrosyl-DNA phosphodiesterase 2類似検索 - 構成要素 |
|---|
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.7 Å 分子置換 / 解像度: 1.7 Å |
|---|
 データ登録者 データ登録者 | Hornyak, P. / Pearl, L.H. / Caldecott, K.W. / Oliver, A.W. |
|---|
| 資金援助 |  英国, 3件 英国, 3件 | 組織 | 認可番号 | 国 |
|---|
| Cancer Research UK | C302/A14532 |  英国 英国 | | Cancer Research UK | C6563/A16771 |  英国 英国 | | Cancer Research UK | C480/A11411 |  英国 英国 |
|
|---|
 引用 引用 |  ジャーナル: Biochem.J. / 年: 2016 ジャーナル: Biochem.J. / 年: 2016
タイトル: Mode of action of DNA-competitive small molecule inhibitors of tyrosyl DNA phosphodiesterase 2.
著者: Hornyak, P. / Askwith, T. / Walker, S. / Komulainen, E. / Paradowski, M. / Pennicott, L.E. / Bartlett, E.J. / Brissett, N.C. / Raoof, A. / Watson, M. / Jordan, A.M. / Ogilvie, D.J. / Ward, S. ...著者: Hornyak, P. / Askwith, T. / Walker, S. / Komulainen, E. / Paradowski, M. / Pennicott, L.E. / Bartlett, E.J. / Brissett, N.C. / Raoof, A. / Watson, M. / Jordan, A.M. / Ogilvie, D.J. / Ward, S.E. / Atack, J.R. / Pearl, L.H. / Caldecott, K.W. / Oliver, A.W. |
|---|
| 履歴 | | 登録 | 2016年3月31日 | 登録サイト: RCSB / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2016年5月4日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2016年7月6日 | Group: Database references |
|---|
| 改定 1.2 | 2018年1月24日 | Group: Source and taxonomy / カテゴリ: entity_src_gen / Item: _entity_src_gen.pdbx_host_org_ncbi_taxonomy_id |
|---|
| 改定 1.3 | 2019年10月16日 | Group: Data collection / カテゴリ: reflns / reflns_shell / Item: _reflns.pdbx_CC_half / _reflns_shell.pdbx_CC_half |
|---|
| 改定 1.4 | 2024年1月10日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / pdbx_struct_conn_angle / struct_conn
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.7 Å
分子置換 / 解像度: 1.7 Å  データ登録者
データ登録者 英国, 3件
英国, 3件  引用
引用 ジャーナル: Biochem.J. / 年: 2016
ジャーナル: Biochem.J. / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5j42.cif.gz
5j42.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5j42.ent.gz
pdb5j42.ent.gz PDB形式
PDB形式 5j42.json.gz
5j42.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5j42_validation.pdf.gz
5j42_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5j42_full_validation.pdf.gz
5j42_full_validation.pdf.gz 5j42_validation.xml.gz
5j42_validation.xml.gz 5j42_validation.cif.gz
5j42_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/j4/5j42
https://data.pdbj.org/pub/pdb/validation_reports/j4/5j42 ftp://data.pdbj.org/pub/pdb/validation_reports/j4/5j42
ftp://data.pdbj.org/pub/pdb/validation_reports/j4/5j42 リンク
リンク 集合体
集合体
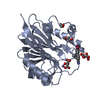

 要素
要素

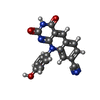










 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I04 / 波長: 0.9795 Å
/ ビームライン: I04 / 波長: 0.9795 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj






