| 登録情報 | データベース: PDB / ID: 5ith
|
|---|
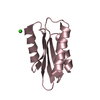
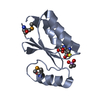
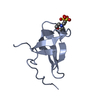
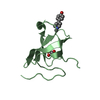
| タイトル | TIA-1 RRM2 recognition of target oligonucleotide |
|---|
 要素 要素 | - DNA (5'-D(*AP*CP*TP*CP*C*TP*TP*TP*TP*T)-3')
- Nucleolysin TIA-1 isoform p40
|
|---|
 キーワード キーワード | rna binding protein/dna / RRM / RNA/DNA binding domain / Poly U binding preference / aromatic base stacking interactions / DNA binding protein-DNA complex / rna binding protein-dna complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
protein localization to cytoplasmic stress granule / nuclear stress granule / poly(A) binding / mRNA 3'-UTR AU-rich region binding / regulation of mRNA splicing, via spliceosome / positive regulation of epithelial cell apoptotic process / FGFR2 alternative splicing / negative regulation of cytokine production / regulation of alternative mRNA splicing, via spliceosome / stress granule assembly ...protein localization to cytoplasmic stress granule / nuclear stress granule / poly(A) binding / mRNA 3'-UTR AU-rich region binding / regulation of mRNA splicing, via spliceosome / positive regulation of epithelial cell apoptotic process / FGFR2 alternative splicing / negative regulation of cytokine production / regulation of alternative mRNA splicing, via spliceosome / stress granule assembly / RNA splicing / mRNA 3'-UTR binding / mRNA processing / cytoplasmic stress granule / negative regulation of translation / ribonucleoprotein complex / apoptotic process / RNA binding / nucleoplasm / nucleus / cytoplasm / cytosol類似検索 - 分子機能 TIA-1, RNA recognition motif 1 / TIA-1, RNA recognition motif 2 / TIAR, RNA recognition motif 3 / RNA recognition motif domain, eukaryote / RNA recognition motif / RRM (RNA recognition motif) domain / RNA recognition motif / RNA recognition motif / Eukaryotic RNA Recognition Motif (RRM) profile. / RNA recognition motif domain ...TIA-1, RNA recognition motif 1 / TIA-1, RNA recognition motif 2 / TIAR, RNA recognition motif 3 / RNA recognition motif domain, eukaryote / RNA recognition motif / RRM (RNA recognition motif) domain / RNA recognition motif / RNA recognition motif / Eukaryotic RNA Recognition Motif (RRM) profile. / RNA recognition motif domain / RNA-binding domain superfamily / Nucleotide-binding alpha-beta plait domain superfamily / Alpha-Beta Plaits / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 DNA / Cytotoxic granule associated RNA binding protein TIA1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.31 Å 分子置換 / 解像度: 2.31 Å |
|---|
 データ登録者 データ登録者 | Waris, S. / Wilce, J.A. / Wilce, M.C. |
|---|
 引用 引用 |  ジャーナル: Nucleic Acids Res. / 年: 2017 ジャーナル: Nucleic Acids Res. / 年: 2017
タイトル: TIA-1 RRM23 binding and recognition of target oligonucleotides.
著者: Waris, S. / Garcia-Maurino, S.M. / Sivakumaran, A. / Beckham, S.A. / Loughlin, F.E. / Gorospe, M. / Diaz-Moreno, I. / Wilce, M.C.J. / Wilce, J.A. |
|---|
| 履歴 | | 登録 | 2016年3月16日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2017年2月22日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年5月17日 | Group: Database references |
|---|
| 改定 1.2 | 2023年9月27日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / diffrn_radiation_wavelength / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.31 Å
分子置換 / 解像度: 2.31 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nucleic Acids Res. / 年: 2017
ジャーナル: Nucleic Acids Res. / 年: 2017 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5ith.cif.gz
5ith.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5ith.ent.gz
pdb5ith.ent.gz PDB形式
PDB形式 5ith.json.gz
5ith.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/it/5ith
https://data.pdbj.org/pub/pdb/validation_reports/it/5ith ftp://data.pdbj.org/pub/pdb/validation_reports/it/5ith
ftp://data.pdbj.org/pub/pdb/validation_reports/it/5ith
 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: TIA1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: TIA1 / 発現宿主: 
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Australian Synchrotron
Australian Synchrotron  / ビームライン: MX2 / 波長: 0.7108 Å
/ ビームライン: MX2 / 波長: 0.7108 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj

