+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5ijd | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | The crystal structure of mouse TLR4/MD-2/lipid A complex | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | IMMUNE SYSTEM / immune response / protein complex / natural agonist | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報MyD88-independent TLR4 cascade / Caspase activation via Death Receptors in the presence of ligand / Toll Like Receptor 4 (TLR4) Cascade / Heme signaling / nitric oxide production involved in inflammatory response / Regulation of TLR by endogenous ligand / MHC class II biosynthetic process / TRIF-mediated programmed cell death / positive regulation of cellular response to macrophage colony-stimulating factor stimulus / TRAF6-mediated induction of TAK1 complex within TLR4 complex ...MyD88-independent TLR4 cascade / Caspase activation via Death Receptors in the presence of ligand / Toll Like Receptor 4 (TLR4) Cascade / Heme signaling / nitric oxide production involved in inflammatory response / Regulation of TLR by endogenous ligand / MHC class II biosynthetic process / TRIF-mediated programmed cell death / positive regulation of cellular response to macrophage colony-stimulating factor stimulus / TRAF6-mediated induction of TAK1 complex within TLR4 complex / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / Regulation of TBK1, IKKε (IKBKE)-mediated activation of IRF3, IRF7 / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / lipopolysaccharide immune receptor activity / positive regulation of nucleotide-binding oligomerization domain containing 1 signaling pathway / positive regulation of matrix metallopeptidase secretion / Toll-like receptor 4 binding / IKK complex recruitment mediated by RIP1 / mast cell activation / detection of lipopolysaccharide / positive regulation of lymphocyte proliferation / regulation of dendritic cell cytokine production / lipopolysaccharide receptor complex / negative regulation of interleukin-23 production / cellular response to oxidised low-density lipoprotein particle stimulus / wound healing involved in inflammatory response / B cell proliferation involved in immune response / lymphocyte proliferation / nucleotide-binding oligomerization domain containing 1 signaling pathway / positive regulation of nucleotide-binding oligomerization domain containing 2 signaling pathway / positive regulation of stress-activated MAPK cascade / intestinal epithelial structure maintenance / activation of NF-kappaB-inducing kinase activity / positive regulation of interleukin-13 production / positive regulation of interleukin-1 production / macrophage activation / TRIF-dependent toll-like receptor signaling pathway / astrocyte development / microglia differentiation / nucleotide-binding oligomerization domain containing 2 signaling pathway / NAD+ nucleosidase activity, cyclic ADP-ribose generating / positive regulation of MHC class II biosynthetic process / positive regulation of macrophage activation / positive regulation of platelet activation / positive regulation of lipopolysaccharide-mediated signaling pathway / negative regulation of interleukin-17 production / positive regulation of cytokine production involved in inflammatory response / positive regulation of chemokine (C-X-C motif) ligand 2 production / positive regulation of extrinsic apoptotic signaling pathway / negative regulation of cold-induced thermogenesis / positive regulation of smooth muscle cell migration / MyD88-dependent toll-like receptor signaling pathway / positive regulation of macrophage cytokine production / toll-like receptor 4 signaling pathway / toll-like receptor signaling pathway / positive regulation of NLRP3 inflammasome complex assembly / positive regulation of reactive oxygen species biosynthetic process / cellular response to lipoteichoic acid / negative regulation of type II interferon production / negative regulation of interleukin-6 production / B cell proliferation / positive regulation of interferon-alpha production / positive regulation of interleukin-10 production / negative regulation of tumor necrosis factor production / phagocytosis / phagocytic cup / stress-activated MAPK cascade / positive regulation of chemokine production / cellular response to platelet-derived growth factor stimulus / ruffle / JNK cascade / positive regulation of B cell proliferation / ERK1 and ERK2 cascade / neurogenesis / positive regulation of interleukin-12 production / nitric oxide biosynthetic process / positive regulation of smooth muscle cell proliferation / activation of innate immune response / positive regulation of interferon-beta production / lipopolysaccharide-mediated signaling pathway / positive regulation of interleukin-1 beta production / positive regulation of interleukin-8 production / positive regulation of JNK cascade / response to bacterium / lipopolysaccharide binding / cellular response to mechanical stimulus / positive regulation of non-canonical NF-kappaB signal transduction / : / negative regulation of ERK1 and ERK2 cascade / cellular response to type II interferon / positive regulation of interleukin-6 production / positive regulation of type II interferon production / cellular response to amyloid-beta / positive regulation of inflammatory response / positive regulation of nitric oxide biosynthetic process / positive regulation of tumor necrosis factor production / transmembrane signaling receptor activity / cellular response to lipopolysaccharide / regulation of inflammatory response / response to lipopolysaccharide 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Eptatretus burgeri (ヌタウナギ) Eptatretus burgeri (ヌタウナギ) | ||||||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.7 Å 分子置換 / 解像度: 2.7 Å | ||||||||||||
 データ登録者 データ登録者 | Wang, Y. / Su, L. / Morin, M.D. / Jones, B.T. / Whitby, L.R. / Surakattula, M. / Huang, H. / Shi, H. / Choi, J.H. / Wang, K. ...Wang, Y. / Su, L. / Morin, M.D. / Jones, B.T. / Whitby, L.R. / Surakattula, M. / Huang, H. / Shi, H. / Choi, J.H. / Wang, K. / Moresco, E.M. / Berger, M. / Zhan, X. / Zhang, H. / Boger, D.L. / Beutler, B. | ||||||||||||
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2016 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2016タイトル: TLR4/MD-2 activation by a synthetic agonist with no similarity to LPS. 著者: Wang, Y. / Su, L. / Morin, M.D. / Jones, B.T. / Whitby, L.R. / Surakattula, M.M. / Huang, H. / Shi, H. / Choi, J.H. / Wang, K.W. / Moresco, E.M. / Berger, M. / Zhan, X. / Zhang, H. / Boger, D.L. / Beutler, B. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5ijd.cif.gz 5ijd.cif.gz | 325 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5ijd.ent.gz pdb5ijd.ent.gz | 256.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5ijd.json.gz 5ijd.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5ijd_validation.pdf.gz 5ijd_validation.pdf.gz | 3.5 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5ijd_full_validation.pdf.gz 5ijd_full_validation.pdf.gz | 3.6 MB | 表示 | |
| XML形式データ |  5ijd_validation.xml.gz 5ijd_validation.xml.gz | 63 KB | 表示 | |
| CIF形式データ |  5ijd_validation.cif.gz 5ijd_validation.cif.gz | 85.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ij/5ijd https://data.pdbj.org/pub/pdb/validation_reports/ij/5ijd ftp://data.pdbj.org/pub/pdb/validation_reports/ij/5ijd ftp://data.pdbj.org/pub/pdb/validation_reports/ij/5ijd | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 2種, 4分子 ABCD
| #1: タンパク質 | 分子量: 67280.703 Da / 分子数: 2 断片: TLR4 ectodomain (UNP residues 26-544) + VLRB (UNP residues 126-200) 由来タイプ: 組換発現 由来: (組換発現)   Eptatretus burgeri (ヌタウナギ) Eptatretus burgeri (ヌタウナギ)遺伝子: Tlr4, Lps, VLRB 発現宿主:  参照: UniProt: Q9QUK6, UniProt: Q4G1L2 #2: タンパク質 | 分子量: 17392.793 Da / 分子数: 2 / 断片: UNP residues 19-160 / 由来タイプ: 組換発現 / 由来: (組換発現)  発現宿主:  参照: UniProt: Q9JHF9 |
|---|
-糖 , 2種, 12分子 
| #3: 多糖 | 2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose #4: 糖 | ChemComp-NAG / |
|---|
-非ポリマー , 5種, 240分子 

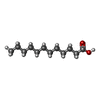






| #5: 化合物 | | #6: 化合物 | #7: 化合物 | #8: 化合物 | #9: 水 | ChemComp-HOH / | |
|---|
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.93 Å3/Da / 溶媒含有率: 68.73 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8 詳細: 0.1 M Tris, pH 8.0, 0.2 M lithium chloride, 14% PEG8000, 2.5% PEG400 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 19-ID / 波長: 0.97918 Å / ビームライン: 19-ID / 波長: 0.97918 Å |
| 検出器 | タイプ: ADSC QUANTUM 315r / 検出器: CCD / 日付: 2015年6月17日 |
| 放射 | モノクロメーター: double crystal Si(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.97918 Å / 相対比: 1 |
| 反射 | 解像度: 2.7→50 Å / Num. obs: 54848 / % possible obs: 96.2 % / 冗長度: 4 % / Rmerge(I) obs: 0.058 / Net I/av σ(I): 22.386 / Net I/σ(I): 8.3 |
| 反射 シェル | 解像度: 2.7→2.75 Å |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB entry 5IJD 解像度: 2.7→39.34 Å / Cor.coef. Fo:Fc: 0.915 / Cor.coef. Fo:Fc free: 0.842 / SU B: 14.782 / SU ML: 0.294 / 交差検証法: THROUGHOUT / ESU R: 1.232 / ESU R Free: 0.41 / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 43.652 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.7→39.34 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj












