[English] 日本語
 Yorodumi
Yorodumi- PDB-5fwr: Breaking down the wall: mutation of the tyrosine gate of the univ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 5fwr | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Breaking down the wall: mutation of the tyrosine gate of the universal Escherichia coli fimbrial adhesin FimH | |||||||||
 Components Components | FimH | |||||||||
 Keywords Keywords | CELL ADHESION / BACTERIAL ADHESIN / TYPE 1 FIMBRIAE / URINARY TRACT INFECTION / VARIABLE IMMUNOGLOBULIN FOLD | |||||||||
| Function / homology |  Function and homology information Function and homology informationpilus tip / mechanosensory behavior / cell adhesion involved in single-species biofilm formation / pilus / cell-substrate adhesion / D-mannose binding / host cell membrane / cell adhesion / metal ion binding Similarity search - Function | |||||||||
| Biological species |  | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.13 Å MOLECULAR REPLACEMENT / Resolution: 2.13 Å | |||||||||
 Authors Authors | Bouckaert, J. | |||||||||
 Citation Citation |  Journal: IUCrJ / Year: 2017 Journal: IUCrJ / Year: 2017Title: Mutation of Tyr137 of the universal Escherichia coli fimbrial adhesin FimH relaxes the tyrosine gate prior to mannose binding. Authors: Rabbani, S. / Krammer, E.M. / Roos, G. / Zalewski, A. / Preston, R. / Eid, S. / Zihlmann, P. / Prevost, M. / Lensink, M.F. / Thompson, A. / Ernst, B. / Bouckaert, J. | |||||||||
| History |
|
- Structure visualization
Structure visualization
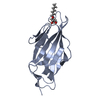
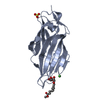
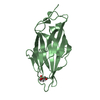
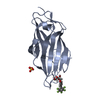
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  5fwr.cif.gz 5fwr.cif.gz | 758.5 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb5fwr.ent.gz pdb5fwr.ent.gz | 644.4 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  5fwr.json.gz 5fwr.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/fw/5fwr https://data.pdbj.org/pub/pdb/validation_reports/fw/5fwr ftp://data.pdbj.org/pub/pdb/validation_reports/fw/5fwr ftp://data.pdbj.org/pub/pdb/validation_reports/fw/5fwr | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  4ca4C 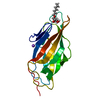 5fs5C  5fx3C  4auuS C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 5 | 
| ||||||||
| 6 | 
| ||||||||
| 7 | 
| ||||||||
| 8 | 
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 16916.828 Da / Num. of mol.: 8 / Fragment: FIMH LECTIN DOMAIN, RESIDUES 22-179 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   #2: Chemical | ChemComp-3X8 / #3: Water | ChemComp-HOH / | Has protein modification | Y | Nonpolymer details | METHYL 4'-(ALPHA-D-MANNOPYRANOSYLOXY)BIPHENYL-3-CARBOXYLATE (3XB): THE BIPHENYL GROUP STACKS ONTO ...METHYL 4'-(ALPHA-D-MANNOPYRAN | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.44 Å3/Da / Density % sol: 46.9 % / Description: NONE |
|---|---|
| Crystal grow | pH: 7.8 Details: 5% W/V PGA-LM 100 MM TRIS PH 7.8 ; 20% W/V PEG 3350 ; 5 MM 4-BIPHENYL ALPHA-D-MANNOPYRANOSIDE |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  BESSY BESSY  / Beamline: 14.2 / Wavelength: 0.91841 / Beamline: 14.2 / Wavelength: 0.91841 |
| Detector | Type: MARRESEARCH / Detector: CCD / Date: Apr 19, 2012 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.91841 Å / Relative weight: 1 |
| Reflection | Resolution: 2.13→47.63 Å / Num. obs: 85074 / % possible obs: 94.5 % / Observed criterion σ(I): 0 / Redundancy: 2.32 % / Rmerge(I) obs: 0.15 / Net I/σ(I): 6.13 |
| Reflection shell | Resolution: 2.13→2.26 Å / Redundancy: 2.32 % / Rmerge(I) obs: 0.48 / Mean I/σ(I) obs: 2.57 / % possible all: 81.9 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB ENTRY 4AUU Resolution: 2.13→47.627 Å / SU ML: 0.39 / σ(F): 2 / Phase error: 38.72 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.13→47.627 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Origin x: 21.8366 Å / Origin y: 8.1274 Å / Origin z: -26.6852 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group | Selection details: ALL |
 Movie
Movie Controller
Controller


















 PDBj
PDBj