| 登録情報 | データベース: PDB / ID: 5f1a
|
|---|
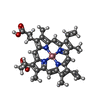
| タイトル | The Crystal Structure of Salicylate Bound to Human Cyclooxygenase-2 |
|---|
 要素 要素 | Prostaglandin G/H synthase 2 |
|---|
 キーワード キーワード | OXIDOREDUCTASE/INHIBITOR / Membrane Protein / Monotopic / Cyclooxygenase / Cyclooxygenase-2 / COX / PGHS / Salicylic acid / Salicylate / Inhibitor / Complex / OXIDOREDUCTASE-INHIBITOR complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
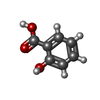
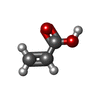
positive regulation of platelet-derived growth factor production / Biosynthesis of EPA-derived SPMs / Biosynthesis of DPAn-3 SPMs / Biosynthesis of DHA-derived SPMs / Biosynthesis of electrophilic ω-3 PUFA oxo-derivatives / oxidoreductase activity, acting on single donors with incorporation of molecular oxygen / Synthesis of 15-eicosatetraenoic acid derivatives / positive regulation of fibroblast growth factor production / cellular response to non-ionic osmotic stress / positive regulation of transforming growth factor beta production ...positive regulation of platelet-derived growth factor production / Biosynthesis of EPA-derived SPMs / Biosynthesis of DPAn-3 SPMs / Biosynthesis of DHA-derived SPMs / Biosynthesis of electrophilic ω-3 PUFA oxo-derivatives / oxidoreductase activity, acting on single donors with incorporation of molecular oxygen / Synthesis of 15-eicosatetraenoic acid derivatives / positive regulation of fibroblast growth factor production / cellular response to non-ionic osmotic stress / positive regulation of transforming growth factor beta production / lipoxygenase pathway / prostaglandin-endoperoxide synthase / prostaglandin-endoperoxide synthase activity / cyclooxygenase pathway / negative regulation of intrinsic apoptotic signaling pathway in response to osmotic stress / oxidoreductase activity, acting on single donors with incorporation of molecular oxygen, incorporation of two atoms of oxygen / positive regulation of prostaglandin biosynthetic process / regulation of neuroinflammatory response / Synthesis of Prostaglandins (PG) and Thromboxanes (TX) / positive regulation of fever generation / : / response to selenium ion / cellular response to fluid shear stress / prostaglandin secretion / response to nematode / nuclear inner membrane / prostaglandin biosynthetic process / positive regulation of cell migration involved in sprouting angiogenesis / long-chain fatty acid biosynthetic process / Interleukin-10 signaling / decidualization / nuclear outer membrane / positive regulation of vascular endothelial growth factor production / brown fat cell differentiation / positive regulation of brown fat cell differentiation / embryo implantation / peroxidase activity / regulation of blood pressure / positive regulation of nitric oxide biosynthetic process / regulation of cell population proliferation / response to oxidative stress / regulation of inflammatory response / Interleukin-4 and Interleukin-13 signaling / cellular response to hypoxia / neuron projection / endoplasmic reticulum lumen / intracellular membrane-bounded organelle / heme binding / endoplasmic reticulum membrane / enzyme binding / endoplasmic reticulum / protein homodimerization activity / metal ion binding / cytosol / cytoplasm類似検索 - 分子機能 : / Myeloperoxidase, subunit C / Haem peroxidase domain superfamily, animal type / Haem peroxidase, animal-type / Haem peroxidase domain superfamily, animal type / Animal haem peroxidase / Animal heme peroxidase superfamily profile. / Laminin / Laminin / EGF-like domain ...: / Myeloperoxidase, subunit C / Haem peroxidase domain superfamily, animal type / Haem peroxidase, animal-type / Haem peroxidase domain superfamily, animal type / Animal haem peroxidase / Animal heme peroxidase superfamily profile. / Laminin / Laminin / EGF-like domain / Haem peroxidase superfamily / EGF-like domain profile. / EGF-like domain / Ribbon / Orthogonal Bundle / Mainly Beta / Mainly Alpha類似検索 - ドメイン・相同性 ACRYLIC ACID / PROTOPORPHYRIN IX CONTAINING CO / 2-HYDROXYBENZOIC ACID / Prostaglandin G/H synthase 2類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.38 Å 分子置換 / 解像度: 2.38 Å |
|---|
 データ登録者 データ登録者 | Lucido, M.J. / Orlando, B.J. / Malkowski, M.G. |
|---|
| 資金援助 |  米国, 1件 米国, 1件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | GM077176 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: Biochemistry / 年: 2016 ジャーナル: Biochemistry / 年: 2016
タイトル: Crystal Structure of Aspirin-Acetylated Human Cyclooxygenase-2: Insight into the Formation of Products with Reversed Stereochemistry.
著者: Lucido, M.J. / Orlando, B.J. / Vecchio, A.J. / Malkowski, M.G. |
|---|
| 履歴 | | 登録 | 2015年11月30日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2016年3月16日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2017年9月27日 | Group: Author supporting evidence / Derived calculations / Refinement description
カテゴリ: pdbx_audit_support / pdbx_struct_oper_list / software
Item: _pdbx_audit_support.funding_organization / _pdbx_struct_oper_list.symmetry_operation / _software.classification |
|---|
| 改定 1.2 | 2019年11月20日 | Group: Derived calculations / カテゴリ: pdbx_struct_conn_angle / struct_conn |
|---|
| 改定 1.3 | 2019年12月25日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 2.0 | 2020年7月29日 | Group: Atomic model / Data collection ...Atomic model / Data collection / Derived calculations / Structure summary
カテゴリ: atom_site / atom_site_anisotrop ...atom_site / atom_site_anisotrop / chem_comp / entity / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_nonpoly_scheme / pdbx_struct_assembly_gen / pdbx_struct_conn_angle / struct_asym / struct_conn / struct_site / struct_site_gen
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.type_symbol / _atom_site_anisotrop.U[1][1] / _atom_site_anisotrop.U[1][2] / _atom_site_anisotrop.U[1][3] / _atom_site_anisotrop.U[2][2] / _atom_site_anisotrop.U[2][3] / _atom_site_anisotrop.U[3][3] / _atom_site_anisotrop.id / _atom_site_anisotrop.pdbx_auth_asym_id / _atom_site_anisotrop.pdbx_auth_atom_id / _atom_site_anisotrop.pdbx_auth_comp_id / _atom_site_anisotrop.pdbx_auth_seq_id / _atom_site_anisotrop.pdbx_label_asym_id / _atom_site_anisotrop.pdbx_label_atom_id / _atom_site_anisotrop.pdbx_label_comp_id / _atom_site_anisotrop.type_symbol / _chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.type / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _struct_conn.conn_type_id / _struct_conn.id / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_ptnr1_label_alt_id / _struct_conn.pdbx_role / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 2.1 | 2024年11月6日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / pdbx_validate_chiral / struct_conn
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI ..._chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_leaving_atom_flag |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.38 Å
分子置換 / 解像度: 2.38 Å  データ登録者
データ登録者 米国, 1件
米国, 1件  引用
引用 ジャーナル: Biochemistry / 年: 2016
ジャーナル: Biochemistry / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5f1a.cif.gz
5f1a.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5f1a.ent.gz
pdb5f1a.ent.gz PDB形式
PDB形式 5f1a.json.gz
5f1a.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5f1a_validation.pdf.gz
5f1a_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5f1a_full_validation.pdf.gz
5f1a_full_validation.pdf.gz 5f1a_validation.xml.gz
5f1a_validation.xml.gz 5f1a_validation.cif.gz
5f1a_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/f1/5f1a
https://data.pdbj.org/pub/pdb/validation_reports/f1/5f1a ftp://data.pdbj.org/pub/pdb/validation_reports/f1/5f1a
ftp://data.pdbj.org/pub/pdb/validation_reports/f1/5f1a リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: PTGS2, COX2 / プラスミド: pFastBac1
Homo sapiens (ヒト) / 遺伝子: PTGS2, COX2 / プラスミド: pFastBac1









 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 17-ID / 波長: 1.033 Å
/ ビームライン: 17-ID / 波長: 1.033 Å 分子置換
分子置換 解析
解析 分子置換 / 解像度: 2.38→33.324 Å / SU ML: 0.24 / 交差検証法: FREE R-VALUE / σ(F): 1.33 / 位相誤差: 21.9 / 立体化学のターゲット値: ML
分子置換 / 解像度: 2.38→33.324 Å / SU ML: 0.24 / 交差検証法: FREE R-VALUE / σ(F): 1.33 / 位相誤差: 21.9 / 立体化学のターゲット値: ML ムービー
ムービー コントローラー
コントローラー

























 PDBj
PDBj











