| 登録情報 | データベース: PDB / ID: 5ci9
|
|---|
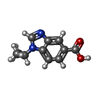
| タイトル | Crystal structure of human Tob in complex with inhibitor fragment 6 |
|---|
 要素 要素 | Protein Tob1 |
|---|
 キーワード キーワード | TRANSCRIPTION/INHIBITOR / Tob/BTG family / TRANSCRIPTION-INHIBITOR complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
negative regulation of nuclear-transcribed mRNA poly(A) tail shortening / regulation of SMAD protein signal transduction / CCR4-NOT complex / positive regulation of nuclear-transcribed mRNA catabolic process, deadenylation-dependent decay / positive regulation of nuclear-transcribed mRNA poly(A) tail shortening / SMAD binding / negative regulation of BMP signaling pathway / negative regulation of osteoblast differentiation / receptor tyrosine kinase binding / transcription corepressor activity ...negative regulation of nuclear-transcribed mRNA poly(A) tail shortening / regulation of SMAD protein signal transduction / CCR4-NOT complex / positive regulation of nuclear-transcribed mRNA catabolic process, deadenylation-dependent decay / positive regulation of nuclear-transcribed mRNA poly(A) tail shortening / SMAD binding / negative regulation of BMP signaling pathway / negative regulation of osteoblast differentiation / receptor tyrosine kinase binding / transcription corepressor activity / regulation of gene expression / negative regulation of translation / negative regulation of cell population proliferation / nucleus / cytoplasm類似検索 - 分子機能 Tob1/2 / Anti-proliferative protein, N-terminal domain / BTG family signature 1. / BTG family signature 2. / Anti-proliferative protein / BTG-like domain superfamily / BTG family / tob/btg1 family / Actin; Chain A, domain 4 / Alpha-Beta Complex / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2.3 Å シンクロトロン / 解像度: 2.3 Å |
|---|
 データ登録者 データ登録者 | Bai, Y. / Tashiro, S. / Nagatoishi, S. / Suzuki, T. / Tsumoto, K. / Bartlam, M. / Yamamoto, T. |
|---|
| 資金援助 |  中国, 中国,  日本, 3件 日本, 3件 | 組織 | 認可番号 | 国 |
|---|
| Ministry of Science and Technology | 2014CB560709 |  中国 中国 | | Ministry of Education, Culture, Sports, Science and Technology (Japan) | |  日本 日本 | | KAKENHI | 25121734 |  日本 日本 |
|
|---|
 引用 引用 |  ジャーナル: Protein Cell / 年: 2015 ジャーナル: Protein Cell / 年: 2015
タイトル: Structural basis for inhibition of the Tob-CNOT7 interaction by a fragment screening approach
著者: Bai, Y. / Tashiro, S. / Nagatoishi, S. / Suzuki, T. / Yan, D. / Liu, R. / Tsumoto, K. / Bartlam, M. / Yamamoto, T. |
|---|
| 履歴 | | 登録 | 2015年7月11日 | 登録サイト: RCSB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2015年11月18日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2015年12月9日 | Group: Database references |
|---|
| 改定 1.2 | 2024年3月20日 | Group: Author supporting evidence / Data collection ...Author supporting evidence / Data collection / Database references / Derived calculations
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_audit_support / pdbx_struct_oper_list
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_audit_support.funding_organization / _pdbx_struct_oper_list.symmetry_operation |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン / 解像度: 2.3 Å
シンクロトロン / 解像度: 2.3 Å  データ登録者
データ登録者 中国,
中国,  日本, 3件
日本, 3件  引用
引用 ジャーナル: Protein Cell / 年: 2015
ジャーナル: Protein Cell / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5ci9.cif.gz
5ci9.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5ci9.ent.gz
pdb5ci9.ent.gz PDB形式
PDB形式 5ci9.json.gz
5ci9.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5ci9_validation.pdf.gz
5ci9_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5ci9_full_validation.pdf.gz
5ci9_full_validation.pdf.gz 5ci9_validation.xml.gz
5ci9_validation.xml.gz 5ci9_validation.cif.gz
5ci9_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ci/5ci9
https://data.pdbj.org/pub/pdb/validation_reports/ci/5ci9 ftp://data.pdbj.org/pub/pdb/validation_reports/ci/5ci9
ftp://data.pdbj.org/pub/pdb/validation_reports/ci/5ci9 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: TOB1, TOB, TROB1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: TOB1, TOB, TROB1 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL17U / 波長: 1 Å
/ ビームライン: BL17U / 波長: 1 Å 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj