+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5bna | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | THE PRIMARY MODE OF BINDING OF CISPLATIN TO A B-DNA DODECAMER: C-G-C-G-A-A-T-T-C-G-C-G | ||||||||||||||||||
 要素 要素 | DNA (5'-D(* キーワード キーワードDNA / B-DNA / DOUBLE HELIX / COMPLEXED WITH DRUG / MODIFIED | 機能・相同性 | PLATINUM TRIAMINE ION / DNA / DNA (> 10) |  機能・相同性情報 機能・相同性情報手法 |  X線回折 / 解像度: 2.6 Å X線回折 / 解像度: 2.6 Å  データ登録者 データ登録者Wing, R.M. / Pjura, P. / Drew, H.R. / Dickerson, R.E. |  引用 引用 ジャーナル: EMBO J. / 年: 1984 ジャーナル: EMBO J. / 年: 1984タイトル: The primary mode of binding of cisplatin to a B-DNA dodecamer: C-G-C-G-A-A-T-T-C-G-C-G 著者: Wing, R.M. / Pjura, P. / Drew, H.R. / Dickerson, R.E. #1:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1983 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1983タイトル: A Random-Walk Model for Helix Bending in B-DNA 著者: Dickerson, R.E. / Kopka, M.L. / Pjura, P. #2:  ジャーナル: Cold Spring Harbor Symp.Quant.Biol. / 年: 1983 ジャーナル: Cold Spring Harbor Symp.Quant.Biol. / 年: 1983タイトル: Helix Geometry and Hydration in A-DNA, B-DNA and Z-DNA 著者: Dickerson, R.E. / Drew, H.R. / Conner, B.N. / Kopka, M.L. / Pjura, P.E. #3:  ジャーナル: Biological Macromolecules and Assemblies / 年: 1985 ジャーナル: Biological Macromolecules and Assemblies / 年: 1985タイトル: Base Sequence, Helix Geometry, Hydration and Helix Stability in B-DNA 著者: Dickerson, R.E. / Kopka, M.L. / Pjura, P. #4:  ジャーナル: Nature / 年: 1980 ジャーナル: Nature / 年: 1980タイトル: Crystal Structure Analysis of a Complete Turn of B-DNA 著者: Wing, R. / Drew, H.R. / Takano, T. / Broka, C. / Tanaka, S. / Itakura, K. / Dickerson, R.E. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5bna.cif.gz 5bna.cif.gz | 28.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5bna.ent.gz pdb5bna.ent.gz | 18.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5bna.json.gz 5bna.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5bna_validation.pdf.gz 5bna_validation.pdf.gz | 365.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5bna_full_validation.pdf.gz 5bna_full_validation.pdf.gz | 380.8 KB | 表示 | |
| XML形式データ |  5bna_validation.xml.gz 5bna_validation.xml.gz | 4.5 KB | 表示 | |
| CIF形式データ |  5bna_validation.cif.gz 5bna_validation.cif.gz | 6.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bn/5bna https://data.pdbj.org/pub/pdb/validation_reports/bn/5bna ftp://data.pdbj.org/pub/pdb/validation_reports/bn/5bna ftp://data.pdbj.org/pub/pdb/validation_reports/bn/5bna | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 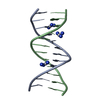
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Atom site foot note | 1: THE ANISOTROPIC TEMPERATURE FACTORS FOR THE PLATINUM ATOMS ARE AS FOLLOWS ATOM B11 B22 B33 B12 B13 B23 PT1 PTN 25 0.01510 0.00438 0.00138 0.00214 0.00056 0.00026 PT1 PTN 26 0.01545 0.00544 0.00131- ...1: THE ANISOTROPIC TEMPERATURE FACTORS FOR THE PLATINUM ATOMS ARE AS FOLLOWS ATOM B11 B22 B33 B12 B13 B23 PT1 PTN 25 0.01510 0.00438 0.00138 0.00214 0.00056 0.00026 PT1 PTN 26 0.01545 0.00544 0.00131-0.00462-0.00072 0.00017 PT1 PTN 27 0.00729 0.00382 0.00120-0.00092-0.00060 0.00039 2: TEMPERATURE FACTORS WHICH WERE CALCULATED TO BE NON-POSITIVE-DEFINITE WERE ARBITRARILY SET TO 0.0 IN THE REFINEMENT PROCEDURE. |
- 要素
要素
| #1: DNA鎖 | 分子量: 3663.392 Da / 分子数: 2 / 由来タイプ: 合成 #2: 化合物 | #3: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.18 Å3/Da / 溶媒含有率: 43.49 % | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 277 K / 手法: 蒸気拡散法 / 詳細: VAPOR DIFFUSION, temperature 277.00K | |||||||||||||||||||||||||
| 溶液の組成 |
| |||||||||||||||||||||||||
| 結晶化 | *PLUS 詳細: Wing, R., (1980) Nature, 287, 755. | |||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 反射 | 最高解像度: 2.6 Å / Num. obs: 2088 |
|---|
- 解析
解析
| ソフトウェア | 名称: JACK-LEVITT / 分類: 精密化 | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2.6→8 Å / σ(F): 2 /
| ||||||||||||||||
| Refine Biso |
| ||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.6→8 Å
| ||||||||||||||||
| 精密化 | *PLUS 最高解像度: 2.6 Å / 最低解像度: 8 Å / Num. reflection all: 2012 / σ(F): 2 / Rfactor all: 0.166 / Rfactor obs: 0.112 | ||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj









































