+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5a42 | ||||||
|---|---|---|---|---|---|---|---|
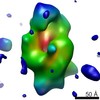
| タイトル | Cryo-EM single particle 3D reconstruction of the native conformation of E. coli alpha-2-macroglobulin (ECAM) | ||||||
 要素 要素 | UNCHARACTERIZED LIPOPROTEIN YFHM | ||||||
 キーワード キーワード | HYDROLASE INHIBITOR / PEPTIDASE INHIBITOR | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報endopeptidase inhibitor activity / defense response / protein homodimerization activity / extracellular space / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 16 Å | ||||||
 データ登録者 データ登録者 | Garcia-Ferrer, I. / Arede, P. / Gomez-Blanco, J. / Luque, D. / Duquerroy, S. / Caston, J.R. / Goulas, T. / Gomis-Ruth, F.X. | ||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2015 ジャーナル: Proc Natl Acad Sci U S A / 年: 2015タイトル: Structural and functional insights into Escherichia coli α2-macroglobulin endopeptidase snap-trap inhibition. 著者: Irene Garcia-Ferrer / Pedro Arêde / Josué Gómez-Blanco / Daniel Luque / Stephane Duquerroy / José R Castón / Theodoros Goulas / F Xavier Gomis-Rüth /   要旨: The survival of commensal bacteria requires them to evade host peptidases. Gram-negative bacteria from the human gut microbiome encode a relative of the human endopeptidase inhibitor, α2- ...The survival of commensal bacteria requires them to evade host peptidases. Gram-negative bacteria from the human gut microbiome encode a relative of the human endopeptidase inhibitor, α2-macroglobulin (α2M). Escherichia coli α2M (ECAM) is a ∼ 180-kDa multidomain membrane-anchored pan-peptidase inhibitor, which is cleaved by host endopeptidases in an accessible bait region. Structural studies by electron microscopy and crystallography reveal that this cleavage causes major structural rearrangement of more than half the 13-domain structure from a native to a compact induced form. It also exposes a reactive thioester bond, which covalently traps the peptidase. Subsequently, peptidase-laden ECAM is shed from the membrane and may dimerize. Trapped peptidases are still active except against very large substrates, so inhibition potentially prevents damage of large cell envelope components, but not host digestion. Mechanistically, these results document a novel monomeric "snap trap." | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5a42.cif.gz 5a42.cif.gz | 280.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5a42.ent.gz pdb5a42.ent.gz | 213 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5a42.json.gz 5a42.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5a42_validation.pdf.gz 5a42_validation.pdf.gz | 694.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5a42_full_validation.pdf.gz 5a42_full_validation.pdf.gz | 709.8 KB | 表示 | |
| XML形式データ |  5a42_validation.xml.gz 5a42_validation.xml.gz | 41.8 KB | 表示 | |
| CIF形式データ |  5a42_validation.cif.gz 5a42_validation.cif.gz | 64.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/a4/5a42 https://data.pdbj.org/pub/pdb/validation_reports/a4/5a42 ftp://data.pdbj.org/pub/pdb/validation_reports/a4/5a42 ftp://data.pdbj.org/pub/pdb/validation_reports/a4/5a42 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 177690.297 Da / 分子数: 1 / 断片: RESIDUES 40-1653 / 由来タイプ: 組換発現 詳細: THE EXPRESSED AND PURIFIED SAMPLE CONTAINED THE SEQUENCE GPM-A40 TO P1653. 由来: (組換発現)   |
|---|---|
| 配列の詳細 | THE PURIFIED SAMPLE CONTAINED FROM A40 TO P1653 |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: NATIVE CONFORMATION OF THE E. COLI ALPHA-2- MACROGLOBULIN (ECAM). タイプ: COMPLEX |
|---|---|
| 緩衝液 | 名称: 10MM TRIS-HCL, 75MM NACL / pH: 7.4 / 詳細: 10MM TRIS-HCL, 75MM NACL |
| 試料 | 濃度: 0.01 mg/ml / 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | 詳細: CARBON |
| 急速凍結 | 装置: LEICA EM CPC / 凍結剤: ETHANE 詳細: VITRIFICATION 1 -- CRYOGEN- ETHANE, INSTRUMENT- LEICA EM CPC, METHOD- BLOTTED FOR 1MIN BEFORE PLUNGING WITH 5UL PURIFIED SAMPLE., |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: FEI TECNAI 20 / 日付: 2014年6月10日 |
|---|---|
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 200 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(補正後): 84269 X / 最大 デフォーカス(公称値): 3500 nm / 最小 デフォーカス(公称値): 1500 nm |
| 撮影 | フィルム・検出器のモデル: FEI EAGLE (4k x 4k) |
| 画像スキャン | デジタル画像の数: 130 |
| 放射波長 | 相対比: 1 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | 詳細: EACH IMAGE | ||||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | ||||||||||||||||
| 3次元再構成 | 手法: PROJECTION MATCHING / 解像度: 16 Å / 粒子像の数: 46842 / ピクセルサイズ(実測値): 4.5 Å 詳細: THE FITTED MODEL IS AN HOMOLOGY MODEL BASED ON A ECAM HOMOLOG FROM SALMONELLA ENTERICA (PDB CODE 4U48) AND THE CRYSTAL STRUCTURES ECAM DOMAINS (PDB CODES 4ZIU,4ZJH,4ZJG,4ZIQ). SUBMISSION ...詳細: THE FITTED MODEL IS AN HOMOLOGY MODEL BASED ON A ECAM HOMOLOG FROM SALMONELLA ENTERICA (PDB CODE 4U48) AND THE CRYSTAL STRUCTURES ECAM DOMAINS (PDB CODES 4ZIU,4ZJH,4ZJG,4ZIQ). SUBMISSION BASED ON EXPERIMENTAL DATA FROM EMDB EMD-3016. (DEPOSITION ID: 13326). 対称性のタイプ: POINT | ||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL / Target criteria: Cross-correlation coefficient / 詳細: METHOD--RIGID BODY | ||||||||||||||||
| 精密化 | 最高解像度: 16 Å | ||||||||||||||||
| 精密化ステップ | サイクル: LAST / 最高解像度: 16 Å
|
 ムービー
ムービー コントローラー
コントローラー

















 PDBj
PDBj






