| 登録情報 | データベース: PDB / ID: 4xhv
|
|---|
| タイトル | Crystal structure of Drosophila Spinophilin-PDZ and a C-terminal peptide of Neurexin |
|---|
 要素 要素 | |
|---|
 キーワード キーワード | SIGNALING PROTEIN / Spinophilin / Neurexin / Praesynaptic density / SYNAPSE |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
vitamin A transport / negative regulation of presynaptic active zone assembly / regulation of neuromuscular synaptic transmission / type I terminal bouton / synaptic assembly at neuromuscular junction / regulation of terminal button organization / neuroligin family protein binding / neurexin family protein binding / apical cortex / positive regulation of synaptic vesicle endocytosis ...vitamin A transport / negative regulation of presynaptic active zone assembly / regulation of neuromuscular synaptic transmission / type I terminal bouton / synaptic assembly at neuromuscular junction / regulation of terminal button organization / neuroligin family protein binding / neurexin family protein binding / apical cortex / positive regulation of synaptic vesicle endocytosis / neuromuscular synaptic transmission / locomotion / regulation of locomotion / associative learning / apolipoprotein binding / synaptic vesicle endocytosis / synapse assembly / actin filament organization / neuromuscular junction / calcium-mediated signaling / synapse organization / neuron projection development / actin filament binding / nervous system development / actin cytoskeleton / actin binding / presynaptic membrane / chemical synaptic transmission / postsynaptic membrane / cell differentiation / cytoskeleton / postsynaptic density / dendrite / synapse / metal ion binding / cytoplasm類似検索 - 分子機能 Neurabin-1/2, PDZ domain / Neurabin-like family / PDZ domain / : / Laminin G domain / Laminin G domain profile. / Laminin G domain / Laminin G domain / SAM domain (Sterile alpha motif) / PDZ domain ...Neurabin-1/2, PDZ domain / Neurabin-like family / PDZ domain / : / Laminin G domain / Laminin G domain profile. / Laminin G domain / Laminin G domain / SAM domain (Sterile alpha motif) / PDZ domain / Pdz3 Domain / EGF-like domain / SAM domain profile. / Sterile alpha motif. / Sterile alpha motif domain / Sterile alpha motif/pointed domain superfamily / Epidermal growth factor-like domain. / EGF-like domain profile. / PDZ domain / EGF-like domain / PDZ domain profile. / Domain present in PSD-95, Dlg, and ZO-1/2. / PDZ domain / PDZ superfamily / Concanavalin A-like lectin/glucanase domain superfamily / Roll / Mainly Beta類似検索 - ドメイン・相同性 LP20995p / Spinophilin, isoform J / Neurexin 1類似検索 - 構成要素 |
|---|
| 生物種 |   Drosophila melanogaster (キイロショウジョウバエ) Drosophila melanogaster (キイロショウジョウバエ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.23 Å 分子置換 / 解像度: 1.23 Å |
|---|
 データ登録者 データ登録者 | Driller, J.H. / Muhammad, K.G.H. / Reddy, S. / Rey, U. / Boehme, M.A. / Hollmann, C. / Ramesh, N. / Depner, H. / Luetzkendorf, J. / Matkovic, T. ...Driller, J.H. / Muhammad, K.G.H. / Reddy, S. / Rey, U. / Boehme, M.A. / Hollmann, C. / Ramesh, N. / Depner, H. / Luetzkendorf, J. / Matkovic, T. / Bergeron, D. / Quentin, C. / Schmoranzer, J. / Goettfert, F. / Holt, M. / Wahl, M.C. / Hell, S.W. / Walter, A. / Sigrist, S.J. / Loll, B. |
|---|
| 資金援助 |  ドイツ, 2件 ドイツ, 2件 | 組織 | 認可番号 | 国 |
|---|
| German Research Foundation | SFB958/A3 |  ドイツ ドイツ | | German Research Foundation | SFB958/A6 |  ドイツ ドイツ |
|
|---|
 引用 引用 |  ジャーナル: Nat Commun / 年: 2015 ジャーナル: Nat Commun / 年: 2015
タイトル: Presynaptic spinophilin tunes neurexin signalling to control active zone architecture and function.
著者: Muhammad, K. / Reddy-Alla, S. / Driller, J.H. / Schreiner, D. / Rey, U. / Bohme, M.A. / Hollmann, C. / Ramesh, N. / Depner, H. / Lutzkendorf, J. / Matkovic, T. / Gotz, T. / Bergeron, D.D. / ...著者: Muhammad, K. / Reddy-Alla, S. / Driller, J.H. / Schreiner, D. / Rey, U. / Bohme, M.A. / Hollmann, C. / Ramesh, N. / Depner, H. / Lutzkendorf, J. / Matkovic, T. / Gotz, T. / Bergeron, D.D. / Schmoranzer, J. / Goettfert, F. / Holt, M. / Wahl, M.C. / Hell, S.W. / Scheiffele, P. / Walter, A.M. / Loll, B. / Sigrist, S.J. |
|---|
| 履歴 | | 登録 | 2015年1月6日 | 登録サイト: RCSB / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2015年7月1日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2015年10月28日 | Group: Database references |
|---|
| 改定 2.0 | 2024年1月10日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Author supporting evidence / Data collection / Database references / Derived calculations / Refinement description
カテゴリ: atom_site / chem_comp_atom ...atom_site / chem_comp_atom / chem_comp_bond / database_2 / pdbx_audit_support / pdbx_initial_refinement_model / pdbx_nonpoly_scheme / pdbx_struct_conn_angle / pdbx_validate_close_contact / struct_conn / struct_site_gen
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.occupancy / _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_audit_support.funding_organization / _pdbx_nonpoly_scheme.auth_seq_num / _pdbx_struct_conn_angle.ptnr1_auth_asym_id / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_symmetry / _pdbx_struct_conn_angle.ptnr3_auth_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _pdbx_validate_close_contact.auth_seq_id_1 / _pdbx_validate_close_contact.auth_seq_id_2 / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_symmetry / _struct_site_gen.auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.23 Å
分子置換 / 解像度: 1.23 Å  データ登録者
データ登録者 ドイツ, 2件
ドイツ, 2件  引用
引用 ジャーナル: Nat Commun / 年: 2015
ジャーナル: Nat Commun / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4xhv.cif.gz
4xhv.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4xhv.ent.gz
pdb4xhv.ent.gz PDB形式
PDB形式 4xhv.json.gz
4xhv.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4xhv_validation.pdf.gz
4xhv_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4xhv_full_validation.pdf.gz
4xhv_full_validation.pdf.gz 4xhv_validation.xml.gz
4xhv_validation.xml.gz 4xhv_validation.cif.gz
4xhv_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/xh/4xhv
https://data.pdbj.org/pub/pdb/validation_reports/xh/4xhv ftp://data.pdbj.org/pub/pdb/validation_reports/xh/4xhv
ftp://data.pdbj.org/pub/pdb/validation_reports/xh/4xhv
 リンク
リンク 集合体
集合体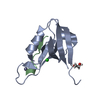
 要素
要素









 X線回折
X線回折 試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  BESSY
BESSY  / ビームライン: 14.2 / 波長: 0.91841 Å
/ ビームライン: 14.2 / 波長: 0.91841 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー






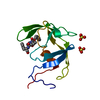






 PDBj
PDBj






