[English] 日本語
 Yorodumi
Yorodumi- PDB-4tvb: Crystal Structure of the Homospermidine Synthase (HSS) from Blast... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 4tvb | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal Structure of the Homospermidine Synthase (HSS) from Blastochloris viridis in Complex with NAD, Putrescine and sym-Homospermidine | ||||||
 Components Components | Homospermidine synthase | ||||||
 Keywords Keywords | TRANSFERASE / homospermidine synthase / oxidoreductase / rossman fold | ||||||
| Function / homology |  Function and homology information Function and homology informationhomospermidine synthase (spermidine-specific) activity / homospermidine synthase / homospermidine synthase activity Similarity search - Function | ||||||
| Biological species |  Blastochloris viridis (bacteria) Blastochloris viridis (bacteria) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.689 Å MOLECULAR REPLACEMENT / Resolution: 1.689 Å | ||||||
 Authors Authors | Krossa, S. | ||||||
 Citation Citation |  Journal: Sci Rep / Year: 2016 Journal: Sci Rep / Year: 2016Title: Comprehensive Structural Characterization of the Bacterial Homospermidine Synthase-an Essential Enzyme of the Polyamine Metabolism. Authors: Krossa, S. / Faust, A. / Ober, D. / Scheidig, A.J. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  4tvb.cif.gz 4tvb.cif.gz | 386.6 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb4tvb.ent.gz pdb4tvb.ent.gz | 313.4 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  4tvb.json.gz 4tvb.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/tv/4tvb https://data.pdbj.org/pub/pdb/validation_reports/tv/4tvb ftp://data.pdbj.org/pub/pdb/validation_reports/tv/4tvb ftp://data.pdbj.org/pub/pdb/validation_reports/tv/4tvb | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  4plpSC  4xq9C  4xqcC  4xqeC  4xqgC  4xr4C  4xrgC S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
| ||||||||
| Details | biological unit is the same as asym. |
- Components
Components
-Protein , 1 types, 2 molecules AB
| #1: Protein | Mass: 52980.441 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Blastochloris viridis (bacteria) / Gene: hss / Plasmid: pETM14 / Production host: Blastochloris viridis (bacteria) / Gene: hss / Plasmid: pETM14 / Production host:  |
|---|
-Non-polymers , 6 types, 1303 molecules 
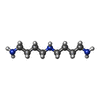
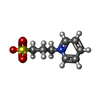

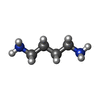


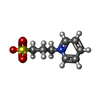



| #2: Chemical | ChemComp-NAI / |
|---|---|
| #3: Chemical | ChemComp-37Z / |
| #4: Chemical | ChemComp-1PS / |
| #5: Chemical | ChemComp-NAD / |
| #6: Chemical | ChemComp-PUT / |
| #7: Water | ChemComp-HOH / |
-Details
| Nonpolymer details | According to the author the molecule 37Z present in the entry depicts the transition state between ...According to the author the molecule 37Z present in the entry depicts the transition state between the oxidised sym-homospermidine and its regular form. The author also suggest that ligand PUT is representing a transition state between 1,4-diaminobutane and non-hydrolysed 4-aminobutanal. |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.4 Å3/Da / Density % sol: 49 % |
|---|---|
| Crystal grow | Temperature: 291 K / Method: vapor diffusion, hanging drop / pH: 4.8 Details: Na-acetate, ammoniumacetate, PEG 10000, NDSB-201, 1,3-diaminopropane PH range: 4.8 |
-Data collection
| Diffraction | Mean temperature: 100 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  PETRA III, EMBL c/o DESY PETRA III, EMBL c/o DESY  / Beamline: P14 (MX2) / Wavelength: 1.23953 Å / Beamline: P14 (MX2) / Wavelength: 1.23953 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: PSI PILATUS 6M / Detector: PIXEL / Date: Sep 6, 2012 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 1.23953 Å / Relative weight: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 1.689→89.717 Å / Num. all: 113442 / Num. obs: 113442 / % possible obs: 98.1 % / Redundancy: 12.8 % / Rpim(I) all: 0.072 / Rrim(I) all: 0.262 / Rsym value: 0.252 / Net I/av σ(I): 2.8 / Net I/σ(I): 8.6 / Num. measured all: 1454896 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell |
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: 4PLP Resolution: 1.689→9.988 Å / Occupancy max: 1 / Occupancy min: 0.17 / SU ML: 0.15 / Cross valid method: FREE R-VALUE / σ(F): 1.33 / Phase error: 21.12 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 56.83 Å2 / Biso mean: 23.1095 Å2 / Biso min: 7.24 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.689→9.988 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 30
|
 Movie
Movie Controller
Controller












 PDBj
PDBj






