| 登録情報 | データベース: PDB / ID: 4qxw
|
|---|
| タイトル | Crystal structure of the human CEACAM1 membrane distal amino terminal (N)-domain |
|---|
 要素 要素 | Carcinoembryonic antigen-related cell adhesion molecule 1 |
|---|
 キーワード キーワード | CELL ADHESION |
|---|
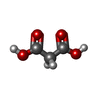
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
regulation of endothelial cell differentiation / insulin receptor internalization / negative regulation of cytotoxic T cell degranulation / granulocyte colony-stimulating factor signaling pathway / regulation of homophilic cell adhesion / regulation of sprouting angiogenesis / regulation of epidermal growth factor receptor signaling pathway / regulation of blood vessel remodeling / negative regulation of hepatocyte proliferation / filamin binding ...regulation of endothelial cell differentiation / insulin receptor internalization / negative regulation of cytotoxic T cell degranulation / granulocyte colony-stimulating factor signaling pathway / regulation of homophilic cell adhesion / regulation of sprouting angiogenesis / regulation of epidermal growth factor receptor signaling pathway / regulation of blood vessel remodeling / negative regulation of hepatocyte proliferation / filamin binding / negative regulation of natural killer cell mediated cytotoxicity directed against tumor cell target / negative regulation of lipid biosynthetic process / bile acid transmembrane transporter activity / negative regulation of T cell mediated cytotoxicity / regulation of endothelial cell migration / negative regulation of granulocyte differentiation / Fibronectin matrix formation / insulin catabolic process / common myeloid progenitor cell proliferation / negative regulation of interleukin-1 production / negative regulation of fatty acid biosynthetic process / positive regulation of vasculogenesis / cell-cell adhesion via plasma-membrane adhesion molecules / negative regulation of platelet aggregation / regulation of immune system process / bile acid and bile salt transport / negative regulation of vascular permeability / wound healing, spreading of cells / transport vesicle membrane / microvillus membrane / negative regulation of T cell receptor signaling pathway / blood vessel development / regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / homophilic cell adhesion via plasma membrane adhesion molecules / tertiary granule membrane / lateral plasma membrane / specific granule membrane / regulation of cell migration / regulation of ERK1 and ERK2 cascade / protein tyrosine kinase binding / basal plasma membrane / integrin-mediated signaling pathway / regulation of cell growth / Cell surface interactions at the vascular wall / adherens junction / negative regulation of protein kinase activity / kinase binding / cellular response to insulin stimulus / cell-cell junction / cell migration / cell junction / actin binding / protein phosphatase binding / angiogenesis / protein dimerization activity / calmodulin binding / cell adhesion / apical plasma membrane / Neutrophil degranulation / cell surface / signal transduction / protein homodimerization activity / extracellular exosome / identical protein binding / membrane / plasma membrane類似検索 - 分子機能 Immunoglobulin domain / Immunoglobulin / Immunoglobulin domain / Immunoglobulin domain / Immunoglobulin subtype 2 / Immunoglobulin C-2 Type / Immunoglobulin V-set domain / Immunoglobulin V-set domain / Immunoglobulin subtype / Immunoglobulin ...Immunoglobulin domain / Immunoglobulin / Immunoglobulin domain / Immunoglobulin domain / Immunoglobulin subtype 2 / Immunoglobulin C-2 Type / Immunoglobulin V-set domain / Immunoglobulin V-set domain / Immunoglobulin subtype / Immunoglobulin / Ig-like domain profile. / Immunoglobulin-like domain / Immunoglobulin-like domain superfamily / Immunoglobulins / Immunoglobulin-like fold / Immunoglobulin-like / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 MALONIC ACID / Carcinoembryonic antigen-related cell adhesion molecule 1類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.04 Å 分子置換 / 解像度: 2.04 Å |
|---|
 データ登録者 データ登録者 | Huang, Y.H. / Gandhi, A.K. / Russell, A. / Kondo, Y. / Chen, Q. / Petsko, G.A. / Blumberg, R.S. |
|---|
 引用 引用 |  ジャーナル: Nature / 年: 2015 ジャーナル: Nature / 年: 2015
タイトル: CEACAM1 regulates TIM-3-mediated tolerance and exhaustion.
著者: Huang, Y.H. / Zhu, C. / Kondo, Y. / Anderson, A.C. / Gandhi, A. / Russell, A. / Dougan, S.K. / Petersen, B.S. / Melum, E. / Pertel, T. / Clayton, K.L. / Raab, M. / Chen, Q. / Beauchemin, N. / ...著者: Huang, Y.H. / Zhu, C. / Kondo, Y. / Anderson, A.C. / Gandhi, A. / Russell, A. / Dougan, S.K. / Petersen, B.S. / Melum, E. / Pertel, T. / Clayton, K.L. / Raab, M. / Chen, Q. / Beauchemin, N. / Yazaki, P.J. / Pyzik, M. / Ostrowski, M.A. / Glickman, J.N. / Rudd, C.E. / Ploegh, H.L. / Franke, A. / Petsko, G.A. / Kuchroo, V.K. / Blumberg, R.S. |
|---|
| 履歴 | | 登録 | 2014年7月22日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2014年11月12日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2015年2月25日 | Group: Database references |
|---|
| 改定 1.2 | 2015年4月29日 | Group: Non-polymer description |
|---|
| 改定 1.3 | 2020年7月29日 | Group: Data collection / Derived calculations / Structure summary
カテゴリ: chem_comp / entity ...chem_comp / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / struct_site / struct_site_gen
Item: _chem_comp.mon_nstd_flag / _chem_comp.name ..._chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.type / _entity.pdbx_description / _pdbx_entity_nonpoly.name
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 1.4 | 2024年2月28日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp / chem_comp_atom ...chem_comp / chem_comp_atom / chem_comp_bond / database_2
Item: _chem_comp.pdbx_synonyms / _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.04 Å
分子置換 / 解像度: 2.04 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nature / 年: 2015
ジャーナル: Nature / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4qxw.cif.gz
4qxw.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4qxw.ent.gz
pdb4qxw.ent.gz PDB形式
PDB形式 4qxw.json.gz
4qxw.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4qxw_validation.pdf.gz
4qxw_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4qxw_full_validation.pdf.gz
4qxw_full_validation.pdf.gz 4qxw_validation.xml.gz
4qxw_validation.xml.gz 4qxw_validation.cif.gz
4qxw_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/qx/4qxw
https://data.pdbj.org/pub/pdb/validation_reports/qx/4qxw ftp://data.pdbj.org/pub/pdb/validation_reports/qx/4qxw
ftp://data.pdbj.org/pub/pdb/validation_reports/qx/4qxw リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: CEACAM1, BGP, BGP1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: CEACAM1, BGP, BGP1 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X6A / 波長: 1.0781 Å
/ ビームライン: X6A / 波長: 1.0781 Å 解析
解析 分子置換 / 解像度: 2.04→47.923 Å / SU ML: 0.21 / σ(F): 1.36 / 位相誤差: 21.68 / 立体化学のターゲット値: Engh & Huber
分子置換 / 解像度: 2.04→47.923 Å / SU ML: 0.21 / σ(F): 1.36 / 位相誤差: 21.68 / 立体化学のターゲット値: Engh & Huber ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj