| Software | | Name | Version | Classification |
|---|
| CrystalClear | | data collection| PHASES | | phasing| CNS | | refinement| PDB_EXTRACT | 3.14 | data extraction| SOLVE | | phasing| d*TREK | | data reduction| d*TREK | | data scaling | | | | | | |
|
|---|
| Refinement | Method to determine structure:  MAD / Resolution: 1.6→20 Å / FOM work R set: 0.7783 / Cross valid method: FREE R-VALUE / σ(F): 0 MAD / Resolution: 1.6→20 Å / FOM work R set: 0.7783 / Cross valid method: FREE R-VALUE / σ(F): 0
| Rfactor | Num. reflection | % reflection |
|---|
| Rfree | 0.2706 | 1430 | 10 % |
|---|
| Rwork | 0.2477 | 12655 | - |
|---|
| obs | - | 14085 | 98.4 % |
|---|
|
|---|
| Solvent computation | Bsol: 57.6509 Å2 |
|---|
| Displacement parameters | Biso max: 66.58 Å2 / Biso mean: 35.855 Å2 / Biso min: 14.27 Å2
| Baniso -1 | Baniso -2 | Baniso -3 |
|---|
| 1- | -1.081 Å2 | 0 Å2 | 3.317 Å2 |
|---|
| 2- | - | -0.598 Å2 | 0 Å2 |
|---|
| 3- | - | - | 1.679 Å2 |
|---|
|
|---|
| Refinement step | Cycle: final / Resolution: 1.6→20 Å
| Protein | Nucleic acid | Ligand | Solvent | Total |
|---|
| Num. atoms | 0 | 970 | 0 | 150 | 1120 |
|---|
| Biso mean | - | - | - | 42.03 | - |
|---|
| Num. residues | - | - | - | - | 46 |
|---|
|
|---|
| Refine LS restraints | | Refine-ID | Type | Dev ideal | Dev ideal target |
|---|
| X-RAY DIFFRACTION | c_bond_d| 0.004 | | | X-RAY DIFFRACTION | c_angle_d| 0.881 | | | X-RAY DIFFRACTION | c_mcbond_it| 0 | 1.5 | | X-RAY DIFFRACTION | c_scbond_it| 1.47 | 2 | | X-RAY DIFFRACTION | c_mcangle_it| 0 | 2 | | X-RAY DIFFRACTION | c_scangle_it| 2.102 | 2.5 | | | | | | |
|
|---|
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 10 | Resolution (Å) | Rfactor Rfree | Num. reflection Rfree | Rfactor Rwork | Num. reflection Rwork | Num. reflection all | % reflection obs (%) |
|---|
| 1.6-1.66 | 0.368 | 141 | 0.3173 | 1265 | 1406 | 100 | | 1.66-1.72 | 0.362 | 131 | 0.3031 | 1296 | 1427 | 100 | | 1.72-1.8 | 0.3377 | 135 | 0.2978 | 1284 | 1419 | 100 | | 1.8-1.9 | 0.3517 | 155 | 0.3025 | 1264 | 1419 | 99.9 | | 1.9-2.02 | 0.3369 | 142 | 0.2892 | 1279 | 1421 | 100 | | 2.02-2.17 | 0.2955 | 129 | 0.2512 | 1304 | 1433 | 100 | | 2.17-2.39 | 0.2925 | 178 | 0.2708 | 1257 | 1435 | 99.9 | | 2.39-2.73 | 0.2879 | 142 | 0.2641 | 1278 | 1420 | 100 | | 2.73-3.44 | 0.2664 | 156 | 0.2432 | 1257 | 1413 | 97.4 | | 3.44-20 | 0.2069 | 121 | 0.2043 | 1171 | 1292 | 87.7 |
|
|---|
| Xplor file | | Refine-ID | Serial no | Param file |
|---|
| X-RAY DIFFRACTION | 1 | dna-rna_free.param | | X-RAY DIFFRACTION | 2 | CNS_TOPPAR:water_rep.param| X-RAY DIFFRACTION | 3 | CNS_TOPPAR:ion.param| X-RAY DIFFRACTION | 4 | bru.param | | |
|
|---|
 Yorodumi
Yorodumi Open data
Open data Basic information
Basic information Components
Components Keywords
Keywords Function and homology information
Function and homology information X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  SYNCHROTRON /
SYNCHROTRON /  MAD / Resolution: 1.6 Å
MAD / Resolution: 1.6 Å  Authors
Authors Japan, 1items
Japan, 1items  Citation
Citation Journal: To Be Published
Journal: To Be Published Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 4p3t.cif.gz
4p3t.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb4p3t.ent.gz
pdb4p3t.ent.gz PDB format
PDB format 4p3t.json.gz
4p3t.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads https://data.pdbj.org/pub/pdb/validation_reports/p3/4p3t
https://data.pdbj.org/pub/pdb/validation_reports/p3/4p3t ftp://data.pdbj.org/pub/pdb/validation_reports/p3/4p3t
ftp://data.pdbj.org/pub/pdb/validation_reports/p3/4p3t Links
Links Assembly
Assembly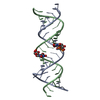
 Components
Components X-RAY DIFFRACTION / Number of used crystals: 1
X-RAY DIFFRACTION / Number of used crystals: 1  Sample preparation
Sample preparation SYNCHROTRON / Site:
SYNCHROTRON / Site:  Photon Factory
Photon Factory  / Beamline: BL-5A / Wavelength: 0.91992, 0.92005, 0.90660, 1.0
/ Beamline: BL-5A / Wavelength: 0.91992, 0.92005, 0.90660, 1.0 MAD
MAD Processing
Processing MAD / Resolution: 1.6→20 Å / FOM work R set: 0.7783 / Cross valid method: FREE R-VALUE / σ(F): 0
MAD / Resolution: 1.6→20 Å / FOM work R set: 0.7783 / Cross valid method: FREE R-VALUE / σ(F): 0  Movie
Movie Controller
Controller






 PDBj
PDBj






























