| 登録情報 | データベース: PDB / ID: 4mny
|
|---|
| タイトル | Crystal structure of urokinase-type plasminogen activator (uPA) complexed with bicyclic peptide UK903 |
|---|
 要素 要素 | - Urokinase-type plasminogen activator chain B
- bicyclic peptide UK903
|
|---|
 キーワード キーワード | HYDROLASE/HYDROLASE INHIBITOR / competitive inhibitor / bicyclic peptide / inhibitor / protease / N / N' / N''-(benzene-1 / 3 / 5-triyl)tris(2-bromoacetamide) (TBAB) cyclization / extracellular / HYDROLASE-HYDROLASE INHIBITOR complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
u-plasminogen activator / regulation of smooth muscle cell-matrix adhesion / urokinase plasminogen activator signaling pathway / regulation of plasminogen activation / regulation of fibrinolysis / regulation of wound healing / protein complex involved in cell-matrix adhesion / regulation of signaling receptor activity / negative regulation of plasminogen activation / regulation of smooth muscle cell migration ...u-plasminogen activator / regulation of smooth muscle cell-matrix adhesion / urokinase plasminogen activator signaling pathway / regulation of plasminogen activation / regulation of fibrinolysis / regulation of wound healing / protein complex involved in cell-matrix adhesion / regulation of signaling receptor activity / negative regulation of plasminogen activation / regulation of smooth muscle cell migration / serine-type endopeptidase complex / Dissolution of Fibrin Clot / smooth muscle cell migration / plasminogen activation / regulation of cell adhesion mediated by integrin / tertiary granule membrane / negative regulation of fibrinolysis / regulation of cell adhesion / specific granule membrane / serine protease inhibitor complex / fibrinolysis / chemotaxis / blood coagulation / regulation of cell population proliferation / response to hypoxia / positive regulation of cell migration / serine-type endopeptidase activity / external side of plasma membrane / focal adhesion / Neutrophil degranulation / cell surface / signal transduction / proteolysis / extracellular space / extracellular exosome / extracellular region / plasma membrane類似検索 - 分子機能 Kringle domain / Kringle / Kringle, conserved site / Kringle superfamily / Kringle domain signature. / Kringle domain profile. / Kringle domain / : / Kringle-like fold / EGF-like domain profile. ...Kringle domain / Kringle / Kringle, conserved site / Kringle superfamily / Kringle domain signature. / Kringle domain profile. / Kringle domain / : / Kringle-like fold / EGF-like domain profile. / EGF-like domain signature 1. / EGF-like domain / Serine proteases, trypsin family, histidine active site / Serine proteases, trypsin family, serine active site / Serine proteases, trypsin family, histidine active site. / Peptidase S1A, chymotrypsin family / Serine proteases, trypsin family, serine active site. / Serine proteases, trypsin domain profile. / Trypsin-like serine protease / Serine proteases, trypsin domain / Trypsin / Trypsin-like serine proteases / Thrombin, subunit H / Peptidase S1, PA clan, chymotrypsin-like fold / Peptidase S1, PA clan / Beta Barrel / Mainly Beta類似検索 - ドメイン・相同性 bicyclic peptide UK903 / Chem-29O / ACETATE ION / Urokinase-type plasminogen activator類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.7 Å 分子置換 / 解像度: 1.7 Å |
|---|
 データ登録者 データ登録者 | Chen, S. / Pojer, F. / Heinis, C. |
|---|
 引用 引用 |  ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2014 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2014
タイトル: Peptide ligands stabilized by small molecules.
著者: Chen, S. / Bertoldo, D. / Angelini, A. / Pojer, F. / Heinis, C. |
|---|
| 履歴 | | 登録 | 2013年9月11日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2014年2月5日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2014年2月26日 | Group: Database references |
|---|
| 改定 1.2 | 2017年11月15日 | Group: Refinement description / カテゴリ: software |
|---|
| 改定 1.3 | 2024年11月6日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / struct_conn / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_value_order / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.7 Å
分子置換 / 解像度: 1.7 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2014
ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4mny.cif.gz
4mny.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4mny.ent.gz
pdb4mny.ent.gz PDB形式
PDB形式 4mny.json.gz
4mny.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4mny_validation.pdf.gz
4mny_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4mny_full_validation.pdf.gz
4mny_full_validation.pdf.gz 4mny_validation.xml.gz
4mny_validation.xml.gz 4mny_validation.cif.gz
4mny_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/mn/4mny
https://data.pdbj.org/pub/pdb/validation_reports/mn/4mny ftp://data.pdbj.org/pub/pdb/validation_reports/mn/4mny
ftp://data.pdbj.org/pub/pdb/validation_reports/mn/4mny リンク
リンク 集合体
集合体
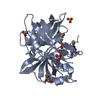

 要素
要素 Homo sapiens (ヒト) / 遺伝子: PLAU / プラスミド: pSecTagA / 細胞株 (発現宿主): HEK 293 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: PLAU / プラスミド: pSecTagA / 細胞株 (発現宿主): HEK 293 / 発現宿主:  Homo sapiens (ヒト) / 参照: UniProt: P00749, u-plasminogen activator
Homo sapiens (ヒト) / 参照: UniProt: P00749, u-plasminogen activator







 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SLS
SLS  / ビームライン: X06DA / 波長: 1 Å
/ ビームライン: X06DA / 波長: 1 Å 解析
解析 分子置換 / 解像度: 1.7→43.24 Å / Cor.coef. Fo:Fc: 0.959 / Cor.coef. Fo:Fc free: 0.935 / SU B: 5.058 / SU ML: 0.075 / 交差検証法: THROUGHOUT / σ(F): 0 / σ(I): 0 / ESU R: 0.173 / ESU R Free: 0.109 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
分子置換 / 解像度: 1.7→43.24 Å / Cor.coef. Fo:Fc: 0.959 / Cor.coef. Fo:Fc free: 0.935 / SU B: 5.058 / SU ML: 0.075 / 交差検証法: THROUGHOUT / σ(F): 0 / σ(I): 0 / ESU R: 0.173 / ESU R Free: 0.109 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj