ソフトウェア 名称 バージョン 分類 データ収集 位相決定 (phenix.refine: 1.7_650)精密化 データ削減 データスケーリング
精密化 構造決定の手法 開始モデル 解像度 / SU ML / Isotropic thermal model / 交差検証法 / σ(F) / 位相誤差 / 立体化学のターゲット値 / 詳細 Rfactor 反射数 %反射 Selection details Rfree 0.2214 2162 5.08 % random Rwork 0.1925 - - - obs 0.194 42577 89.08 % - all - 48257 - -
溶媒の処理 減衰半径 / VDWプローブ半径 / 溶媒モデル / Bsol 2 / ksol 3 原子変位パラメータ Baniso -1 Baniso -2 Baniso -3 1- -1.3797 Å2 0.0644 Å2 0.0992 Å2 2- - 0.34 Å2 -2.7349 Å2 3- - - 1.0398 Å2
精密化ステップ サイクル / 解像度 タンパク質 核酸 リガンド 溶媒 全体 原子数 2686 0 59 742 3487
拘束条件 Refine-ID タイプ Dev ideal 数 X-RAY DIFFRACTION f_bond_d0.009 2794 X-RAY DIFFRACTION f_angle_d1.22 3769 X-RAY DIFFRACTION f_dihedral_angle_d15.066 1059 X-RAY DIFFRACTION f_chiral_restr0.075 431 X-RAY DIFFRACTION f_plane_restr0.004 478
LS精密化 シェル 解像度 (Å)Rfactor Rfree Num. reflection Rfree Rfactor Rwork Num. reflection Rwork Refine-ID % reflection obs (%)1.6-1.6372 0.3149 111 0.2483 2019 X-RAY DIFFRACTION 67 1.6372-1.6782 0.2863 139 0.2505 2530 X-RAY DIFFRACTION 83 1.6782-1.7235 0.2859 140 0.2371 2554 X-RAY DIFFRACTION 86 1.7235-1.7743 0.2784 128 0.2275 2614 X-RAY DIFFRACTION 85 1.7743-1.8315 0.2629 142 0.2253 2616 X-RAY DIFFRACTION 86 1.8315-1.897 0.2382 141 0.2145 2686 X-RAY DIFFRACTION 89 1.897-1.9729 0.2295 145 0.1928 2683 X-RAY DIFFRACTION 89 1.9729-2.0627 0.2461 135 0.1826 2744 X-RAY DIFFRACTION 90 2.0627-2.1715 0.23 148 0.1816 2754 X-RAY DIFFRACTION 92 2.1715-2.3075 0.209 154 0.1725 2806 X-RAY DIFFRACTION 93 2.3075-2.4857 0.2225 174 0.1805 2816 X-RAY DIFFRACTION 94 2.4857-2.7358 0.2162 142 0.1936 2838 X-RAY DIFFRACTION 94 2.7358-3.1316 0.2079 165 0.1898 2875 X-RAY DIFFRACTION 95 3.1316-3.9451 0.1808 156 0.1698 2913 X-RAY DIFFRACTION 96 3.9451-44.5271 0.2002 142 0.1906 2967 X-RAY DIFFRACTION 97
精密化 TLS S33 / 手法 / Refine-ID
大きな表を表示 (24 x 3) 大きな表を隠す ID L11 (°2 )L12 (°2 )L13 (°2 )L22 (°2 )L23 (°2 )L33 (°2 )S11 (Å °)S12 (Å °)S13 (Å °)S21 (Å °)S22 (Å °)S23 (Å °)S31 (Å °)S32 (Å °)T11 (Å2 )T12 (Å2 )T13 (Å2 )T22 (Å2 )T23 (Å2 )T33 (Å2 )Origin x (Å)Origin y (Å)Origin z (Å)1 0.2697 0.2738 0.1008 0.1213 -0.0109 0.0206 -0.0101 0.0327 -0.0189 -0.0228 0.0032 -0.0004 -0.0112 -0.0024 0.0595 0.0055 0.006 0.0615 -0.0011 0.0538 13.0582 43.6881 7.1716 2 0.2956 -0.1016 0.0087 0.0918 0.0222 0.1058 -0.0015 -0.0089 0.0134 0.0223 -0.0046 0.0016 0.0107 -0.0188 0.0596 -0.0013 -0.0023 0.0546 -0.0016 0.0497 -5.4317 12.0375 26.5028 3 0.103 0.0566 -0.0398 0.0766 0.0549 0.0871 -0.0001 0.0007 -0.0067 -0.0075 0.0003 -0.0047 -0.0106 0.0015 0.0466 -0.003 -0.0097 0.0338 0.005 0.0425 4.0926 27.1403 16.5326
精密化 TLSグループ ID Refine-ID Refine TLS-ID Selection details 1 X-RAY DIFFRACTION 1 CHAIN A AND (RESSEQ 7:195)2 X-RAY DIFFRACTION 2 CHAIN B AND (RESSEQ 6:194)3 X-RAY DIFFRACTION 3 CHAIN A AND (RESSEQ 301:769) OR CHAIN B AND (RESSEQ 600:1073)
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Cryphonectria parasitica (菌類)
Cryphonectria parasitica (菌類) X線回折 /
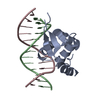
X線回折 /  分子置換 / 解像度: 1.6 Å
分子置換 / 解像度: 1.6 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4ku4.cif.gz
4ku4.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4ku4.ent.gz
pdb4ku4.ent.gz PDB形式
PDB形式 4ku4.json.gz
4ku4.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/ku/4ku4
https://data.pdbj.org/pub/pdb/validation_reports/ku/4ku4 ftp://data.pdbj.org/pub/pdb/validation_reports/ku/4ku4
ftp://data.pdbj.org/pub/pdb/validation_reports/ku/4ku4
 リンク
リンク 集合体
集合体


 要素
要素 Cryphonectria parasitica (菌類) / 発現宿主:
Cryphonectria parasitica (菌類) / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.5418 Å
回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.5418 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj




