+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3u9c | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of a C-terminal deletion mutant of human protein kinase CK2 catalytic subunit with the ATP-competitive inhibitor resorufin | ||||||
 要素 要素 | Casein kinase II subunit alpha | ||||||
 キーワード キーワード | TRANSFERASE/TRANSFERASE INHIBITOR / protein kinase CK2 casein kinase 2 / eukaryotic protein kinase fold / ATP:protein phosphotransferase / protein kinase / ATP CK2beta / TRANSFERASE-TRANSFERASE INHIBITOR complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of chromosome separation / positive regulation of aggrephagy / WNT mediated activation of DVL / Condensation of Prometaphase Chromosomes / protein kinase CK2 complex / symbiont-mediated disruption of host cell PML body / Receptor Mediated Mitophagy / Synthesis of PC / Sin3-type complex / Maturation of hRSV A proteins ...regulation of chromosome separation / positive regulation of aggrephagy / WNT mediated activation of DVL / Condensation of Prometaphase Chromosomes / protein kinase CK2 complex / symbiont-mediated disruption of host cell PML body / Receptor Mediated Mitophagy / Synthesis of PC / Sin3-type complex / Maturation of hRSV A proteins / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / negative regulation of signal transduction by p53 class mediator / negative regulation of apoptotic signaling pathway / positive regulation of Wnt signaling pathway / negative regulation of double-strand break repair via homologous recombination / : / negative regulation of proteasomal ubiquitin-dependent protein catabolic process / Signal transduction by L1 / Hsp90 protein binding / PML body / Regulation of PTEN stability and activity / Wnt signaling pathway / positive regulation of protein catabolic process / KEAP1-NFE2L2 pathway / kinase activity / rhythmic process / Cooperation of PDCL (PhLP1) and TRiC/CCT in G-protein beta folding / double-strand break repair / positive regulation of cell growth / Regulation of TP53 Activity through Phosphorylation / non-specific serine/threonine protein kinase / regulation of cell cycle / negative regulation of translation / protein stabilization / protein serine kinase activity / protein serine/threonine kinase activity / positive regulation of cell population proliferation / apoptotic process / DNA damage response / signal transduction / nucleoplasm / ATP binding / identical protein binding / nucleus / plasma membrane / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.2 Å 分子置換 / 解像度: 3.2 Å | ||||||
 データ登録者 データ登録者 | Klopffleisch, K. / Issinger, O.-G. / Niefind, K. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2012 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2012タイトル: Low-density crystal packing of human protein kinase CK2 catalytic subunit in complex with resorufin or other ligands: a tool to study the unique hinge-region plasticity of the enzyme without packing bias. 著者: Klopffleisch, K. / Issinger, O.G. / Niefind, K. #1: ジャーナル: Biochim.Biophys.Acta / 年: 2010 タイトル: Conformational plasticity of the catalytic subunit of protein kinase CK2 and its consequences for regulation and drug design. 著者: Niefind, K. / Issinger, O.G. #2:  ジャーナル: Chem.Biol. / 年: 2008 ジャーナル: Chem.Biol. / 年: 2008タイトル: The CK2 alpha/CK2 beta interface of human protein kinase CK2 harbors a binding pocket for small molecules. 著者: Raaf, J. / Brunstein, E. / Issinger, O.G. / Niefind, K. #3: ジャーナル: Cell.Mol.Life Sci. / 年: 2009 タイトル: Protein kinase CK2 in health and disease: Protein kinase CK2: from structures to insights. 著者: Niefind, K. / Raaf, J. / Issinger, O.G. #4:  ジャーナル: J.Mol.Biol. / 年: 2005 ジャーナル: J.Mol.Biol. / 年: 2005タイトル: Inclining the purine base binding plane in protein kinase CK2 by exchanging the flanking side-chains generates a preference for ATP as a cosubstrate. 著者: Yde, C.W. / Ermakova, I. / Issinger, O.G. / Niefind, K. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3u9c.cif.gz 3u9c.cif.gz | 294.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3u9c.ent.gz pdb3u9c.ent.gz | 241.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3u9c.json.gz 3u9c.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3u9c_validation.pdf.gz 3u9c_validation.pdf.gz | 472.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3u9c_full_validation.pdf.gz 3u9c_full_validation.pdf.gz | 480.1 KB | 表示 | |
| XML形式データ |  3u9c_validation.xml.gz 3u9c_validation.xml.gz | 26.5 KB | 表示 | |
| CIF形式データ |  3u9c_validation.cif.gz 3u9c_validation.cif.gz | 34.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/u9/3u9c https://data.pdbj.org/pub/pdb/validation_reports/u9/3u9c ftp://data.pdbj.org/pub/pdb/validation_reports/u9/3u9c ftp://data.pdbj.org/pub/pdb/validation_reports/u9/3u9c | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 | 分子量: 40066.742 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: CSNK2A1, CK2A1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: CSNK2A1, CK2A1 / 発現宿主:  参照: UniProt: P68400, non-specific serine/threonine protein kinase |
|---|
-非ポリマー , 5種, 70分子 
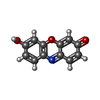







| #2: 化合物 | ChemComp-SO4 / #3: 化合物 | #4: 化合物 | ChemComp-GOL / #5: 化合物 | ChemComp-CL / | #6: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.2 Å3/Da / 溶媒含有率: 61.6 % |
|---|---|
| 結晶化 | 温度: 293 K / pH: 6.5 詳細: reservoir: 30 % polyethylene glycol 8000, 0.2 M ammonium sulfate, 0.1 M sodium cacodylate buffer; drop: 2 microliters preincubated CK2alpha/resorufin mixture (5 mM resorufin, 5 mg/ml Ck2alpha) ...詳細: reservoir: 30 % polyethylene glycol 8000, 0.2 M ammonium sulfate, 0.1 M sodium cacodylate buffer; drop: 2 microliters preincubated CK2alpha/resorufin mixture (5 mM resorufin, 5 mg/ml Ck2alpha), 1 reservoir solution , pH 6.5, VAPOR DIFFUSION, SITTING DROP, temperature 293K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  BESSY BESSY  / ビームライン: 14.1 / 波長: 0.91841 / ビームライン: 14.1 / 波長: 0.91841 |
| 検出器 | タイプ: MAR CCD 165 mm / 検出器: CCD / 日付: 2008年2月15日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.91841 Å / 相対比: 1 |
| 反射 | 解像度: 3.2→38.5 Å / Num. obs: 17653 / % possible obs: 99.6 % / 冗長度: 10.2 % / Rmerge(I) obs: 0.178 / Rsym value: 0.178 / Net I/σ(I): 14.6 |
| 反射 シェル | 解像度: 3.2→3.37 Å / 冗長度: 10.4 % / Rmerge(I) obs: 0.689 / Mean I/σ(I) obs: 3.9 / Rsym value: 0.689 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 3NGA 解像度: 3.2→19.948 Å / SU ML: 0.32 / σ(F): 1.36 / 位相誤差: 22.03 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.86 Å / VDWプローブ半径: 1.1 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL / Bsol: 28.111 Å2 / ksol: 0.343 e/Å3 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3.2→19.948 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー




























 PDBj
PDBj








