+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3tmn | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | THE BINDING OF L-VALYL-L-TRYPTOPHAN TO CRYSTALLINE THERMOLYSIN ILLUSTRATES THE MODE OF INTERACTION OF A PRODUCT OF PEPTIDE HYDROLYSIS | |||||||||
 要素 要素 | THERMOLYSIN | |||||||||
 キーワード キーワード | HYDROLASE/HYDROLASE INHIBITOR / HYDROLASE / METALLOPROTEINASE / HYDROLASE-HYDROLASE INHIBITOR COMPLEX | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報thermolysin / metalloendopeptidase activity / proteolysis / extracellular region / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 |  X線回折 / MOLECULAR SUPSTATUTION / 解像度: 1.7 Å X線回折 / MOLECULAR SUPSTATUTION / 解像度: 1.7 Å | |||||||||
 データ登録者 データ登録者 | Holden, H.M. / Matthews, B.W. | |||||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 1988 ジャーナル: J.Biol.Chem. / 年: 1988タイトル: The binding of L-valyl-L-tryptophan to crystalline thermolysin illustrates the mode of interaction of a product of peptide hydrolysis. 著者: Holden, H.M. / Matthews, B.W. #1:  ジャーナル: Science / 年: 1987 ジャーナル: Science / 年: 1987タイトル: Structures of Two Thermolysin-Inhibitor Complexes that Differ by a Single Hydrogen Bond 著者: Tronrud, D.E. / Holden, H.M. / Matthews, B.W. #2:  ジャーナル: Eur.J.Biochem. / 年: 1986 ジャーナル: Eur.J.Biochem. / 年: 1986タイトル: Crystallographic Structural Analysis of Phosphoramidates as Inhibitors and Transition-State Analogs of Thermolysin 著者: Tronrud, D.E. / Monzingo, A.F. / Matthews, B.W. #3:  ジャーナル: Biochemistry / 年: 1984 ジャーナル: Biochemistry / 年: 1984タイトル: Binding of N-Carboxymethyl Dipepetide Inhibitors to Thermolysin Determined by X-Ray Crystallography. A Novel Class of Transition-State Analogues for Zinc Peptidases 著者: Monzingo, A.F. / Matthews, B.W. #4:  ジャーナル: Biochemistry / 年: 1984 ジャーナル: Biochemistry / 年: 1984タイトル: An Interactive Computer Graphics Study of Thermolysin-Catalyzed Peptide Cleavage and Inhibition by N-Carboxymethyl Dipeptides 著者: Hangauer, D.G. / Monzingo, A.F. / Matthews, B.W. #5:  ジャーナル: Biochemistry / 年: 1983 ジャーナル: Biochemistry / 年: 1983タイトル: Structural Analysis of the Inhibition of Thermolysin by an Active-Site-Directed Irreversible Inhibitor 著者: Holmes, M.A. / Tronrud, D.E. / Matthews, B.W. #6:  ジャーナル: Biochemistry / 年: 1982 ジャーナル: Biochemistry / 年: 1982タイトル: Structure of a Mercaptan-Thermolysin Complex Illustrates Mode of Inhibition of Zinc Proteases by Substrate-Analogue Mercaptans 著者: Monzingo, A.F. / Matthews, B.W. #7:  ジャーナル: J.Mol.Biol. / 年: 1982 ジャーナル: J.Mol.Biol. / 年: 1982タイトル: Structure of Thermolysin Refined at 1.6 Angstroms Resolution 著者: Holmes, M.A. / Matthews, B.W. #8:  ジャーナル: Biochemistry / 年: 1981 ジャーナル: Biochemistry / 年: 1981タイトル: Binding of Hydroxamic Acid Inhibitors to Crystalline Thermolysin Suggests a Pentacoordinate Zinc Intermediate in Catalysis 著者: Holmes, M.A. / Matthews, B.W. #9:  ジャーナル: J.Biol.Chem. / 年: 1979 ジャーナル: J.Biol.Chem. / 年: 1979タイトル: Binding of the Biproduct Analog L-Benzylsuccinic Acid to Thermolysin Determined by X-Ray Crystallography 著者: Bolognesi, M.C. / Matthews, B.W. #10:  ジャーナル: J.Biol.Chem. / 年: 1977 ジャーナル: J.Biol.Chem. / 年: 1977タイトル: Comparison of the Structures of Carboxypeptidase a and Thermolysin 著者: Kester, W.R. / Matthews, B.W. #11:  ジャーナル: J.Mol.Biol. / 年: 1977 ジャーナル: J.Mol.Biol. / 年: 1977タイトル: A Crystallographic Study of the Complex of Phosphoramidon with Thermolysin. A Model for the Presumed Catalytic Transition State and for the Binding of Extended Substrates 著者: Weaver, L.H. / Kester, W.R. / Matthews, B.W. #12:  ジャーナル: Biochemistry / 年: 1977 ジャーナル: Biochemistry / 年: 1977タイトル: Crystallographic Study of the Binding of Dipeptide Inhibitors to Thermolysin. Implications for the Mechanism of Catalysis 著者: Kester, W.R. / Matthews, B.W. #13:  ジャーナル: Biochemistry / 年: 1976 ジャーナル: Biochemistry / 年: 1976タイトル: Role of Calcium in the Thermal Stability of Thermolysin 著者: Dahlquist, F.W. / Long, J.W. / Bigbee, W.L. #14:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1975 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1975タイトル: Evidence of Homologous Relationship between Thermolysin and Neutral Protease a of Bacillus Subtilis 著者: Levy, P.L. / Pangburn, M.K. / Burstein, Y. / Ericsson, L.H. / Neurath, H. / Walsh, K.A. #15:  ジャーナル: Experientia,Suppl. / 年: 1976 ジャーナル: Experientia,Suppl. / 年: 1976タイトル: The Structure and Stability of Thermolysin 著者: Weaver, L.H. / Kester, W.R. / Teneyck, L.F. / Matthews, B.W. #16:  ジャーナル: J.Biol.Chem. / 年: 1974 ジャーナル: J.Biol.Chem. / 年: 1974タイトル: The Conformation of Thermolysin 著者: Matthews, B.W. / Weaver, L.H. / Kester, W.R. #17:  ジャーナル: Biochemistry / 年: 1974 ジャーナル: Biochemistry / 年: 1974タイトル: Binding of Lanthanide Ions to Thermolysin 著者: Matthews, B.W. / Weaver, L.H. #18:  ジャーナル: J.Mol.Biol. / 年: 1972 ジャーナル: J.Mol.Biol. / 年: 1972タイトル: The Structure of Thermolysin. An Electron Density Map at 2.3 Angstroms Resolution 著者: Colman, P.M. / Jansonius, J.N. / Matthews, B.W. #19:  ジャーナル: Nature New Biol. / 年: 1972 ジャーナル: Nature New Biol. / 年: 1972タイトル: Amino-Acid Sequence of Thermolysin 著者: Titani, K. / Hermodson, M.A. / Ericsson, L.H. / Walsh, K.A. / Neurath, H. #20:  ジャーナル: Nature New Biol. / 年: 1972 ジャーナル: Nature New Biol. / 年: 1972タイトル: Three Dimensional Structure of Thermolysin 著者: Matthews, B.W. / Jansonius, J.N. / Colman, P.M. / Schoenborn, B.P. / Duporque, D. #21:  ジャーナル: Nature New Biol. / 年: 1972 ジャーナル: Nature New Biol. / 年: 1972タイトル: Structure of Thermolysin 著者: Matthews, B.W. / Colman, P.M. / Jansonius, J.N. / Titani, K. / Walsh, K.A. / Neurath, H. #22:  ジャーナル: Macromolecules / 年: 1972 ジャーナル: Macromolecules / 年: 1972タイトル: The Gamma Turn. Evidence for a New Folded Conformation in Proteins 著者: Matthews, B.W. #23:  ジャーナル: Biochem.Biophys.Res.Commun. / 年: 1972 ジャーナル: Biochem.Biophys.Res.Commun. / 年: 1972タイトル: Rare Earths as Isomorphous Calcium Replacements for Protein Crystallography 著者: Colman, P.M. / Weaver, L.H. / Matthews, B.W. | |||||||||
| 履歴 |
| |||||||||
| Remark 700 | SHEET THE *ACTIVE-SITE* SHEET SUBSTRUCTURE OF THIS MOLECULE HAS ONE EDGE-STRAND COMPRISED OF TWO ...SHEET THE *ACTIVE-SITE* SHEET SUBSTRUCTURE OF THIS MOLECULE HAS ONE EDGE-STRAND COMPRISED OF TWO DISTINCT SEQUENCES OF THE POLYPEPTIDE CHAIN. TO REPRESENT THIS FEATURE AN EXTRA SHEET IS DEFINED. STRANDS 2,3,4,5 OF S1 ARE IDENTICAL TO STRANDS STRANDS 2,3,4,5 OF S2. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3tmn.cif.gz 3tmn.cif.gz | 82 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3tmn.ent.gz pdb3tmn.ent.gz | 61 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3tmn.json.gz 3tmn.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3tmn_validation.pdf.gz 3tmn_validation.pdf.gz | 456.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3tmn_full_validation.pdf.gz 3tmn_full_validation.pdf.gz | 466.3 KB | 表示 | |
| XML形式データ |  3tmn_validation.xml.gz 3tmn_validation.xml.gz | 16.2 KB | 表示 | |
| CIF形式データ |  3tmn_validation.cif.gz 3tmn_validation.cif.gz | 23.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/tm/3tmn https://data.pdbj.org/pub/pdb/validation_reports/tm/3tmn ftp://data.pdbj.org/pub/pdb/validation_reports/tm/3tmn ftp://data.pdbj.org/pub/pdb/validation_reports/tm/3tmn | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Atom site foot note | 1: RESIDUE 51 IS A CIS-PROLINE. / 2: ATOMS 2616 - 2618 LIE IN SUBSITE S1(PRIME). / 3: ATOMS 2623 - 2632 LIE IN SUBSITE S2(PRIME). |
- 要素
要素
-タンパク質 , 1種, 1分子 E
| #1: タンパク質 | 分子量: 34362.305 Da / 分子数: 1 / 由来タイプ: 天然 由来: (天然)  参照: UniProt: P00800, thermolysin |
|---|
-非ポリマー , 5種, 180分子 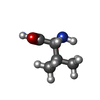







| #2: 化合物 | ChemComp-VAL / | ||||
|---|---|---|---|---|---|
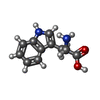
| #3: 化合物 | ChemComp-TRP / | ||||
| #4: 化合物 | ChemComp-CA / #5: 化合物 | ChemComp-ZN / | #6: 水 | ChemComp-HOH / | |
-詳細
| 構成要素の詳細 | THE ACTIVE SITE CLEFT OF THERMOLYSIN CONTAINS AT LEAST FOUR SUBSITES S1, S1(PRIME), S2, AND ...THE ACTIVE SITE CLEFT OF THERMOLYSI |
|---|---|
| 非ポリマーの詳細 | THE DIPEPTIDE VAL-TRP IS PRODUCT OF MERCAPTOAC |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.42 Å3/Da / 溶媒含有率: 49.21 % | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 7.2 / 詳細: pH 7.2 | ||||||||||||||||||||
| 結晶化 | *PLUS 手法: unknown | ||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 285 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: ELLIOTT GX-21 / 波長: 1.54 回転陽極 / タイプ: ELLIOTT GX-21 / 波長: 1.54 |
| 検出器 | タイプ: KODAK / 検出器: FILM / 日付: 1988 |
| 放射 | モノクロメーター: Graphite monochromator / プロトコル: SINGLE CRYSTAL / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.54 Å / 相対比: 1 |
| 反射 | 解像度: 1.7→29.8 Å / Num. obs: 28779 / Observed criterion σ(I): 0 / Rmerge(I) obs: 0.049 / Rsym value: 0.031 |
| 反射 | *PLUS 最高解像度: 1.7 Å / Num. obs: 28779 / Num. measured all: 45350 / Rmerge(I) obs: 0.049 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法: MOLECULAR SUPSTATUTION / 解像度: 1.7→10 Å / 交差検証法: NONE / σ(F): 0 / 立体化学のターゲット値: TNT PROTGEO VERSION 1.0 /
| ||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: NONE | ||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.7→10 Å
| ||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Num. reflection obs: 28604 | ||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー























 PDBj
PDBj





