ソフトウェア 名称 バージョン 分類 データ収集 位相決定 5.2.0019精密化 データ削減 データスケーリング
精密化 構造決定の手法 / 解像度 / Cor.coef. Fo :Fc / Cor.coef. Fo :Fc free / SU ML / 交差検証法 / ESU R Free / 立体化学のターゲット値 / 詳細 Rfactor 反射数 %反射 Selection details Rfree 0.28134 1678 10.1 % RANDOM Rwork 0.25338 - - - obs 0.25616 14911 93.14 % - all - 16009 - -
溶媒の処理 イオンプローブ半径 / 減衰半径 / VDWプローブ半径 / 溶媒モデル 精密化ステップ サイクル / 解像度 タンパク質 核酸 リガンド 溶媒 全体 原子数 0 2903 90 111 3104
拘束条件 大きな表を表示 (5 x 33) 大きな表を隠す Refine-ID タイプ Dev ideal Dev ideal target 数 X-RAY DIFFRACTION r_bond_refined_d0.006 0.021 3324 X-RAY DIFFRACTION r_bond_other_dX-RAY DIFFRACTION r_angle_refined_deg0.983 3 5167 X-RAY DIFFRACTION r_angle_other_degX-RAY DIFFRACTION r_dihedral_angle_1_degX-RAY DIFFRACTION r_dihedral_angle_2_degX-RAY DIFFRACTION r_dihedral_angle_3_degX-RAY DIFFRACTION r_dihedral_angle_4_degX-RAY DIFFRACTION r_chiral_restrX-RAY DIFFRACTION r_gen_planes_refined0.003 0.02 1432 X-RAY DIFFRACTION r_gen_planes_otherX-RAY DIFFRACTION r_nbd_refined0.141 0.2 1167 X-RAY DIFFRACTION r_nbd_otherX-RAY DIFFRACTION r_nbtor_refined0.267 0.2 2012 X-RAY DIFFRACTION r_nbtor_otherX-RAY DIFFRACTION r_xyhbond_nbd_refined0.136 0.2 148 X-RAY DIFFRACTION r_xyhbond_nbd_otherX-RAY DIFFRACTION r_metal_ion_refinedX-RAY DIFFRACTION r_metal_ion_otherX-RAY DIFFRACTION r_symmetry_vdw_refined0.164 0.2 53 X-RAY DIFFRACTION r_symmetry_vdw_otherX-RAY DIFFRACTION r_symmetry_hbond_refined0.195 0.2 7 X-RAY DIFFRACTION r_symmetry_hbond_otherX-RAY DIFFRACTION r_symmetry_metal_ion_refinedX-RAY DIFFRACTION r_symmetry_metal_ion_otherX-RAY DIFFRACTION r_mcbond_itX-RAY DIFFRACTION r_mcbond_otherX-RAY DIFFRACTION r_mcangle_itX-RAY DIFFRACTION r_scbond_itX-RAY DIFFRACTION r_scangle_itX-RAY DIFFRACTION r_rigid_bond_restrX-RAY DIFFRACTION r_sphericity_freeX-RAY DIFFRACTION r_sphericity_bonded
LS精密化 シェル 解像度 / Total num. of bins used Rfactor 反射数 %反射 Rfree 0.415 121 - Rwork 0.377 1021 - obs - - 89.08 %
精密化 TLS 手法 / Refine-ID
大きな表を表示 (25 x 6) 大きな表を隠す ID L11 (°2 )L12 (°2 )L13 (°2 )L22 (°2 )L23 (°2 )L33 (°2 )S11 (Å °)S12 (Å °)S13 (Å °)S21 (Å °)S22 (Å °)S23 (Å °)S31 (Å °)S32 (Å °)S33 (Å °)T11 (Å2 )T12 (Å2 )T13 (Å2 )T22 (Å2 )T23 (Å2 )T33 (Å2 )Origin x (Å)Origin y (Å)Origin z (Å)1 2.8795 0.6804 -0.1266 6.0832 3.3841 1.9736 0.3311 0.655 -0.207 -0.0445 0.2055 -0.1763 0.0948 0.2804 -0.5366 -0.0479 0.0393 -0.0655 -0.2088 -0.0652 -0.0433 -1.685 0.82 -71.03 2 1.2661 -0.386 -1.8066 0.1322 0.3522 5.2996 -0.1173 0.006 0.0303 -0.0147 0.1218 0.0139 0.225 -0.3219 -0.0044 -0.1284 -0.0832 0.0051 -0.3713 0.031 -0.0507 -9.351 3.697 -42.756 3 2.2288 -0.7278 0.0303 1.2453 0.7949 2.4269 -0.074 -0.3104 -0.1283 -0.2036 0.3427 0.0818 -0.1635 0.5471 -0.2687 -0.1179 -0.0589 0.0143 -0.1607 0.0293 -0.1033 4.583 -0.665 -42.461 4 3.9118 0.7056 3.411 5.2935 5.3826 18.5109 0.8536 0.6486 0.2399 -0.1163 0.1144 -0.3147 2.3736 -0.5355 -0.968 0.4151 -0.044 -0.0569 0.2419 0.0544 -0.1289 -0.911 0.039 24.524 5 2.8171 0.6493 2.8087 2.379 -0.402 9.7111 0.3411 -0.5201 0.0722 0.6086 -0.0426 -0.0383 0.5862 -0.6287 -0.2985 0.0868 -0.0636 0.0237 -0.0408 -0.0097 -0.0601 -9.026 -1.746 -3.566 6 5.5962 -0.0382 -0.723 1.4716 -0.2654 0.1431 0.4406 -0.6005 -0.0438 0.6609 0.038 0.0264 -0.1261 0.7141 -0.4786 0.2538 -0.0235 0.0964 0.0797 0.0139 -0.0564 4.683 3.197 -3.514
精密化 TLSグループ ID Refine-ID Refine TLS-ID Auth asym-ID Auth seq-ID 1 X-RAY DIFFRACTION 1 A22 - 30 2 X-RAY DIFFRACTION 1 A81 - 89 3 X-RAY DIFFRACTION 2 A120 4 X-RAY DIFFRACTION 2 A31 - 60 5 X-RAY DIFFRACTION 3 A61 - 80 6 X-RAY DIFFRACTION 4 B22 - 30 7 X-RAY DIFFRACTION 4 B81 - 89 8 X-RAY DIFFRACTION 5 B120 9 X-RAY DIFFRACTION 5 B31 - 60 10 X-RAY DIFFRACTION 6 B61 - 80
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 X線回折 /
X線回折 /  シンクロトロン /
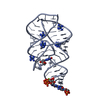
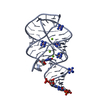
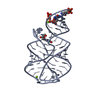
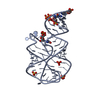
シンクロトロン /  分子置換 / 解像度: 2.3 Å
分子置換 / 解像度: 2.3 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Nat.Chem.Biol. / 年: 2011
ジャーナル: Nat.Chem.Biol. / 年: 2011 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3ski.cif.gz
3ski.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3ski.ent.gz
pdb3ski.ent.gz PDB形式
PDB形式 3ski.json.gz
3ski.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3ski_validation.pdf.gz
3ski_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3ski_full_validation.pdf.gz
3ski_full_validation.pdf.gz 3ski_validation.xml.gz
3ski_validation.xml.gz 3ski_validation.cif.gz
3ski_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/sk/3ski
https://data.pdbj.org/pub/pdb/validation_reports/sk/3ski ftp://data.pdbj.org/pub/pdb/validation_reports/sk/3ski
ftp://data.pdbj.org/pub/pdb/validation_reports/sk/3ski リンク
リンク 集合体
集合体
 要素
要素








 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X29A / 波長: 1.080901 Å
/ ビームライン: X29A / 波長: 1.080901 Å 解析
解析 分子置換 / 解像度: 2.3→20 Å / Cor.coef. Fo:Fc: 0.932 / Cor.coef. Fo:Fc free: 0.919 / SU ML: 0.208 / 交差検証法: THROUGHOUT / ESU R Free: 0.314 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
分子置換 / 解像度: 2.3→20 Å / Cor.coef. Fo:Fc: 0.932 / Cor.coef. Fo:Fc free: 0.919 / SU ML: 0.208 / 交差検証法: THROUGHOUT / ESU R Free: 0.314 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj

































