+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3k35 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal Structure of Human SIRT6 | ||||||
 要素 要素 | NAD-dependent deacetylase sirtuin-6 | ||||||
 キーワード キーワード | HYDROLASE / Rossmann fold / Zn-binding domain / Structural Genomics / Structural Genomics Consortium / SGC / ADP-ribosylation / Metal-binding / NAD / NADP / Nucleus / Phosphoprotein | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報histone H3K56 deacetylase activity, NAD-dependent / ketone biosynthetic process / histone H3K18 deacetylase activity, NAD-dependent / histone H3K9 deacetylase activity, hydrolytic mechanism / histone H3K9 deacetylase activity, NAD-dependent / protein delipidation / NAD+-protein-lysine ADP-ribosyltransferase activity / regulation of lipid catabolic process / chromosome, subtelomeric region / positive regulation of protein localization to chromatin ...histone H3K56 deacetylase activity, NAD-dependent / ketone biosynthetic process / histone H3K18 deacetylase activity, NAD-dependent / histone H3K9 deacetylase activity, hydrolytic mechanism / histone H3K9 deacetylase activity, NAD-dependent / protein delipidation / NAD+-protein-lysine ADP-ribosyltransferase activity / regulation of lipid catabolic process / chromosome, subtelomeric region / positive regulation of protein localization to chromatin / NAD-dependent protein demyristoylase activity / NAD-dependent protein depalmitoylase activity / NAD+-protein-arginine ADP-ribosyltransferase activity / DNA damage sensor activity / positive regulation of stem cell differentiation / negative regulation of D-glucose import / NAD-dependent protein lysine deacetylase activity / positive regulation of chondrocyte proliferation / transposable element silencing / protein acetyllysine N-acetyltransferase / cardiac muscle cell differentiation / pericentric heterochromatin formation / protein deacetylation / histone deacetylase activity, NAD-dependent / protein localization to site of double-strand break / positive regulation of blood vessel branching / negative regulation of glycolytic process / negative regulation of protein localization to chromatin / TORC2 complex binding / positive regulation of vascular endothelial cell proliferation / histone deacetylase regulator activity / regulation of double-strand break repair via homologous recombination / positive regulation of double-strand break repair / negative regulation of protein import into nucleus / regulation of protein secretion / lncRNA binding / DNA repair-dependent chromatin remodeling / negative regulation of gene expression, epigenetic / positive regulation of stem cell proliferation / positive regulation of stem cell population maintenance / NAD+-protein mono-ADP-ribosyltransferase activity / positive regulation of telomere maintenance / pericentric heterochromatin / site of DNA damage / regulation of lipid metabolic process / NAD+ poly-ADP-ribosyltransferase activity / negative regulation of transcription elongation by RNA polymerase II / negative regulation of cellular senescence / 転移酵素; グリコシル基を移すもの; 五炭糖残基を移すもの / NAD+ binding / positive regulation of fat cell differentiation / negative regulation of gluconeogenesis / subtelomeric heterochromatin formation / regulation of protein localization to plasma membrane / response to UV / nucleosome binding / enzyme regulator activity / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / nucleotidyltransferase activity / positive regulation of protein export from nucleus / determination of adult lifespan / chromatin DNA binding / circadian regulation of gene expression / regulation of circadian rhythm / protein destabilization / base-excision repair / positive regulation of insulin secretion / Pre-NOTCH Transcription and Translation / positive regulation of fibroblast proliferation / protein import into nucleus / transcription corepressor activity / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / glucose homeostasis / site of double-strand break / double-strand break repair / Processing of DNA double-strand break ends / positive regulation of cold-induced thermogenesis / damaged DNA binding / chromatin remodeling / negative regulation of cell population proliferation / intracellular membrane-bounded organelle / chromatin binding / chromatin / endoplasmic reticulum / negative regulation of transcription by RNA polymerase II / protein homodimerization activity / DNA binding / zinc ion binding / nucleoplasm / nucleus 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2 Å 分子置換 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Pan, P.W. / Dong, A. / Qiu, W. / Loppnau, P. / Wang, J. / Ravichandran, M. / Bochkarev, A. / Bountra, C. / Weigelt, J. / Arrowsmith, C.H. ...Pan, P.W. / Dong, A. / Qiu, W. / Loppnau, P. / Wang, J. / Ravichandran, M. / Bochkarev, A. / Bountra, C. / Weigelt, J. / Arrowsmith, C.H. / Min, J. / Edwards, A.M. / Structural Genomics Consortium (SGC) | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2011 ジャーナル: J.Biol.Chem. / 年: 2011タイトル: Structure and biochemical functions of SIRT6. 著者: Pan, P.W. / Feldman, J.L. / Devries, M.K. / Dong, A. / Edwards, A.M. / Denu, J.M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3k35.cif.gz 3k35.cif.gz | 355.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3k35.ent.gz pdb3k35.ent.gz | 283.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3k35.json.gz 3k35.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3k35_validation.pdf.gz 3k35_validation.pdf.gz | 1.9 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3k35_full_validation.pdf.gz 3k35_full_validation.pdf.gz | 2 MB | 表示 | |
| XML形式データ |  3k35_validation.xml.gz 3k35_validation.xml.gz | 71 KB | 表示 | |
| CIF形式データ |  3k35_validation.cif.gz 3k35_validation.cif.gz | 97.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/k3/3k35 https://data.pdbj.org/pub/pdb/validation_reports/k3/3k35 ftp://data.pdbj.org/pub/pdb/validation_reports/k3/3k35 ftp://data.pdbj.org/pub/pdb/validation_reports/k3/3k35 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 5 | 
| ||||||||
| 6 | 
| ||||||||
| 7 | 
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | monomer |
- 要素
要素
-タンパク質 , 1種, 6分子 ABCDEF
| #1: タンパク質 | 分子量: 35147.238 Da / 分子数: 6 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: SIR2L6, SIRT6 / プラスミド: pET28-LIC / 発現宿主: Homo sapiens (ヒト) / 遺伝子: SIR2L6, SIRT6 / プラスミド: pET28-LIC / 発現宿主:  参照: UniProt: Q8N6T7, 加水分解酵素; ペプチド以外のCN結合加水分解酵素; 鎖状アミドに作用 |
|---|
-非ポリマー , 5種, 1004分子 
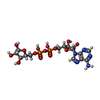







| #2: 化合物 | ChemComp-ZN / #3: 化合物 | ChemComp-APR / #4: 化合物 | ChemComp-SO4 / #5: 化合物 | ChemComp-UNX / #6: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.25 Å3/Da / 溶媒含有率: 45.23 % |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 5.8 詳細: 2M NH4SO4, 2%PEG400, 0.1M Bis-Tris pH 5.8, VAPOR DIFFUSION, HANGING DROP, temperature 293.0K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT / 波長: 1.54 Å 回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT / 波長: 1.54 Å |
| 検出器 | タイプ: RIGAKU RAXIS / 検出器: IMAGE PLATE / 日付: 2009年7月30日 |
| 放射 | モノクロメーター: Si / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.54 Å / 相対比: 1 |
| 反射 | 解像度: 2→100 Å / Num. all: 120514 / Num. obs: 120514 / % possible obs: 97.1 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 3.7 % / Biso Wilson estimate: 30 Å2 / Rmerge(I) obs: 0.086 / Rsym value: 0.086 / Net I/σ(I): 9.5 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1YC5 解像度: 2→19.95 Å / Cor.coef. Fo:Fc: 0.949 / Cor.coef. Fo:Fc free: 0.913 / SU B: 4.661 / SU ML: 0.132 / 交差検証法: THROUGHOUT / σ(F): 2 / σ(I): 0 / ESU R: 0.19 / ESU R Free: 0.184 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 30.84 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→19.95 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.002→2.053 Å / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj





