| 登録情報 | データベース: PDB / ID: 3gv4
|
|---|
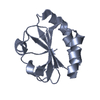
| タイトル | Crystal structure of human HDAC6 zinc finger domain and ubiquitin C-terminal peptide RLRGG |
|---|
 要素 要素 | - Histone deacetylase 6
- ubiquitin C-terminal peptide RLRGG
|
|---|
 キーワード キーワード | HYDROLASE / HDAC6 / zinc finger / ubiquitin C-terminal peptide RLRGG / SGC / Actin-binding / Chromatin regulator / Cytoplasm / Metal-binding / Nucleus / Phosphoprotein / Polymorphism / Repressor / Transcription / Transcription regulation / Ubl conjugation / Zinc / Zinc-finger / Structural Genomics / Structural Genomics Consortium |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
negative regulation of hydrogen peroxide metabolic process / cellular response to topologically incorrect protein / polyubiquitinated misfolded protein transport / positive regulation of cellular response to oxidative stress / negative regulation of aggrephagy / positive regulation of cholangiocyte proliferation / response to misfolded protein / positive regulation of protein oligomerization / negative regulation of axon extension involved in axon guidance / type 2 mitophagy ...negative regulation of hydrogen peroxide metabolic process / cellular response to topologically incorrect protein / polyubiquitinated misfolded protein transport / positive regulation of cellular response to oxidative stress / negative regulation of aggrephagy / positive regulation of cholangiocyte proliferation / response to misfolded protein / positive regulation of protein oligomerization / negative regulation of axon extension involved in axon guidance / type 2 mitophagy / positive regulation of RIG-I signaling pathway / negative regulation of protein-containing complex disassembly / peroxidase inhibitor activity / erythrocyte enucleation / regulation of autophagy of mitochondrion / tubulin deacetylation / Cilium Assembly / regulation of microtubule-based movement / regulation of establishment of protein localization / protein-containing complex disassembly / collateral sprouting / tubulin deacetylase activity / Transcriptional regulation by RUNX2 / lysosome localization / negative regulation of microtubule depolymerization / positive regulation of dendrite morphogenesis / cilium disassembly / ATPase inhibitor activity / histone deacetylase activity, hydrolytic mechanism / misfolded protein binding / ubiquitin-dependent protein catabolic process via the multivesicular body sorting pathway / positive regulation of type 2 mitophagy / dendritic spine morphogenesis / protein deacetylation / aggresome assembly / regulation of androgen receptor signaling pathway / regulation of mitochondrion organization / cellular response to misfolded protein / 転移酵素; アシル基を移すもの; アミノアシル基を移すもの / regulation of fat cell differentiation / protein lysine deacetylase activity / 加水分解酵素; ペプチド以外のCN結合加水分解酵素; 鎖状アミドに作用 / aggresome / microtubule associated complex / cellular response to parathyroid hormone stimulus / response to corticosterone / histone deacetylase activity / positive regulation of intracellular estrogen receptor signaling pathway / response to dexamethasone / Notch-HLH transcription pathway / negative regulation of gene expression, epigenetic / axonal transport of mitochondrion / dynein complex binding / RUNX2 regulates osteoblast differentiation / cell leading edge / response to immobilization stress / protein quality control for misfolded or incompletely synthesized proteins / histone deacetylase complex / positive regulation of epithelial cell migration / cilium assembly / polyubiquitin modification-dependent protein binding / alpha-tubulin binding / regulation of macroautophagy / HSF1 activation / beta-tubulin binding / positive regulation of synaptic transmission, glutamatergic / negative regulation of protein-containing complex assembly / inclusion body / multivesicular body / negative regulation of proteolysis / antiviral innate immune response / axon cytoplasm / actin filament organization / response to amphetamine / epigenetic regulation of gene expression / ubiquitin binding / transcription corepressor binding / Late endosomal microautophagy / intracellular protein transport / Hsp90 protein binding / NOTCH1 Intracellular Domain Regulates Transcription / Constitutive Signaling by NOTCH1 PEST Domain Mutants / Constitutive Signaling by NOTCH1 HD+PEST Domain Mutants / protein destabilization / beta-catenin binding / regulation of protein stability / Chaperone Mediated Autophagy / caveola / tau protein binding / Aggrephagy / histone deacetylase binding / cellular response to hydrogen peroxide / epidermal growth factor receptor signaling pathway / protein polyubiquitination / actin binding / cellular response to heat / perikaryon / microtubule binding / microtubule / regulation of autophagy類似検索 - 分子機能 Ubiquitin Carboxyl-terminal Hydrolase-like zinc finger / Zinc finger, UBP-type / Zn-finger in ubiquitin-hydrolases and other protein / Zinc finger UBP-type profile. / : / Histone deacetylase family / Histone deacetylase domain / Histone deacetylase domain superfamily / Histone deacetylase domain / Zinc/RING finger domain, C3HC4 (zinc finger) ...Ubiquitin Carboxyl-terminal Hydrolase-like zinc finger / Zinc finger, UBP-type / Zn-finger in ubiquitin-hydrolases and other protein / Zinc finger UBP-type profile. / : / Histone deacetylase family / Histone deacetylase domain / Histone deacetylase domain superfamily / Histone deacetylase domain / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 / Ureohydrolase domain superfamily / Zinc finger, RING/FYVE/PHD-type / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.72 Å 分子置換 / 解像度: 1.72 Å |
|---|
 データ登録者 データ登録者 | Dong, A. / Ravichandran, M. / Loppnau, P. / Li, Y. / MacKenzie, F. / Kozieradzki, I. / Edwards, A.M. / Arrowsmith, C.H. / Weigelt, J. / Bountra, C. ...Dong, A. / Ravichandran, M. / Loppnau, P. / Li, Y. / MacKenzie, F. / Kozieradzki, I. / Edwards, A.M. / Arrowsmith, C.H. / Weigelt, J. / Bountra, C. / Bochkarev, A. / Dhe-Paganon, S. / Min, J. / Ouyang, H. / Structural Genomics Consortium (SGC) |
|---|
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Published
タイトル: Crystal structure of human HDAC6 zinc finger domain and ubiquitin C-terminal peptide RLRGG
著者: Dong, A. / Ravichandran, M. / Loppnau, P. / Li, Y. / MacKenzie, F. / Kozieradzki, I. / Edwards, A.M. / Arrowsmith, C.H. / Weigelt, J. / Bountra, C. / Bochkarev, A. / Dhe-Paganon, S. / Min, J. / Ouyang, H. |
|---|
| 履歴 | | 登録 | 2009年3月30日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2009年4月28日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2017年11月1日 | Group: Refinement description / カテゴリ: software / Item: _software.name |
|---|
| 改定 1.3 | 2023年9月6日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  分子置換 / 解像度: 1.72 Å
分子置換 / 解像度: 1.72 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3gv4.cif.gz
3gv4.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3gv4.ent.gz
pdb3gv4.ent.gz PDB形式
PDB形式 3gv4.json.gz
3gv4.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3gv4_validation.pdf.gz
3gv4_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3gv4_full_validation.pdf.gz
3gv4_full_validation.pdf.gz 3gv4_validation.xml.gz
3gv4_validation.xml.gz 3gv4_validation.cif.gz
3gv4_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/gv/3gv4
https://data.pdbj.org/pub/pdb/validation_reports/gv/3gv4 ftp://data.pdbj.org/pub/pdb/validation_reports/gv/3gv4
ftp://data.pdbj.org/pub/pdb/validation_reports/gv/3gv4
 リンク
リンク 集合体
集合体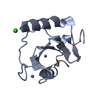
 要素
要素 Homo sapiens (ヒト) / 遺伝子: HDAC6, KIAA0901, JM21 / プラスミド: P28A-LIC / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: HDAC6, KIAA0901, JM21 / プラスミド: P28A-LIC / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT / 波長: 1.54 Å
回転陽極 / タイプ: RIGAKU FR-E SUPERBRIGHT / 波長: 1.54 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj










