+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3e6p | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of human meizothrombin desF1 | |||||||||
 要素 要素 | (Prothrombin) x 2 | |||||||||
 キーワード キーワード | HYDROLASE / Thrombin / meizothrombin / allostery / linkage / Na+ binding / Acute phase / Blood coagulation / Cleavage on pair of basic residues / Disease mutation / Gamma-carboxyglutamic acid / Glycoprotein / Kringle / Protease / Secreted / Serine protease / Zymogen | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytolysis by host of symbiont cells / thrombospondin receptor activity / Defective factor XII causes hereditary angioedema / thrombin / thrombin-activated receptor signaling pathway / negative regulation of astrocyte differentiation / regulation of blood coagulation / positive regulation of phospholipase C-activating G protein-coupled receptor signaling pathway / neutrophil-mediated killing of gram-negative bacterium / Defective F8 cleavage by thrombin ...cytolysis by host of symbiont cells / thrombospondin receptor activity / Defective factor XII causes hereditary angioedema / thrombin / thrombin-activated receptor signaling pathway / negative regulation of astrocyte differentiation / regulation of blood coagulation / positive regulation of phospholipase C-activating G protein-coupled receptor signaling pathway / neutrophil-mediated killing of gram-negative bacterium / Defective F8 cleavage by thrombin / Platelet Aggregation (Plug Formation) / ligand-gated ion channel signaling pathway / positive regulation of collagen biosynthetic process / negative regulation of platelet activation / negative regulation of blood coagulation / positive regulation of blood coagulation / negative regulation of fibrinolysis / Transport of gamma-carboxylated protein precursors from the endoplasmic reticulum to the Golgi apparatus / regulation of cytosolic calcium ion concentration / Gamma-carboxylation of protein precursors / Common Pathway of Fibrin Clot Formation / Removal of aminoterminal propeptides from gamma-carboxylated proteins / fibrinolysis / Intrinsic Pathway of Fibrin Clot Formation / negative regulation of proteolysis / negative regulation of cytokine production involved in inflammatory response / Peptide ligand-binding receptors / positive regulation of release of sequestered calcium ion into cytosol / Regulation of Complement cascade / acute-phase response / Cell surface interactions at the vascular wall / positive regulation of receptor signaling pathway via JAK-STAT / growth factor activity / lipopolysaccharide binding / positive regulation of insulin secretion / platelet activation / response to wounding / Golgi lumen / positive regulation of protein localization to nucleus / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / positive regulation of reactive oxygen species metabolic process / blood coagulation / antimicrobial humoral immune response mediated by antimicrobial peptide / heparin binding / regulation of cell shape / Thrombin signalling through proteinase activated receptors (PARs) / : / positive regulation of cell growth / blood microparticle / G alpha (q) signalling events / cell surface receptor signaling pathway / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / receptor ligand activity / endoplasmic reticulum lumen / signaling receptor binding / serine-type endopeptidase activity / positive regulation of cell population proliferation / calcium ion binding / proteolysis / extracellular space / extracellular exosome / extracellular region / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.1 Å 分子置換 / 解像度: 2.1 Å | |||||||||
 データ登録者 データ登録者 | Papaconstantinou, M.E. / Gandhi, P. / Chen, Z. / Bah, A. / Di Cera, E. | |||||||||
 引用 引用 |  ジャーナル: Cell.Mol.Life Sci. / 年: 2008 ジャーナル: Cell.Mol.Life Sci. / 年: 2008タイトル: Na(+) binding to meizothrombin desF1. 著者: Papaconstantinou, M.E. / Gandhi, P.S. / Chen, Z. / Bah, A. / Di Cera, E. #1:  ジャーナル: Structure / 年: 1997 ジャーナル: Structure / 年: 1997タイトル: New insights into the regulation of the blood clotting cascade derived from the X-ray crystal structure of bovine meizothrombin des F1 in complex with PPACK. 著者: Martin, P.D. / Malkowski, M.G. / Box, J. / Esmon, C.T. / Edwards, B.F. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3e6p.cif.gz 3e6p.cif.gz | 103.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3e6p.ent.gz pdb3e6p.ent.gz | 76.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3e6p.json.gz 3e6p.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3e6p_validation.pdf.gz 3e6p_validation.pdf.gz | 1.1 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3e6p_full_validation.pdf.gz 3e6p_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  3e6p_validation.xml.gz 3e6p_validation.xml.gz | 20.1 KB | 表示 | |
| CIF形式データ |  3e6p_validation.cif.gz 3e6p_validation.cif.gz | 28.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/e6/3e6p https://data.pdbj.org/pub/pdb/validation_reports/e6/3e6p ftp://data.pdbj.org/pub/pdb/validation_reports/e6/3e6p ftp://data.pdbj.org/pub/pdb/validation_reports/e6/3e6p | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 2種, 2分子 LH
| #1: タンパク質 | 分子量: 17586.156 Da / 分子数: 1 断片: Activation peptide fragment 2 and thrombin light chain: UNP residues 206-363 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: F2 / プラスミド: HPC4-pNUT / Cell (発現宿主): baby hamster kidney cells Homo sapiens (ヒト) / 遺伝子: F2 / プラスミド: HPC4-pNUT / Cell (発現宿主): baby hamster kidney cells発現宿主:  参照: UniProt: P00734, thrombin |
|---|---|
| #2: タンパク質 | 分子量: 29780.219 Da / 分子数: 1 / 断片: Thrombin heavy chain: UNP residues 364-622 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: F2 / プラスミド: HPC4-pNUT / Cell (発現宿主): baby hamster kidney cells Homo sapiens (ヒト) / 遺伝子: F2 / プラスミド: HPC4-pNUT / Cell (発現宿主): baby hamster kidney cells発現宿主:  参照: UniProt: P00734, thrombin |
-糖 , 1種, 1分子
| #3: 多糖 | 2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose |
|---|
-非ポリマー , 4種, 211分子 
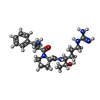





| #4: 化合物 | ChemComp-SO4 / |
|---|---|
| #5: 化合物 | ChemComp-DFK / |
| #6: 化合物 | ChemComp-NA / |
| #7: 水 | ChemComp-HOH / |
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.89 Å3/Da / 溶媒含有率: 68.39 % |
|---|---|
| 結晶化 | 温度: 295 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 6.6 詳細: 0.2M Na2(SO4), 23% PEG 3350, pH 6.6, VAPOR DIFFUSION, HANGING DROP, temperature 295K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 14-BM-C / 波長: 0.9 Å / ビームライン: 14-BM-C / 波長: 0.9 Å |
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2008年6月28日 |
| 放射 | モノクロメーター: Si(111) / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9 Å / 相対比: 1 |
| 反射 | 解像度: 2.1→40 Å / Num. all: 43798 / Num. obs: 40469 / % possible obs: 92.4 % / Observed criterion σ(I): -0.5 / 冗長度: 11.8 % / Rmerge(I) obs: 0.112 / Net I/σ(I): 17.7 |
| 反射 シェル | 解像度: 2.1→2.18 Å / 冗長度: 5.2 % / Rmerge(I) obs: 0.438 / Mean I/σ(I) obs: 2 / Num. unique all: 3398 / % possible all: 79 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB entries 1A0H, 1SHH 解像度: 2.1→32.58 Å / Cor.coef. Fo:Fc: 0.951 / Cor.coef. Fo:Fc free: 0.931 / Isotropic thermal model: isotropic / 交差検証法: THROUGHOUT / σ(F): 0 / σ(I): 0 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: 1. HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. 2. Residues L251-L286 were modeled using bovine meizothrombin (PDB ID code 1A0H) and assigned an occupancy of zero due to lack of ...詳細: 1. HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. 2. Residues L251-L286 were modeled using bovine meizothrombin (PDB ID code 1A0H) and assigned an occupancy of zero due to lack of electron density. 3. Residues with missing atoms listed in Remark 470 are due to lack of electron density for side chains and were modeled as alanines.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 44.5 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.1→32.58 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.1→2.16 Å / Total num. of bins used: 20
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj











