| 登録情報 | データベース: PDB / ID: 3cal
|
|---|
| タイトル | Crystal structure of the second and third fibronectin F1 modules in complex with a fragment of staphylococcus aureus fnbpa-5 |
|---|
 要素 要素 | - Fibronectin
- peptide from Fibronectin-binding protein A
|
|---|
 キーワード キーワード | CELL ADHESION / Fibronectin / 2F13F1 / Beta zipper / Staphylococcus Aureus / Acute phase / Alternative splicing / Extracellular matrix / Glycoprotein / Heparin-binding / Phosphoprotein / Polymorphism / Pyrrolidone carboxylic acid / Secreted / Sulfation / Cell wall / Peptidoglycan-anchor / Virulence |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
aggregation of unicellular organisms / negative regulation of monocyte activation / negative regulation of transforming growth factor beta production / Extracellular matrix organization / positive regulation of substrate-dependent cell migration, cell attachment to substrate / Fibronectin matrix formation / calcium-independent cell-matrix adhesion / neural crest cell migration involved in autonomic nervous system development / fibrinogen complex / peptide cross-linking ...aggregation of unicellular organisms / negative regulation of monocyte activation / negative regulation of transforming growth factor beta production / Extracellular matrix organization / positive regulation of substrate-dependent cell migration, cell attachment to substrate / Fibronectin matrix formation / calcium-independent cell-matrix adhesion / neural crest cell migration involved in autonomic nervous system development / fibrinogen complex / peptide cross-linking / integrin activation / fibrinogen binding / ALK mutants bind TKIs / cell-substrate junction assembly / regulation of protein phosphorylation / proteoglycan binding / biological process involved in interaction with symbiont / extracellular matrix structural constituent / Molecules associated with elastic fibres / MET activates PTK2 signaling / peptidase activator activity / Syndecan interactions / p130Cas linkage to MAPK signaling for integrins / response to muscle activity / endodermal cell differentiation / endoplasmic reticulum-Golgi intermediate compartment / GRB2:SOS provides linkage to MAPK signaling for Integrins / fibronectin binding / basement membrane / Non-integrin membrane-ECM interactions / ECM proteoglycans / Integrin cell surface interactions / endothelial cell migration / regulation of ERK1 and ERK2 cascade / positive regulation of axon extension / collagen binding / Degradation of the extracellular matrix / Integrin signaling / extracellular matrix / substrate adhesion-dependent cell spreading / platelet alpha granule lumen / cell-matrix adhesion / Turbulent (oscillatory, disturbed) flow shear stress activates signaling by PIEZO1 and integrins in endothelial cells / integrin-mediated signaling pathway / acute-phase response / Cell surface interactions at the vascular wall / Post-translational protein phosphorylation / wound healing / Signaling by high-kinase activity BRAF mutants / MAP2K and MAPK activation / response to wounding / integrin binding / Signaling by ALK fusions and activated point mutants / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / positive regulation of fibroblast proliferation / Signaling by RAF1 mutants / Signaling by moderate kinase activity BRAF mutants / Paradoxical activation of RAF signaling by kinase inactive BRAF / Signaling downstream of RAS mutants / Signaling by BRAF and RAF1 fusions / GPER1 signaling / Platelet degranulation / nervous system development / regulation of cell shape / heparin binding / : / heart development / protease binding / blood microparticle / angiogenesis / Interleukin-4 and Interleukin-13 signaling / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / cell adhesion / apical plasma membrane / receptor ligand activity / endoplasmic reticulum lumen / signaling receptor binding / positive regulation of cell population proliferation / positive regulation of gene expression / extracellular space / extracellular exosome / extracellular region / identical protein binding / plasma membrane類似検索 - 分子機能 Fibronectin binding repeat / Fibronectin binding repeat / Fibrinogen-binding domain 2 / C-terminus of bacterial fibrinogen-binding adhesin / SDR-like Ig domain / Bacterial Ig domain / Fibrogen-binding domain 1 / Fibronectin type I domain / Fibronectin, type I / Fibronectin type-I domain signature. ...Fibronectin binding repeat / Fibronectin binding repeat / Fibrinogen-binding domain 2 / C-terminus of bacterial fibrinogen-binding adhesin / SDR-like Ig domain / Bacterial Ig domain / Fibrogen-binding domain 1 / Fibronectin type I domain / Fibronectin, type I / Fibronectin type-I domain signature. / Fibronectin type-I domain profile. / Fibronectin type 1 domain / : / Complement Module, domain 1 / Complement Module; domain 1 / Fibronectin type II domain / Fibronectin type II domain superfamily / Fibronectin type II domain / Fibronectin type-II collagen-binding domain signature. / Fibronectin type-II collagen-binding domain profile. / Fibronectin type 2 domain / YSIRK type signal peptide / Adhesion domain superfamily / YSIRK Gram-positive signal peptide / LPXTG cell wall anchor motif / Gram-positive cocci surface proteins LPxTG motif profile. / LPXTG cell wall anchor domain / Kringle-like fold / Fibronectin type III domain / EGF-like domain signature 1. / Fibronectin type 3 domain / Fibronectin type-III domain profile. / Fibronectin type III / Fibronectin type III superfamily / Ribbon / Immunoglobulin-like fold / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)
  Staphylococcus aureus (黄色ブドウ球菌) Staphylococcus aureus (黄色ブドウ球菌) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.7 Å 分子置換 / 解像度: 1.7 Å |
|---|
 データ登録者 データ登録者 | Bingham, R.J. |
|---|
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2008 ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2008
タイトル: Crystal structures of fibronectin-binding sites from Staphylococcus aureus FnBPA in complex with fibronectin domains
著者: Bingham, R.J. / Meenan, N.A. / Schwarz-Linek, U. / Turkenburg, J.P. / Garman, E.F. / Potts, J.R. |
|---|
| 履歴 | | 登録 | 2008年2月20日 | 登録サイト: RCSB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2008年8月5日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 2.0 | 2019年6月19日 | Group: Advisory / Atomic model ...Advisory / Atomic model / Data collection / Database references / Derived calculations / Polymer sequence / Source and taxonomy / Structure summary
カテゴリ: atom_site / entity ...atom_site / entity / entity_name_com / entity_poly / entity_poly_seq / entity_src_gen / pdbx_entity_nonpoly / pdbx_entity_src_syn / pdbx_nonpoly_scheme / pdbx_poly_seq_scheme / pdbx_struct_assembly_gen / pdbx_struct_sheet_hbond / pdbx_unobs_or_zero_occ_residues / struct / struct_asym / struct_conn / struct_ref_seq / struct_ref_seq_dif / struct_sheet_range
Item: _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ..._atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.group_PDB / _atom_site.label_alt_id / _atom_site.label_asym_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.label_entity_id / _atom_site.label_seq_id / _atom_site.occupancy / _atom_site.type_symbol / _entity_name_com.name / _entity_poly.nstd_monomer / _entity_poly.pdbx_seq_one_letter_code / _entity_poly.pdbx_seq_one_letter_code_can / _entity_src_gen.gene_src_common_name / _entity_src_gen.pdbx_beg_seq_num / _entity_src_gen.pdbx_end_seq_num / _entity_src_gen.pdbx_gene_src_gene / _entity_src_gen.pdbx_seq_type / _pdbx_entity_src_syn.ncbi_taxonomy_id / _pdbx_entity_src_syn.organism_scientific / _pdbx_entity_src_syn.pdbx_beg_seq_num / _pdbx_entity_src_syn.pdbx_end_seq_num / _pdbx_struct_assembly_gen.asym_id_list / _pdbx_struct_sheet_hbond.range_2_label_seq_id / _struct.pdbx_descriptor / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_label_seq_id / _struct_ref_seq.seq_align_beg / _struct_ref_seq.seq_align_end / _struct_sheet_range.beg_label_seq_id / _struct_sheet_range.end_label_seq_id |
|---|
| 改定 2.1 | 2023年11月1日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_conn
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id |
|---|
| 改定 2.2 | 2024年10月30日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.7 Å
分子置換 / 解像度: 1.7 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2008
ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2008 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3cal.cif.gz
3cal.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3cal.ent.gz
pdb3cal.ent.gz PDB形式
PDB形式 3cal.json.gz
3cal.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3cal_validation.pdf.gz
3cal_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3cal_full_validation.pdf.gz
3cal_full_validation.pdf.gz 3cal_validation.xml.gz
3cal_validation.xml.gz 3cal_validation.cif.gz
3cal_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ca/3cal
https://data.pdbj.org/pub/pdb/validation_reports/ca/3cal ftp://data.pdbj.org/pub/pdb/validation_reports/ca/3cal
ftp://data.pdbj.org/pub/pdb/validation_reports/ca/3cal リンク
リンク 集合体
集合体
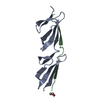

 要素
要素 Homo sapiens (ヒト) / 遺伝子: FN1 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: FN1 / 発現宿主:  Pichia pastoris (菌類) / 株 (発現宿主): GS115 / 参照: UniProt: P02751
Pichia pastoris (菌類) / 株 (発現宿主): GS115 / 参照: UniProt: P02751 Staphylococcus aureus (strain NCTC 8325) (黄色ブドウ球菌)
Staphylococcus aureus (strain NCTC 8325) (黄色ブドウ球菌) X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID14-4 / 波長: 0.979 Å
/ ビームライン: ID14-4 / 波長: 0.979 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー





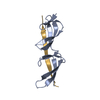









 PDBj
PDBj

















