+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3ae7 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of porcine heart mitochondrial complex II bound with 2-Iodo-N-(3-isopropoxy-phenyl)-benzamide | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | OXIDOREDUCTASE/OXIDOREDUCTASE INHIBITOR / respiratory complex II / inhibitors / Electron transport / Iron / Iron-sulfur / Metal-binding / Mitochondrion / Mitochondrion inner membrane / Oxidoreductase / Transit peptide / Transport / Tricarboxylic acid cycle / Heme / Transmembrane / FAD-binding protein / OXIDOREDUCTASE-OXIDOREDUCTASE INHIBITOR complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Maturation of TCA enzymes and regulation of TCA cycle / Citric acid cycle (TCA cycle) / 酸化還元酵素; CH-OHの結合に対し酸化酵素として働く; キノンあるいはその類似化合物を用いる / succinate metabolic process / respiratory chain complex II (succinate dehydrogenase) / mitochondrial electron transport, succinate to ubiquinone / succinate dehydrogenase (quinone) activity / succinate dehydrogenase / 3 iron, 4 sulfur cluster binding / ubiquinone binding ...Maturation of TCA enzymes and regulation of TCA cycle / Citric acid cycle (TCA cycle) / 酸化還元酵素; CH-OHの結合に対し酸化酵素として働く; キノンあるいはその類似化合物を用いる / succinate metabolic process / respiratory chain complex II (succinate dehydrogenase) / mitochondrial electron transport, succinate to ubiquinone / succinate dehydrogenase (quinone) activity / succinate dehydrogenase / 3 iron, 4 sulfur cluster binding / ubiquinone binding / tricarboxylic acid cycle / protein-membrane adaptor activity / aerobic respiration / respiratory electron transport chain / mitochondrial membrane / 2 iron, 2 sulfur cluster binding / flavin adenine dinucleotide binding / nervous system development / 4 iron, 4 sulfur cluster binding / electron transfer activity / mitochondrial inner membrane / heme binding / nucleolus / metal ion binding 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.62 Å 分子置換 / 解像度: 3.62 Å | ||||||
 データ登録者 データ登録者 | Harada, S. / Sasaki, T. / Shindo, M. / Kido, Y. / Inaoka, D.K. / Omori, J. / Osanai, A. / Sakamoto, K. / Mao, J. / Matsuoka, S. ...Harada, S. / Sasaki, T. / Shindo, M. / Kido, Y. / Inaoka, D.K. / Omori, J. / Osanai, A. / Sakamoto, K. / Mao, J. / Matsuoka, S. / Inoue, M. / Honma, T. / Tanaka, A. / Kita, K. | ||||||
 引用 引用 |  ジャーナル: Int J Mol Sci / 年: 2015 ジャーナル: Int J Mol Sci / 年: 2015タイトル: Structural Insights into the Molecular Design of Flutolanil Derivatives Targeted for Fumarate Respiration of Parasite Mitochondria 著者: Inaoka, D.K. / Shiba, T. / Sato, D. / Balogun, E.O. / Sasaki, T. / Nagahama, M. / Oda, M. / Matsuoka, S. / Ohmori, J. / Honma, T. / Inoue, M. / Kita, K. / Harada, S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3ae7.cif.gz 3ae7.cif.gz | 449.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3ae7.ent.gz pdb3ae7.ent.gz | 369.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3ae7.json.gz 3ae7.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ae/3ae7 https://data.pdbj.org/pub/pdb/validation_reports/ae/3ae7 ftp://data.pdbj.org/pub/pdb/validation_reports/ae/3ae7 ftp://data.pdbj.org/pub/pdb/validation_reports/ae/3ae7 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  3abvC  3ae9C  3aeaC  4ysxC  4ysyC  4yszC  4yt0C  4ytmC  4ytpC  4yxdC  5c2tC  1zoyS C: 同じ文献を引用 ( S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-Succinate dehydrogenase [ubiquinone] ... , 3種, 3分子 ABD
| #1: タンパク質 | 分子量: 68313.172 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|---|
| #2: タンパク質 | 分子量: 28764.217 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #4: タンパク質 | 分子量: 10936.758 Da / 分子数: 1 / Fragment: residues 57-159 / 由来タイプ: 天然 / 由来: (天然)  |
-タンパク質 , 1種, 1分子 C
| #3: タンパク質 | 分子量: 15304.081 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|
-非ポリマー , 6種, 6分子 



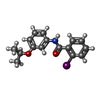






| #5: 化合物 | ChemComp-FAD / |
|---|---|
| #6: 化合物 | ChemComp-FES / |
| #7: 化合物 | ChemComp-SF4 / |
| #8: 化合物 | ChemComp-F3S / |
| #9: 化合物 | ChemComp-12J / |
| #10: 化合物 | ChemComp-HEM / |
-詳細
| 構成要素の詳細 | THESE COMPLEX FORMS MITOCHONDR| Has protein modification | Y | 配列の詳細 | THE SEQUENCE OF CHAIN D IS REFERRED IN REF 2 IN A5GZW8, UNIPROT. | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.56 Å3/Da / 溶媒含有率: 65.44 % / Mosaicity: 0.76 ° |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.2 詳細: 25mM HEPES-NAOH, 6% PEG 4000, 200mM Sucrose, 100mM NaCl, 10mM CaCl2, 0.5mM EDTA, 3% 1,6-haxanediol, 0.5% n-decyl-beta-D-maltoside, pH 7.2, VAPOR DIFFUSION, HANGING DROP, temperature 293K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Photon Factory Photon Factory  / ビームライン: BL-17A / 波長: 0.98 Å / ビームライン: BL-17A / 波長: 0.98 Å |
| 検出器 | タイプ: ADSC QUANTUM 270 / 検出器: CCD / 日付: 2009年11月7日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.98 Å / 相対比: 1 |
| 反射 | 解像度: 3.6→50 Å / Num. obs: 20323 / % possible obs: 97 % / 冗長度: 6.1 % / Rmerge(I) obs: 0.149 / Χ2: 1.64 / Net I/σ(I): 16.143 |
| 反射 シェル | 解像度: 3.6→3.66 Å / 冗長度: 4.4 % / Rmerge(I) obs: 0.553 / Mean I/σ(I) obs: 2.22 / Num. unique all: 809 / Χ2: 1.262 / % possible all: 77.8 |
-位相決定
| 位相決定 | 手法:  分子置換 分子置換 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Phasing MR |
|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1ZOY 解像度: 3.62→48.13 Å / Cor.coef. Fo:Fc: 0.906 / Cor.coef. Fo:Fc free: 0.86 / WRfactor Rfree: 0.272 / WRfactor Rwork: 0.226 / Occupancy max: 1 / Occupancy min: 1 / FOM work R set: 0.786 / SU B: 81.032 / SU ML: 0.587 / SU R Cruickshank DPI: 0.637 / SU Rfree: 0.765 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R Free: 0.765 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS U VALUES : RESIDUAL ONLY
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 142.68 Å2 / Biso mean: 145.11 Å2 / Biso min: 20 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3.62→48.13 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 3.616→3.71 Å / Total num. of bins used: 20
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー




























 PDBj
PDBj



















