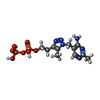
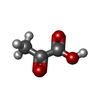
| 登録構造単位 | A: PYRUVATE DECARBOXYLASE
B: PYRUVATE DECARBOXYLASE
E: PYRUVATE DECARBOXYLASE
F: PYRUVATE DECARBOXYLASE
V: PYRUVATE DECARBOXYLASE
X: PYRUVATE DECARBOXYLASE
Y: PYRUVATE DECARBOXYLASE
Z: PYRUVATE DECARBOXYLASE
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 491,647 | 27 |
|---|
| ポリマ- | 487,923 | 8 |
|---|
| 非ポリマー | 3,725 | 19 |
|---|
| 水 | 47,974 | 2663 |
|---|
|
|---|
| 1 | V: PYRUVATE DECARBOXYLASE
X: PYRUVATE DECARBOXYLASE
Y: PYRUVATE DECARBOXYLASE
Z: PYRUVATE DECARBOXYLASE
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 245,868 | 14 |
|---|
| ポリマ- | 243,961 | 4 |
|---|
| 非ポリマー | 1,906 | 10 |
|---|
| 水 | 72 | 4 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
| Buried area | 28420 Å2 |
|---|
| ΔGint | -167.1 kcal/mol |
|---|
| Surface area | 63630 Å2 |
|---|
| 手法 | PISA |
|---|
|
|---|
| 2 | A: PYRUVATE DECARBOXYLASE
B: PYRUVATE DECARBOXYLASE
E: PYRUVATE DECARBOXYLASE
F: PYRUVATE DECARBOXYLASE
ヘテロ分子
| 分子量 (理論値) | 分子数 |
|---|
| 合計 (水以外) | 245,780 | 13 |
|---|
| ポリマ- | 243,961 | 4 |
|---|
| 非ポリマー | 1,818 | 9 |
|---|
| 水 | 72 | 4 |
|---|
| タイプ | 名称 | 対称操作 | 数 |
|---|
| identity operation | 1_555 | x,y,z | 1 |
| Buried area | 28140 Å2 |
|---|
| ΔGint | -173.5 kcal/mol |
|---|
| Surface area | 63760 Å2 |
|---|
| 手法 | PISA |
|---|
|
|---|
| 単位格子 | | Length a, b, c (Å) | 70.619, 111.585, 167.432 |
|---|
| Angle α, β, γ (deg.) | 89.82, 90.11, 78.94 |
|---|
| Int Tables number | 1 |
|---|
| Space group name H-M | P1 |
|---|
|
|---|
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: | Dom-ID | Component-ID | Ens-ID | Selection details |
|---|
| 1 | 1 | 1 | CHAIN A AND (RESSEQ 2:565 )| 2 | 1 | 1 | CHAIN B AND (RESSEQ 2:565 )| 3 | 1 | 1 | CHAIN E AND (RESSEQ 2:565 )| 4 | 1 | 1 | CHAIN F AND (RESSEQ 2:565 )| 5 | 1 | 1 | CHAIN X AND (RESSEQ 2:565 )| 6 | 1 | 1 | CHAIN Y AND (RESSEQ 2:565 )| 7 | 1 | 1 | CHAIN V AND (RESSEQ 2:565 )| 8 | 1 | 1 | CHAIN Z AND (RESSEQ 2:565 ) | | | | | | | |
NCS oper: | ID | Code | Matrix | ベクター |
|---|
| 1 | given(-0.9998, 0.01807, -0.008517), (0.01866, 0.6925, -0.7212), (-0.007135, -0.7212, -0.6927)-0.2284, -0.00322, -0.02674| 2 | given(-1, 0.001253, -0.002173), (-0.001261, -1, 0.003988), (-0.002168, 0.003991, 1)-7.003, 0.8857, 83.73| 3 | given(0.9996, -0.02682, -0.01051), (-0.02687, -0.9996, -0.004498), (-0.01038, 0.004779, -0.9999)-0.01826, -0.01709, 0.0526| 4 | given(0.9998, -0.01939, -0.007185), (-0.01865, -0.6954, -0.7184), (0.008936, 0.7184, -0.6956)5.943, -59.95, -58.58| 5 | given(-0.9998, 0.007992, 0.02018), (0.008962, -0.6947, 0.7192), (0.01977, 0.7192, 0.6945)-0.2668, -0.01691, -0.03341| 6 | given(0.9997, -0.007679, 0.02118), (-0.009836, 0.6969, 0.7171), (-0.02027, -0.7171, 0.6967)8.382, 59.51, 58.59| 7 | given(-0.9996, 0.0258, -0.01184), (0.02589, 0.9996, -0.007765), (0.01163, -0.008069, -0.9999)| -7.812, -1.029, -83.54 | | | | | | | | | | | | | | | | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 ZYMOMONAS MOBILIS (バクテリア)
ZYMOMONAS MOBILIS (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.2 Å
分子置換 / 解像度: 2.2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Biochemistry / 年: 2010
ジャーナル: Biochemistry / 年: 2010 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2wva.cif.gz
2wva.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2wva.ent.gz
pdb2wva.ent.gz PDB形式
PDB形式 2wva.json.gz
2wva.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/wv/2wva
https://data.pdbj.org/pub/pdb/validation_reports/wv/2wva ftp://data.pdbj.org/pub/pdb/validation_reports/wv/2wva
ftp://data.pdbj.org/pub/pdb/validation_reports/wv/2wva リンク
リンク 集合体
集合体


 要素
要素 ZYMOMONAS MOBILIS (バクテリア) / プラスミド: PPL450 / 発現宿主:
ZYMOMONAS MOBILIS (バクテリア) / プラスミド: PPL450 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  Diamond
Diamond  / ビームライン: I02 / 波長: 0.951
/ ビームライン: I02 / 波長: 0.951  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー
















 PDBj
PDBj