[English] 日本語
 Yorodumi
Yorodumi- PDB-2onm: Human Mitochondrial Aldehyde Dehydrogenase Asian Variant, ALDH2*2... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 2onm | ||||||
|---|---|---|---|---|---|---|---|
| Title | Human Mitochondrial Aldehyde Dehydrogenase Asian Variant, ALDH2*2, complexed with NAD+ | ||||||
 Components Components | Aldehyde dehydrogenase, mitochondrial precursor | ||||||
 Keywords Keywords | OXIDOREDUCTASE / ALDH / NAD / Rossmann Fold | ||||||
| Function / homology |  Function and homology information Function and homology informationMetabolism of serotonin / regulation of dopamine biosynthetic process / regulation of serotonin biosynthetic process / phenylacetaldehyde dehydrogenase (NAD+) activity / nitroglycerin metabolic process / aldehyde catabolic process / alcohol metabolic process / ethanol catabolic process / Ethanol oxidation / aldehyde dehydrogenase [NAD(P)+] activity ...Metabolism of serotonin / regulation of dopamine biosynthetic process / regulation of serotonin biosynthetic process / phenylacetaldehyde dehydrogenase (NAD+) activity / nitroglycerin metabolic process / aldehyde catabolic process / alcohol metabolic process / ethanol catabolic process / Ethanol oxidation / aldehyde dehydrogenase [NAD(P)+] activity / aldehyde dehydrogenase (NAD+) / carboxylesterase activity / aldehyde dehydrogenase (NAD+) activity / Smooth Muscle Contraction / Mitochondrial protein degradation / NAD binding / carbohydrate metabolic process / electron transfer activity / mitochondrial matrix / mitochondrion / extracellular exosome Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.5 Å MOLECULAR REPLACEMENT / Resolution: 2.5 Å | ||||||
 Authors Authors | Larson, H.N. / Hurley, T.D. | ||||||
 Citation Citation |  Journal: J.Biol.Chem. / Year: 2007 Journal: J.Biol.Chem. / Year: 2007Title: Structural and functional consequences of coenzyme binding to the inactive asian variant of mitochondrial aldehyde dehydrogenase: roles of residues 475 and 487. Authors: Larson, H.N. / Zhou, J. / Chen, Z. / Stamler, J.S. / Weiner, H. / Hurley, T.D. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  2onm.cif.gz 2onm.cif.gz | 1.1 MB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb2onm.ent.gz pdb2onm.ent.gz | 955.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  2onm.json.gz 2onm.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/on/2onm https://data.pdbj.org/pub/pdb/validation_reports/on/2onm ftp://data.pdbj.org/pub/pdb/validation_reports/on/2onm ftp://data.pdbj.org/pub/pdb/validation_reports/on/2onm | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  2onnC  2onoC  2onpC  1o05S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| Unit cell |
| ||||||||
| Details | The biological unit is a tetramer. There are 3 biological untils in the assymmetric unit. The biological unit 1 consists of chain(s) A, B, C, D. The biological unit 2 consists of chain(s) E, F, G, H. The biological unit 3 consists of chain(s) I, J, K, L. |
- Components
Components
-Protein , 1 types, 12 molecules ABCDEFGHIJKL
| #1: Protein | Mass: 54499.695 Da / Num. of mol.: 12 / Mutation: E487K Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: ALDH2, ALDM / Plasmid: pT-7-7 / Species (production host): Escherichia coli / Production host: Homo sapiens (human) / Gene: ALDH2, ALDM / Plasmid: pT-7-7 / Species (production host): Escherichia coli / Production host:  |
|---|
-Non-polymers , 6 types, 1995 molecules 


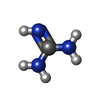







| #2: Chemical | ChemComp-NA / #3: Chemical | ChemComp-ADP / #4: Chemical | ChemComp-EDO / #5: Chemical | ChemComp-GAI / #6: Chemical | ChemComp-NAD / #7: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.44 Å3/Da / Density % sol: 49.53 % |
|---|---|
| Crystal grow | Temperature: 293 K / Method: vapor diffusion / pH: 6.8 Details: 100 mM ACES (N-[2-Acetamido]-2-aminoethane sulfonic acid), 10mM MgCl2, 100 mM Guanidine HCl, 15% w/v PEG 6000, 8mM DTT, pH 6.8, VAPOR DIFFUSION, temperature 293K |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ALS ALS  / Beamline: 4.2.2 / Wavelength: 1.072 Å / Beamline: 4.2.2 / Wavelength: 1.072 Å |
| Detector | Type: NOIR-1 / Detector: CCD / Date: Mar 29, 2005 Details: Rosenbaum-Rock monochromator 1: high-resolution double-crystal sagittal focusing, Rosenbaum-Rock monochromator 2: double crystal, Rosenbaum-Rock vertical focusing mirror |
| Radiation | Monochromator: double crystal / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.072 Å / Relative weight: 1 |
| Reflection | Resolution: 2.5→48.95 Å / Num. all: 213379 / Num. obs: 208258 / % possible obs: 97.6 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / Redundancy: 1.95 % / Biso Wilson estimate: 50.682 Å2 / Rmerge(I) obs: 0.058 / Χ2: 1.01 / Net I/σ(I): 9.1 / Scaling rejects: 3067 |
| Reflection shell | Resolution: 2.5→2.59 Å / Redundancy: 1.95 % / Rmerge(I) obs: 0.325 / Mean I/σ(I) obs: 2 / Num. measured all: 40530 / Num. unique all: 20681 / Χ2: 1.08 / % possible all: 97 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB ENTRY 1O05 Resolution: 2.5→44.01 Å / Rfactor Rfree error: 0.003 / FOM work R set: 0.791 / Data cutoff high absF: 2764167 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / Cross valid method: THROUGHOUT / σ(F): 0 / σ(I): 0 / Stereochemistry target values: Engh & Huber
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Solvent model: FLAT MODEL / Bsol: 30.95 Å2 / ksol: 0.307 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 58.4 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.5→44.01 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 50
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
|
 Movie
Movie Controller
Controller












 PDBj
PDBj










