| 登録情報 | データベース: PDB / ID: 2mcr
|
|---|
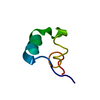
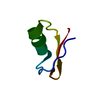
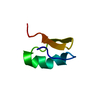
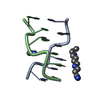
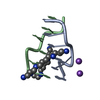
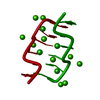
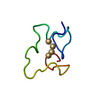
| タイトル | Solution structure of ShK-like immunomodulatory peptide from Brugia malayi (filarial worm) |
|---|
 要素 要素 | Probable zinc metalloproteinase, putative |
|---|
 キーワード キーワード | IMMUNE SYSTEM / worms / BmK1 / ShK-like peptide / Kv1.3 |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; その他のペプチターゼ / metalloendopeptidase activity / proteolysis / zinc ion binding類似検索 - 分子機能 ShK toxin domain / ShK domain-like / Astacin-like metallopeptidase domain / ShKT domain / ShKT domain profile. / Astacin-like domain profile. / Peptidase M12A / Astacin (Peptidase family M12A) / Peptidase, metallopeptidase / Zinc-dependent metalloprotease / Metallopeptidase, catalytic domain superfamily類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Brugia malayi (マレー糸状虫) Brugia malayi (マレー糸状虫) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
| Model details | closest to the average, model1 |
|---|
 データ登録者 データ登録者 | Chhabra, S. / Swarbrick, J.D. / Pennington, M.W. / Chang, S.C. / Norton, R.S. |
|---|
 引用 引用 |  ジャーナル: Faseb J. / 年: 2014 ジャーナル: Faseb J. / 年: 2014
タイトル: Kv1.3 channel-blocking immunomodulatory peptides from parasitic worms: implications for autoimmune diseases.
著者: Chhabra, S. / Chang, S.C. / Nguyen, H.M. / Huq, R. / Tanner, M.R. / Londono, L.M. / Estrada, R. / Dhawan, V. / Chauhan, S. / Upadhyay, S.K. / Gindin, M. / Hotez, P.J. / Valenzuela, J.G. / ...著者: Chhabra, S. / Chang, S.C. / Nguyen, H.M. / Huq, R. / Tanner, M.R. / Londono, L.M. / Estrada, R. / Dhawan, V. / Chauhan, S. / Upadhyay, S.K. / Gindin, M. / Hotez, P.J. / Valenzuela, J.G. / Mohanty, B. / Swarbrick, J.D. / Wulff, H. / Iadonato, S.P. / Gutman, G.A. / Beeton, C. / Pennington, M.W. / Norton, R.S. / Chandy, K.G. |
|---|
| 履歴 | | 登録 | 2013年8月22日 | 登録サイト: BMRB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2014年7月2日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2015年4月8日 | Group: Database references |
|---|
| 改定 1.2 | 2016年4月27日 | Group: Structure summary |
|---|
| 改定 1.3 | 2023年6月14日 | Group: Data collection / Database references / Other
カテゴリ: database_2 / pdbx_database_status ...database_2 / pdbx_database_status / pdbx_nmr_software / pdbx_nmr_spectrometer / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_nmr_data / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model / _struct_ref_seq_dif.details |
|---|
| 改定 1.4 | 2024年11月27日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature
Item: _database_2.pdbx_DOI |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Brugia malayi (マレー糸状虫)
Brugia malayi (マレー糸状虫) データ登録者
データ登録者 引用
引用 ジャーナル: Faseb J. / 年: 2014
ジャーナル: Faseb J. / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2mcr.cif.gz
2mcr.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2mcr.ent.gz
pdb2mcr.ent.gz PDB形式
PDB形式 2mcr.json.gz
2mcr.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/mc/2mcr
https://data.pdbj.org/pub/pdb/validation_reports/mc/2mcr ftp://data.pdbj.org/pub/pdb/validation_reports/mc/2mcr
ftp://data.pdbj.org/pub/pdb/validation_reports/mc/2mcr リンク
リンク 集合体
集合体
 要素
要素 Brugia malayi (マレー糸状虫) / 参照: UniProt: A8PQK5, UniProt: A0A0J9XQE3*PLUS
Brugia malayi (マレー糸状虫) / 参照: UniProt: A8PQK5, UniProt: A0A0J9XQE3*PLUS 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj


 HSQC
HSQC