+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2lxy | ||||||
|---|---|---|---|---|---|---|---|
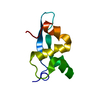
| タイトル | NMR structure of 2-MERCAPTOPHENOL-ALPHA3C | ||||||
 要素 要素 | 2-mercaptophenol-alpha3C | ||||||
 キーワード キーワード | DE NOVO PROTEIN / three-helix bundle / redox protein / phenol oxidation-reduction / proton-coupled electron transfer | ||||||
| 機能・相同性 | Designed single chain three-helix bundle / Substrate Binding Domain Of Dnak; Chain:A; Domain 2 / Up-down Bundle / Mainly Alpha / 2-MERCAPTOPHENOL 機能・相同性情報 機能・相同性情報 | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
| Model details | lowest energy, model1 | ||||||
 データ登録者 データ登録者 | Tommos, C. / Valentine, K.G. / Martinez-Rivera, M.C. / Liang, L. / Moorman, V.R. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2013 ジャーナル: Biochemistry / 年: 2013タイトル: Reversible phenol oxidation and reduction in the structurally well-defined 2-Mercaptophenol-alpha(3)C protein. 著者: Tommos, C. / Valentine, K.G. / Martinez-Rivera, M.C. / Liang, L. / Moorman, V.R. #1: ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2012 タイトル: Reversible voltammograms and a Pourbaix diagram for a protein tyrosine radical. 著者: Berry, B.W. / Martinez-Rivera, M.C. / Tommos, C. #2: ジャーナル: J.Am.Chem.Soc. / 年: 2011 タイトル: Electrochemical and structural properties of a protein system designed to generate tyrosine Pourbaix diagrams. 著者: Martinez-Rivera, M.C. / Berry, B.W. / Valentine, K.G. / Westerlund, K. / Hay, S. / Tommos, C. #3: ジャーナル: Biochemistry / 年: 2005 タイトル: Moving a phenol hydroxyl group from the surface to the interior of a protein: effects on the phenol potential and pK(A). 著者: Hay, S. / Westerlund, K. / Tommos, C. #4:  ジャーナル: J.Am.Chem.Soc. / 年: 2002 ジャーナル: J.Am.Chem.Soc. / 年: 2002タイトル: Structure of a de novo designed protein model of radical enzymes. 著者: Dai, Q.H. / Tommos, C. / Fuentes, E.J. / Blomberg, M.R. / Dutton, P.L. / Wand, A.J. #5: ジャーナル: Biochemistry / 年: 1999 タイトル: De novo proteins as models of radical enzymes. 著者: Tommos, C. / Skalicky, J.J. / Pilloud, D.L. / Wand, A.J. / Dutton, P.L. #6: ジャーナル: Biochemistry / 年: 1999 タイトル: De novo proteins as models of radical enzymes 著者: Tommos, C. / Skalicky, J.J. / Pilloud, D.L. / Wand, A. / Dutton, P. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2lxy.cif.gz 2lxy.cif.gz | 674.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2lxy.ent.gz pdb2lxy.ent.gz | 569.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2lxy.json.gz 2lxy.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2lxy_validation.pdf.gz 2lxy_validation.pdf.gz | 450.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2lxy_full_validation.pdf.gz 2lxy_full_validation.pdf.gz | 660.7 KB | 表示 | |
| XML形式データ |  2lxy_validation.xml.gz 2lxy_validation.xml.gz | 40.5 KB | 表示 | |
| CIF形式データ |  2lxy_validation.cif.gz 2lxy_validation.cif.gz | 64.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/lx/2lxy https://data.pdbj.org/pub/pdb/validation_reports/lx/2lxy ftp://data.pdbj.org/pub/pdb/validation_reports/lx/2lxy ftp://data.pdbj.org/pub/pdb/validation_reports/lx/2lxy | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ | |
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 7479.832 Da / 分子数: 1 / 由来タイプ: 合成 / 発現宿主:  |
|---|---|
| #2: 化合物 | ChemComp-HTS / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 試料状態 | イオン強度: 60 / pH: 5.5 / 圧: ambient / 温度: 303 K |
-NMR測定
| NMRスペクトロメーター | タイプ: Varian INOVA / 製造業者: Varian / モデル: INOVA / 磁場強度: 750 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 | ||||||||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 1000 / 登録したコンフォーマーの数: 32 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj


 HSQC
HSQC